| Главная | Семестры | Обо мне | Ссылки |
|---|
Поиск мотивов в Prosite
Множественное выравнивание: часть последовательностей из практикума 9 (aln1_1.fasta), загрузить.
Проект Jalview с выравниванием: загрузить.
Вначале был осуществлен поиск мотивов в последовательностях с помощью Prosite.
Входные данные: последовательности (не более 10 и без гэпов!), отображение Graphical view.
Обнаружился 1 мотив под названием G_PROTEIN_RECEP_F1_2 (G-protein coupled receptors family 1), то есть семейство рецепторов, сопряженных с G-белком.
Это группа рецепторов, которые передают сигнал во внеклеточное пространство путем взаимодействия с белками, связывающими гуанин нуклеотиды (по информации Prosite).
Лигандами для этих рецепторов выступают различные вещества, в том числе гормоны, нейромедиаторы, светочувствительные вещества, пахучие вещества, феромоны.
В частности, данное множественное выравнивание содержит последовательности рецептора холецистокинина - нейромедиатора.
Оно показано на рис. 1, мотив отмечен буквой М. На рис. 2 отдельно выделен обнаруженный мотив. Заметим, что лишь одна позиция абсолютно консервативна: это позиция №13 с аргинином.

Рис. 1. Множественное выравнивание последовательностей с мотивом G_PROTEIN_RECEP_F1_2 (отмечен буквой М в строке Motif). Раскраска BLOSUM62, консервативность 70%
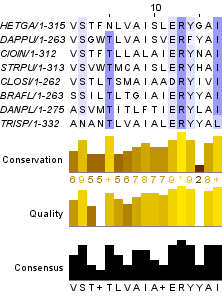
Рис. 2. Результат PSI-BLAST и выбранные последовательности
Далее построим паттерн мотива. Паттерн мотива аналогичен маске файла или регулярному выражению программ bash. Для каждой позиции мотива я выписала все встретившиеся аминокислоты. При построении сильного паттерна позиции, в которых было 4 разных аминокислоты, были расширены до функциональной группы, а позиции с 5-6 разными аминокислотами - до любой аминокислоты (х). Чтобы получить слабый паттерн, позиции с 2 аминокислотами были также расширены до функциональной группы, а с 3-6 - до любой аминокислоты (х). Результат представлен в таблице 1.
Таблица 1. Составление паттернов для мотива G_PROTEIN_RECEP_F1_2
| № позиции | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| А.о. в выравнивании | VSA | SN | TGVIA | FWLMN | NT | LMSI | VLCMT | AGL | ILF | SAT | LVIA | ED | R | YF | GYNHIL | AV | IL |
| Сильный паттерн | VSA | SN | X | X | NT | LMSTVI | X | AGL | ILF | SAT | AGVIL | ED | R | YF | X | AV | IL |
| Слабый паттерн | X | STND | X | X | STND | X | X | X | X | X | X | ED | R | YF | X | AGVIL | ILV |
Сильный паттерн: [VSA]-[SN]-X(2)-[NT]-[LMSTVI]-X-[AGL]-[ILF]-[SAT]-[AGVIL]-[ED]-R-[YF]-X-[AV]-[IL]
Слабый паттерн: X-[STND]-X(2)-[STND]-X(6)-[ED]-R-[YF]-X-[AGVIL]-[ILV]
Теперь посмотрим, сколько последовательностей обнаружит Prosite для каждого паттерна. Вначале проведем поиск по сильному паттерну.
В базе данных Swiss-Prot: 38 находок в 38 последовательностях, если включить опцию Include splice variants, иначе 29 находок в 29 последовательностях. Все исходные последовательности были найдены.
В базе данных TrEMBL: 804 находки в 796 последовательностях.
Получилось так, что две последовательности содержали по 5 (!) искомых мотивов.
Графическое отображение одной из таких последовательностей показано на рис. 3.
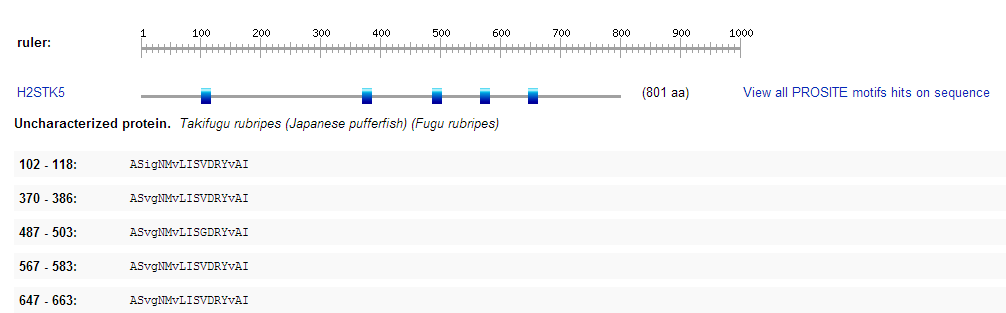
Рис. 3. Графическое изображение искомых мотивов в последовательности с АС H2STK5. Получено с помощью Prosite
Теперь проведем поиск по слабому паттерну.
В базе данных Swiss-Prot: 754 находки в 754 последовательностях, если включить опцию Include splice variants, иначе 656 находок в 656 последовательностях.
В базе данных TrEMBL: 10013 находки в 10000 последовательностях. 10000 последовательностей - лимит для поиска, то есть вполне вероятно, что действительно их можно найти больше.
Таким образом, в каждом случае обнаружилось довольно много последовательностей. Это неудивительно, ведь искомый мотив небольшой и содержит часто встречаемые аминокислоты. К тому же, исследование нейромедиаторов и их рецепторов очень актуально, например, для разработки лекарств.