| Главная | Семестры | Обо мне | Ссылки |
|---|
Работа в EMBOSS
Программа getorf
Вначале был получен файл с записью D89965 банка EMBL.
entret embl:D89965
Как указано в записи, в ней приведена информация о гене серой крысы.
Он кодирует белок, отвечающий за передачу сигнала с помощью серотониновых рецепторов в крысином желудке.
getorf - программа для поиска открытых рамок считывания в последовательности нуклеотидов.
Опция -minsize задает минимальное число нуклеотидов в рамке.
Чтобы искать только те рамки, которые начинаются со старт-кодона и заканчиваются стоп-кодоном, используется опция -find 1.
getorf d89965.entret -find 1 -minsize 90
Полученный файл: d89965.orf
Было найдено 5 открытых рамок, 2 из них - на комплементарной цепи.
Одна из них [163..432] совпадает с той, что указана в CDS [163..435], видимо, с учетом последнего триплета, кодирующего стоп-кодон.
В записи EMBL есть ссылка на запись о белке в Swiss-Prot, AC P0A7B8. Файл: hslv_ecoli.entret. Это последовательность белка протеазы E.coli. Она частично совпадает с рамкой считывания [294..1]. Скорее всего, при получении ДНК из желудка крысы в образец попала ДНК бактерии E.coli, которая в норме обитает в кишечнике. И в результате именно бактериальная ДНК была секвенирована.
Файлы-списки
Для начала нужно было получить все последовательности алкогольдегидрогеназ, доступные в Swiss-Prot:
entret sw:adh*_*
seqret adh_drope.entret adh.fasta
Результат: adh.fasta
С помощью программы infoseq можно создать список универсальных адресов (USA) этих последовательностей.
infoseq sw:adh*_* -only -usa > adh.list
Если нужны последовательности не всех организмов, а только некоторых:
grep -E 'DROMO|SAAHA|NEUCR|CUPNE|MOUSE|STAAB|ARCFU' 'adh.list' > my_adh.list
seqret @my_adh.list my_adh.fasta
Полученный файл: my_adh.fasta.
Случайная модель для оценки достоверности выравнивания
Из файла с последовательностями алкогольдегидрогеназ была выбрана одна из организма Neurospora crassa, это гриб из отдела аскомицетов.
Программа shuffleseq создает нужное количество случайных последовательностей той же длины и с тем же набором букв, что и в исходной последовательности.
shuffleseq -shuffle 100 neucr.fasta neucr_shuffled.fasta
Файлы: neucr.fasta, neucr_shuffled.fasta
Домовая мышь - сильно далекий от гриба организм.
Но если последовательности кодируют белки с одной и той же функцией, они могут быть гомологичны.
Для проверки этого построим парные локальные выравнивания мышиной последовательности с исходной и с каждой из 100 перемешанных последовательностей.
Далее объединим все выравнивания в один файл.
water mouse.fasta neucr_shuffled.fasta alignment
water mouse.fasta neucr.fasta aln_1
cat aln_1 >> alignment
Файлы: mouse.fasta, alignment.
Получить список весов всех выравниваний можно так:
grep Score alignment|sed 's/^[# Score: ]*//' > score
На основе полученных данных была построена гистограмма распределения полученных весов. Она представлена на рис. 1.
Таблица Excel: pr5.xlsx
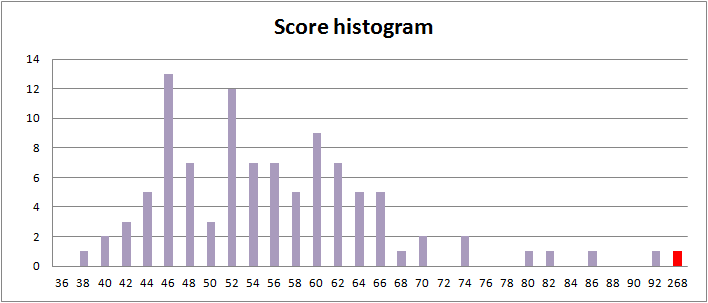
Рис. 1. Гистограмма распределения весов выравниваний белковых последовательностей
Вес выравнивания с исходной последовательностью равен 267.0. Он оказался в крайнем правом месте гистограммы. Здесь и далее значение веса исходного выравнивания отмечены красным. Его значение почти в 3 раза превышает предыдущее. Значит, полученное выравнивание сильно отличается от случайного, то есть можно утверждать о гомологии последовательностей.
Далее было сделано то же самое для последовательностей генов. Для этого были использованы ссылки на базу данных EMBL в записях. У мыши ссылок оказалось очень много:
DR EMBL; M18480; AAA37178.1; -; Genomic_DNA.
DR EMBL; M18472; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18473; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18474; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18475; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18476; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18477; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M18478; AAA37178.1; JOINED; Genomic_DNA.
DR EMBL; M22679; AAA37179.1; -; Genomic_DNA.
DR EMBL; M22671; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22672; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22673; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22674; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22675; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22676; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M22677; AAA37179.1; JOINED; Genomic_DNA.
DR EMBL; M11307; AAA37180.1; -; mRNA.
DR EMBL; BC013477; AAH13477.1; -; mRNA.
DR EMBL; BC054467; AAH54467.1; -; mRNA.
DR EMBL; Z32540; -; NOT_ANNOTATED_CDS; Genomic_DNA.
DR EMBL; M22611; AAA37181.1; -; mRNA.
Я работала с двумя записями из первой строки ссылок: m18480.embl, aaa37178.embl. Получились заметно различающиеся результаты. Гистограмма для первого выравнивания показана на рис.2, для второго - на рис. 3.
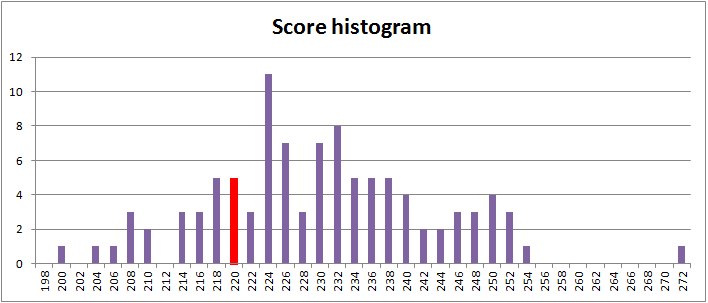
Рис. 2. Гистограмма распределения весов выравниваний последовательностей генов
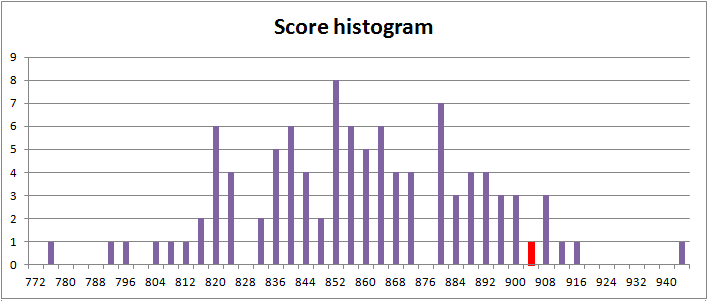
Рис. 3. Гистограмма распределения весов выравниваний последовательностей генов
218.5, 901.5 - веса исходных выравниваний.
Видим, что выравнивание с последовательностью гена белка не отличается от выравниваний со случайными последовательностями.
Возможно, был выбран не самый лучший файл с геном белка мыши. Но из полученных данных нельзя сделать вывод, что есть гомология последовательностей.