| Главная | Семестры | Обо мне | Ссылки |
|---|
Онлайн BLAST
Поиск организма по фрагменту нуклеотидной последовательности
Фрагмент последовательности: seq.fasta.
С помощью программы megablast на сайте NCBI можно найти исходную последовательность, откуда был взят фрагмент.
Поиск был в базе данных refseq_genomic, только среди бактерий и архей.
В результате была найдена запись с Refseq AC NC_015636, это геном археи Methanothermococcus okinawensis IH1.
Фрагмент имеет координаты в геноме 1145..1444. Его расположение показано на рис. 1.
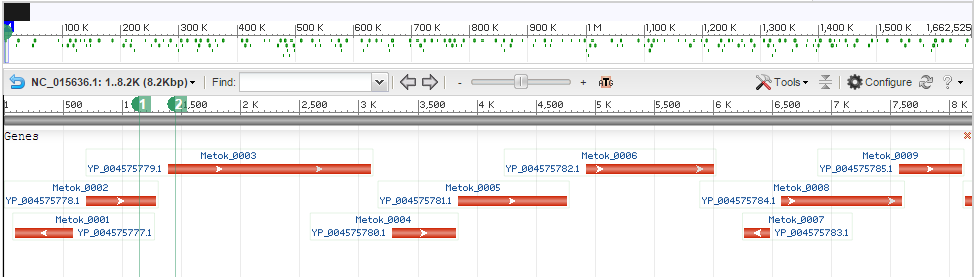
Рис. 1. Участок генома Methanothermococcus okinawensis IH1. Между маркерами 1 и 2 находится исходный фрагмент
Как видно из рис. 1, фрагмент содержит конец одного гена и начало другого. Вот описание этих генов:
CDS: YP_004575778.1
Title: hypothetical protein
Comment: PFAM: Protein of unknown function DUF95, transmembrane~KEGG: mmq:MmarC5_1813 hypothetical protein
Location: 692..1,285
CDS: YP_004575779.1
Title: PUA domain-containing protein
Comment: KEGG: mae:Maeo_1488 PUA domain-containing protein~PFAM: Pseudouridine synthase/archaeosine
transglycosylase~SMART: Pseudouridine synthase/archaeosine transglycosylase
Location: 1,383..3,101
Второй белок участвует в синтезе псевдоуридина, функция первого не выяснена. Возможно, оба белка находятся в одном опероне, так как их гены сонаправлены.
Поиск гомолога белка человека у африканского слона
Командой bash
infoseq sw:vas*_human -only -name -desc -out vas.txt
были найдены белки, начинающиеся на первые три буквы моей фамилии.
Файл: vas.txt.
Далее будем работать с белком, чей AC VAS1L_HUMAN (V-type proton ATPase subunit S1-like protein (Vacuolar proton pump subunit S1-like protein)), это протонный канал в клеточной мембране.
Скорее всего, этот белок очень консервативен, и можно найти его гомолога у довольно близкого организма - африканского слона Loxodonta africana.
Сначала была получена последовательность данного белка:
seqret sw:vas1l_human -auto
Поиск велся на сайте ENA, а именно spliced translated nucleotide search.
Всего 2 находки, они показаны на рис. 2. У лучшей длина выравнивания 101, identity 79%, e-value 1E-34, координаты в геноме слона 85953709..85954011, интронов нет.

Рис. 2. Результат поиска на сайте ENA
Поиск некодирующих последовательностей программой BLAST
Требовалось создать файл с последовательностью любой тРНК из археи Methanococcus voltae A3, далее провести поиск по всем археям того же порядка (Methanococcales) тремя вариантами:
- алгоритмом megablast;
- алгоритмом blastn с параметрами по умолчанию;
- алгоритмом blastn с длиной слова = 7, match/mismatch = 1/-1
Получить последовательность тРНК удалось только через полный геном археи, так как отдельных файлов для тРНК нет.
Я вырезала участок гена тРНК, кодирующей глутаминовую кислоту, в отдельный файл и перевела ДНК в комплементарную ей РНК онлайн-конвертером.
Файл: trna.fasta.
Чтобы определить, к какому порядку принадлежит данная архея, был проведен поиск на сайте NCBI по базе данных Taxonomy. Число находок с e-value < 0,001 для трех вариантов приведено в таблице 1.
Таблица 1. Результаты blastn с разными параметрами
| Программа | Число находок | Число геномов |
|---|---|---|
| megablast | 32 | 17 |
| blastn по умолчанию | 137 | 21 |
| blastn с длиной слова = 7, match/mismatch = 1/-1 | 189 | 21 |
Интересно, что лучшая находка, не считая исходной, с Identity 100% принадлежит архее Methanothermococcus okinawensis IH1 из задания 1. В целом результаты предсказуемы, так как чем меньше длина слова и штраф за mismatch, тем больше последовательностей будет найдено.
Сравнение программ BLASTN и MegaBLAST
Все, что нашел megablast, имеет query cover 100%, найдено и расположено в том же порядке при поиске blastn. blastn обнаружил еще 4 генома, причем при разных параметрах получились немного различающиеся значения, это показано на рис. 3. Длины выравниваний одинаковы, а вес определяется значением match/mismatch. По умолчанию это 2/-3. Во втором случае это 1/-1,то есть максимально чувствительные параметры. Как сказано в справке NCBI, такое соотношение match/mismatch используется для поиска последовательностей, которые идентичны на 75%.

Рис. 3. Сравнение отличающихся находок blastn при различных параметрах поиска: сверху от линии - поиск по умолчанию, снизу - с длиной слова = 7, match/mismatch = 1/-1