| Главная | Семестры | Обо мне | Ссылки |
|---|
Standalone BLAST
Поиск некодирующих РНК, аннотированных в одном штамме, в геноме другого штамма
Вначале был скачан файл со всеми misc_RNA штамма Bacillus_subtilis_168_uid57675 с FTP-сервера NCBI NC_000964.frn.
В нем приведены последовательности рибосомальных РНК (ribosomal RNA), тРНК (tRNA) и некодирующих РНК (misc_RNA) данного штамма.
Чтобы получить файл с последовательностями только misc_RNA:
infoseq NC_000964.frn -only -usa -desc > list
grep 'misc_RNA' 'list' > 'misc_list'
Из файла misc_list было удалено все, кроме 1-го столбца. Теперь этот файл - список адресов (USA) нужных последовательностей, которые можно получить командой seqret:
seqret @misc_list misc_rna.fasta
Файлы: misc_list, misc_rna.fasta
Также с FTP-сервера NCBI были получены файлы с геномом штамма Bacillus_subtilis_spizizenii_TU_B_10_uid73967 NC_016047.gbk, NC_016047.fna.
Они используются для создания локальной базы данных, по которой можно искать программой blast.
Проведем поиск тех же некодирующих РНК, что и у первого штамма, алгоритмом megablast.
makeblastdb -in NC_016047.fna -dbtype nucl
blastn -task megablast -query misc_rna.fasta -db NC_016047.fna -out blast.txt -outfmt 7 -num_alignments 1
Результаты поиска - в таблице Excel(задание 1).
Поиск гомологов РНК Bacillus subtilis в геноме другой бактерии
Будем искать гомологи misc_RNA из первого задания в геноме Bacillus_cereus_Q1_uid58529.
Для этого применим разные алгоритмы программы blastn: megablast, blastn с параметрами по умолчанию, blastn с параметрами: длина слова = 4, награда за совпадение = 1, штраф за несовпадение = –1.
makeblastdb -in NC_011969.fna -dbtype nucl
blastn -task megablast -query misc_rna.fasta -db NC_011969.fna -out blast2mega.txt -outfmt 7 -evalue 0.001
blastn -task blastn -query misc_rna.fasta -db NC_011969.fna -out blast2n.txt -outfmt 7 -evalue 0.001
blastn -task blastn -query misc_rna.fasta -db NC_011969.fna -out blast2n2.txt -outfmt 7 -evalue 0.001 -word_size 4 -penalty -1 -reward 1
Файлы: NC_011969.gbk, NC_011969.fna
Количество гомологов c e-value < 0.001 приведено в таблице Excel (задание 2).
Как правило, число находок увеличивалось в ряду megablast > blastn с параметрами по умолчанию > blastn с установленными параметрами (менее чувствительными).
Есть случаи, когда blastn с параметрами по умолчанию нашел больше гомологов, чем blastn с установленными параметрами.
Скорее всего, эти находки имеют большой e-value, из-за разных баллов за совпадение/несовпадение их e-value оказался меньше порогового 0.001 в первом случае и больше во втором.
Поиск неправильно аннотированных генов программой blastx
Был скачан файл с последовательностями предсказанных белков штамма Bacillus_subtilis_spizizenii_TU_B_10_uid73967 NC_016047.faa.
Из него я получила локальную базу данных белков.
В ней программой blastx проведем поиск гомологов misc_RNA из предыдущих заданий.
makeblastdb -in NC_016047.faa -dbtype prot
blastx -query misc_rna.fasta -db NC_016047.faa -out blastx.txt -outfmt 7 -evalue 0.001
Нашлись гомологи для: 1, 25, 41, 46, 53, 57, 58, 61, 62. В таблице Excel (задание 3) приведены идентификаторы и e-value находок.
Для misc РНК под номерами 57 и 58, 61 и 62 были найдены одни и те же белки. В таблице эти пары выделены цветом. E-value у 61 и 62 отличается более чем на 13 порядков, а у 57 и 58 - всего в 2,5 раза.
Получим описания всех найденных белков:
grep -E 'YP_004875421.1|YP_004877162.1|YP_004878230.1|YP_004878330.1|YP_004878785.1|YP_004879343.1|YP_004879490.1' 'NC_016047.faa' > desc.txt
Результат: desc.txt.
Описание "гомолога" misc РНК №1 (YP_004875421.1) - серил-тРНК-синтетаза, misc РНК №46 (YP_004878330.1) - аспартаткиназа, для остальных это гипотетический белок.
Видимо, белки YP_004875421.1 и YP_004878330.1 аннотированы неправильно.
Зависимость времени работы blastn от длины затравки
Чтобы узнать время работы программы, используется команда time, которая пишется перед командной строкой. XX - длина затравки для blastn. Было выполнено для длин от 4 до 16.
time blastn -task blastn -query misc_rna.fasta -db NC_011969.fna -out blast.txt -outfmt 7 -word_size XX
На рис. 1 показан график зависимости десятичного логарифма времени от длины затравки. При длине от 4 до 8 зависимость близка к линейной, то есть время спадает экспоненциально. При длине больше 8 время работы blastn приближается к 0.5 секундам.
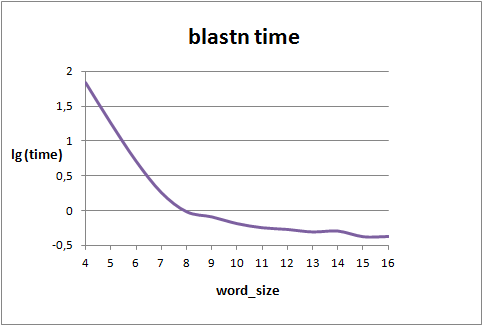
Рис. 1. Зависимость логарифма времени работы blastn от длины затравки