| Главная | Семестры | Обо мне | Ссылки |
|---|
Предсказание генов прокариот
Дан контиг contig.txt микробиома кардиального отдела желудка болотного валлаби Wallabia bicolor. Задача – при помощи программ getorf, BLASTP и GeneMark проаннотировать этот фрагмент: определить границы белок-кодирующих генов, по возможности определить их функцию и обосновать свои выводы.
Использование getorf
Будем искать открытые рамки считывания длиной от 60 триплетов, используя бактериальный генетический код (11).
getorf contig.txt -minsize 180 -table 11
Результат:
>Vasyutkina_1 [92 - 1315] VTPKYSTWKQIMSNLDGSNQQNTENTPKDVEVVTTESSKFDDIRHYRNDEVPNAIQSILN NDQVISGISGYMFKRVPKFLSFIVKPFVKAVLKKKFAKVKTIREVQEYVAQFMNGIIRDT TDGFTYSGFDKLDKNKGYLFISNHRDISLDPAFINMACFAEDLDTVKIAIGDNLLRMPVA TDLMKLNKSFIVKRSISSPKEKLKAFSELSEYIGLAIKENHNVWIAEREGRAKDGNDIAE AAIMKMFYIYGKKQGFSFKDYIKQLNIVPVSITYEYDPRDIQKARELYESAQNGEYIKSE FEDIESIVGGITGYKGHVHISAGAPLCGDYENAQELADEIDRYVHKHYRMFPSILAAANV TENLDPKDKEKFEKHMAEVPQELKEIVRSMYAKAYENNQKAQTIQAKH >Vasyutkina_2 [2635 - 2880] GLWKINCSSFFKSPHKWGCAPGLQHVDIKISTVLLCISFMMSLYLWVRNALTLYLLYLKI YNDLTFKLLNVNISLIPLRLWF >Vasyutkina_3 [2665 - 1487] (REVERSE SENSE) KKMNNLFSTNLNYQKLVSQFGSPLLILDKATIRYQYLALHKALPNVTLHYALKPLPLNTV VSVLKELGSSFDLASNGEVDIVKSANIDPKTCIHTHPIKKDQDIKYALEYGCNVFVYDNE TELEKFTKYKDQVKLLLRVSFPNPETKVDLSKKFGTTPENVLNLLQKAKDKGFNIYGLSF HVGSQVPNSKRHVEAITSCNNLINQAQDLGINISVLDIGGGFPVDYQNAEPIDIDSFCAP IREALKNTPKNVQILAEPGRFISAPAMHNICTVTGISRRFDKNWYYLDDGVYCSYSGQIF DHVCYPKFTPYNSHINVESCVLAGPTCDSIDVIAEDIKLPKLELNDLLVGKMMGAYTIAT ATEFNFIPKSKIIELDLADSSSTIYTEELLNAA >Vasyutkina_4 [750 - 523] (REVERSE SENSE) FSLIAKPMYSLNSENAFNFSFGELIERLTIKLLFNFIKSVATGILNKLSPIAIFTVSKSS AKQAIFIKAGSNEMSL >Vasyutkina_5 [465 - 280] (REVERSE SENSE) VNPSVVSRIIPFINCATYSCTSRIVFTLANFFLSTAFTKGFTMKDKNLGTLLNMYPEIPL IT
Для каждой из рамок программой blastp на сайте NCBI был проведен поиск гомологов по банку swissprot. Дополнительный параметр - поиск только бактериальных последовательностей. [Organism: bacteria (taxid:2)]
Будем считать последовательности близкими, если E-value находки меньше 0.001 и не менее 80% банковской последовательности покрыто выравниванием. Только для рамки №3 были найдены последовательности с такими параметрами. Для рамки №1 обнаруживается одна последовательность с E-value 0.001, но выравнивание покрывает ее не более чем на 40%. Данные о находке приведены в таблице 1. Организмы из рода Selenomonas обнаружены в ЖКТ жвачных животных (источник). Это подтверждает правильность находки.
Использование GeneMark
Контиг был обработан программой GeneMark. Она использует эвристические алгоритмы. Дополнительный параметр - запрос файла выдачи в формате PDF. В результате был получен файл с данными о найденных генах gmhmmp.out, а также график кодирующего потенциала, который показан на рис. 1. Горизонтальная ось графика - это номер (позиция) каждой буквы контига, а вертикальная ось показывает, какова вероятность у этой позиции кодировать ген. Жирные горизонтальные полосы выделяют области с высоким кодирующим потенциалом, то есть те, которые, возможно, являются генами. Данные о предсказанных генах приведены в таблице 1.
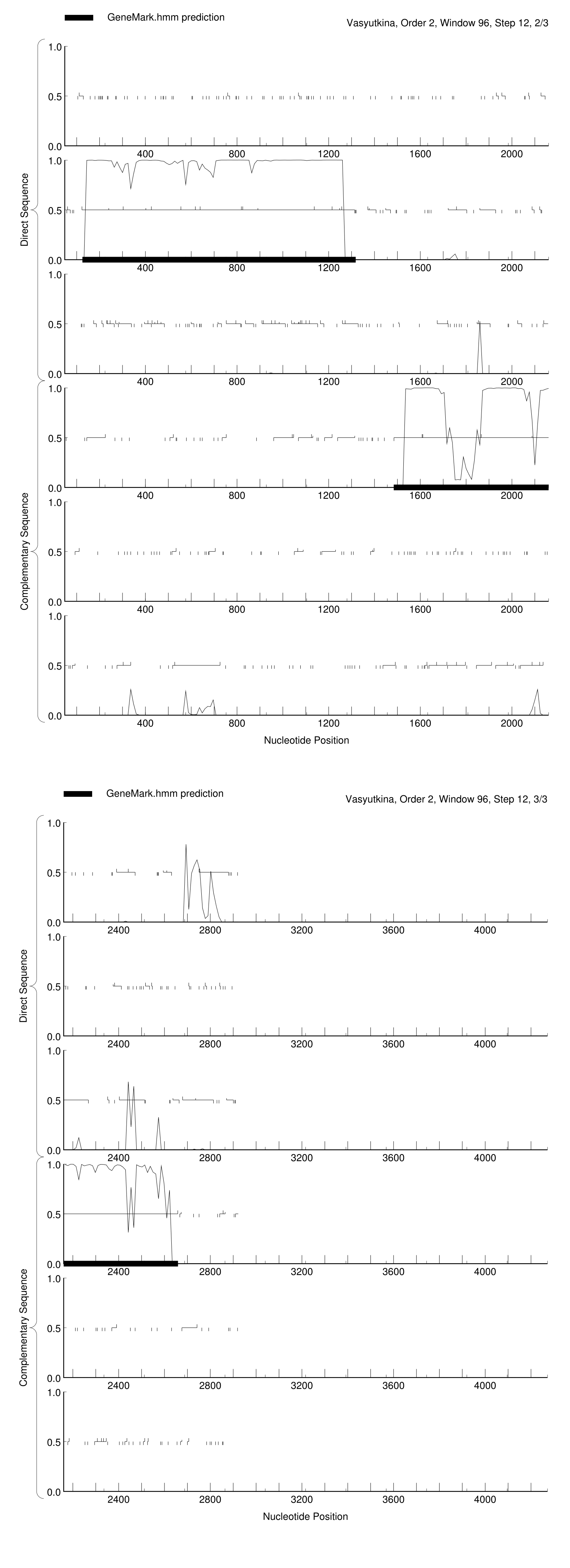
Рис. 1. График кодирующего потенциала данного контига. Получено с помощью GeneMark
Сравнение предсказаний
Таблица 1. Гены, найденные в контиге
| Программа | Начало | Конец | Длина в а.о. | Цепь | Описание | Идентификатор гомолога | Организм | E-value | Score |
|---|---|---|---|---|---|---|---|---|---|
| getorf; blastp | 1487 | 2665 | 393 | - | лизин/орнитин декарбоксилаза | O50657 | Selenomonas ruminantium | 1,00E-70 | 232 |
| GeneMark | 125 | 1318 | 398 | + | |||||
| GeneMark | 1484 | 2659 | 392 | - |
Всего программой GeneMark предсказаны 2 возможных гена, они соответствуют рамкам №1 и №3 с незначительным сдвигом. Также на рис. 1 можно заметить остальные рамки считывания, найденные getorf, в этих областях кодирующий потенциал существенно больше 0. Но он недостаточно высок для того, чтобы программа GeneMark считала эти области возможными генами. Из имеющихся данных можно сделать вывод, что GeneMark успешно находит области, где высока вероятность нахождения гена. Однако нельзя утверждать, что для найденных областей обязательно найдутся гомологи с высоким сходством. В целом, удобнее использовать GeneMark для поиска наиболее вероятных кодирующих областей, чем перебирать все найденные getorf рамки считывания.
Использование GeneMark с другими эвристическими параметрами
Есть 2 варианта работы GeneMark: с эвристическими параметрами 1999 и 2010 года. В этой работе использовались более старые параметры. Результат работы GeneMark с параметрами 2010 года: gmhmmp2010.out, файл PDF. Для данного контига изменений нет. Я думаю, для сравнения результатов нужно подобрать другой файл со "спорными" областями.