| Главная | Семестры | Обо мне | Ссылки |
|---|
Филогенетическое дерево бактерий
Цель занятия: поработать с филогенетическим деревом нескольких бактерий, построить собственное дерево на основе данного. В таблице 1 приведены названия и мнемонические обозначения видов бактерий.
Таблица 1. Отобранные бактерии
| Мнемоника | Название |
|---|---|
| CLOTE | Clostridium tetani |
| CLOB1 | Clostridium botulinum |
| LACAC | Lactobacillus acidophilus |
| ENTFA | Enterococcus faecalis |
| STRP1 | Streptococcus pyogenes |
| STRPN | Streptococcus pneumoniae |
| BACAN | Bacillus anthracis |
| GEOKA | Geobacillus kaustophilus |
Скобочная формула дерева
((CLOTE:4,CLOB1:4):1,((LACAC:3,(ENTFA:2,(STRP1:1,STRPN:1):1):1):1,(BACAN:3,GEOKA:3):1):1);
Изображение дерева приведено на рис. 1.
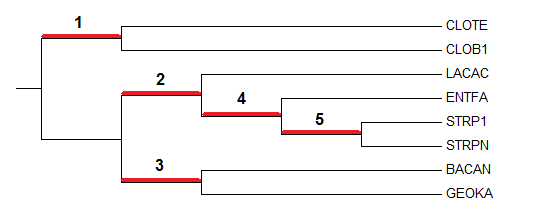
Рис. 1. Филогенетическое дерево отобранных бактерий
Ветви дерева
Нетривиальные ветви дерева разбивают множество листьев на подмножества, в каждом из которых более одного элемента. В дереве, представленном на рис. 1, пять нетривиальных ветвей:
- {CLOTE, CLOB1} против {LACAC, ENTFA, STRP1, STRPN, BACAN, GEOKA}
- {LACAC, ENTFA, STRP1, STRPN} против {CLOTE, CLOB1, BACAN, GEOKA}
- {CLOTE, CLOB1, LACAC, ENTFA, STRP1, STRPN} против {BACAN, GEOKA}
- {ENTFA, STRP1, STRPN} против {CLOTE, CLOB1, LACAC, BACAN, GEOKA}
- {STRP1, STRPN} против {CLOTE, CLOB1, LACAC, ENTFA, BACAN, GEOKA}
Реконструкция филогении
Вначале определим, к каким таксонам относятся выбранные бактерии. Для этого использовался сервис Taxonomy на сайте NCBI. Данные приведены в таблице 2.
Нетривиальная ветвь 1 отделяет класс Clostridia от класса Bacilli. Ветвь 2 относится к порядку Lactobacillales,а ветвь 3 - к порядку Bacillales. Ветвь 5 выделяет семейство Streptococcaceae.
Таблица 1. Отобранные бактерии
| Мнемоника | Название | Класс | Порядок | Семейство |
|---|---|---|---|---|
| CLOTE | Clostridium tetani | Clostridia | Clostridiales | Clostridiaceae |
| CLOB1 | Clostridium botulinum | Clostridia | Clostridiales | Clostridiaceae |
| LACAC | Lactobacillus acidophilus | Bacilli | Lactobacillales | Lactobacillaceae |
| ENTFA | Enterococcus faecalis | Bacilli | Lactobacillales | Enterococcaceae |
| STRP1 | Streptococcus pyogenes | Bacilli | Lactobacillales | Streptococcaceae |
| STRPN | Streptococcus pneumoniae | Bacilli | Lactobacillales | Streptococcaceae |
| BACAN | Bacillus anthracis | Bacilli | Bacillales | Bacillaceae |
| GEOKA | Geobacillus kaustophilus | Bacilli | Bacillales | Bacillaceae |
Построим филогенетическое дерево данных бактерий на основе сродства последовательностей белка шаперонина (мнемоника HSLO). Для выравнивания я использовала алгоритм Muscle. Дерево было реконструировано методом "Neighbour Joining Using % Identity".
Выравнивание последовательностей белков из семейства шаперонинов данных бактерий показано на рис. 2.
Реконструированное дерево представлено на рис. 3.
Скобочная формула полученного дерева (в формате Newick):
(((((CLOTE:11.700001,CLOB1:13.299999):15.513889,(GEOKA:17.6875,BACAN:16.645836):6.4861107):3.052084,ENTFA:20.197916):0.96875,(STRPN:12.194443,STRP1:11.805557):19.34375):13.828125,LACAC:13.828125);
Проект JalView: загрузить.
Выравнивание в формате FASTA: загрузить.

Рис. 2. Выравнивание последовательностей белков из семейства шаперонинов выбранных бактерий
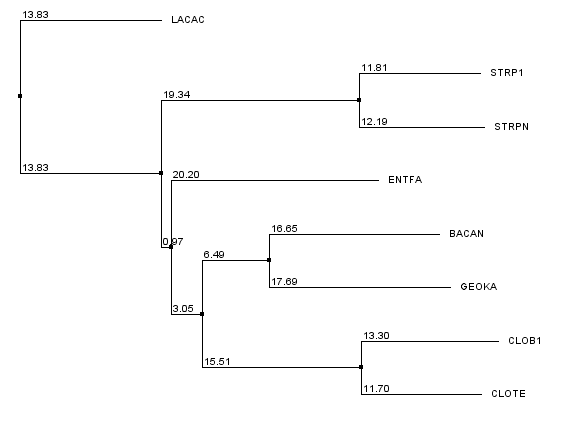
Рис. 3. Филогенетическое дерево выбранных бактерий, полученное на основе выравнивания белковых последовательностей
Укоренение в среднюю точку
С помощью программы retree пакета PHYLIP удалось укоренить в среднюю точку реконструированное дерево. За корень принимается середина самой длинной ветви.
На рис. 4 показано изображение дерева до укоренения, на рис. 5 - после. Рис. 4 повторяет рис. 3, но получен с помощью программы MEGA для удобства сравнения. В свою очередь, на рис. 3 приведены длины ветвей, в отличие от рис. 4.
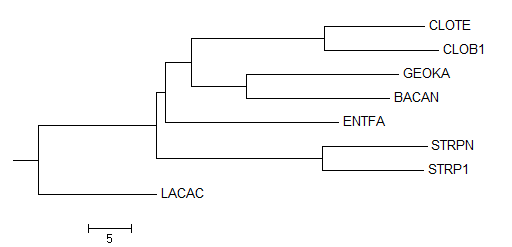
Рис. 4. Филогенетическое дерево выбранных бактерий. Получено с помощью MEGA
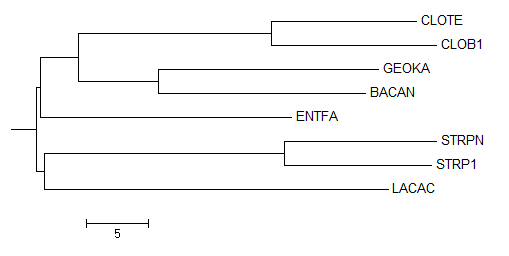
Рис. 5. Укорененное филогенетическое дерево выбранных бактерий. Получено с помощью MEGA
Анализ реконструкции деревьев
В целом, реконструированное дерево (рис. 3) получилось очень близким к исходному (рис. 1). Единственное различие заключается в том, что в исходном дереве ветвь с бактериями STRP1 и STRPN (семейство Streptococcaceae) образует узел вначале с ENTFA (семейство Enterococcaceae), а потом с LACAC (семейство Lactobacillaceae). В реконструкции дерева ветвь STRP1 и STRPN соединяется с LACAC, а потом с ENTFA.
Укоренение произошло в ветвь {LACAC, STRP1, STRPN} против {CLOTE, CLOB1, ENTFA, BACAN, GEOKA}. Судя по длинам ветвей, приведенным на рис. 3, это действительно середина самой длинной ветви. Корень расположен в неправильном месте, так как он разделяет бактерии BACAN, GEOKA, ENTFA, и LACAC, STRP1, STRPN, принадлежащие к одному и тому же классу Bacilli, но при этом объединяет одной ветвью CLOTE, CLOB1 и BACAN, GEOKA, которые относятся соответственно к классам Clostridia и Bacilli.