| Главная | Семестры | Обо мне | Ссылки |
|---|
Построение и визуализация электронной плотности
В данной работе продолжается работа над белком репарации и рекомбинации RadA из археи Methanococcus voltae, идентификатор PDB 3NTU. Данные о нем удовлетворяют всем критериям выбора объекта изучения в этом семестре. С сервера EDS была скачана карта электронной плотности белка в формате по умолчанию (O и 2mFo-DFc). Разрешение структуры составляет 1.90 Å.
Электронная плотность (ЭП) для мономера белка RadA показана на рис. 1. Боковые цепи на рисунке не изображены. Для визуализации ЭП в PyMOL использовались следующие команды:
load 3ntu.pdb, 3ntu
load 3ntu.omap, 3ntu_map #чтобы объекты получили разные имена
isomesh new_map, 3ntu_map, 1.5, backbone, 2.5, carve=2.5
Здесь:
new_map # имя нового объекта с изображением поверхности
3ntu_map # имя объекта с электронной плотностью
1.5 # уровень - значения электронной плотности, по которым строится поверхность
backbone # множество, электронную плотности вокруг которого надо изобразить,
в данном случае - только полипептидный остов
2.5 # (необязательный) аргумент – "buffer". Если его не задать, то будет изображена электронная
плотность в наименьшем параллелепипеде (со сторонами, параллельными координатным осям), в
который помещаются центры атомов данного множества. Если же задать ему значение, скажем, 2.5,
то этот параллелепипед будет расширен на 2.5 ангстрема в каждую сторону.
carve=2.5 # параметр подрезки, чтобы отсекать сигнал от соседних атомов. Это расстояние от выбранного
множества атомов, в пределах которого будет учитываться электронная плотность.
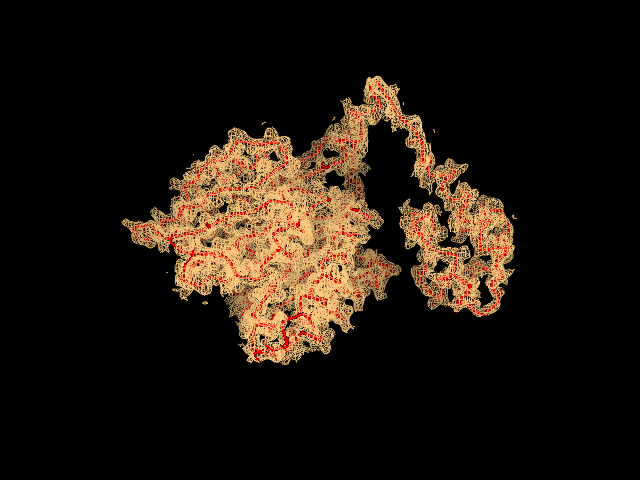
Рис. 1. Электронная плотность для полипептидной цепи белка RadA. Получено с помощью PyMOL
Изображение ЭП вокруг трех аминокислотных остатков
Построим изображение ЭП для остатков, которые участвуют в связывании АТФ (они изучены в работе 2-го семестра).
Это LYS 111, THR 112, GLN 113. Изображение приведено на рис.2. Значения порога ЭП от 0.5 до 2.5.
Команды:
select resi 111-113 # по умолчанию выделенный сет атомов называется "sele"
show sticks, sele # Палочная модель. Выглядит как вежливое обращение к выделенному множеству
util.cbaw sele # util.cba* красит по типу атомов, w от white - цвет атомов углерода
isomesh new_map, 3ntu_map, 0.5, sele, 2.5, carve=1.5 #посмотрим ЭП на разных уровнях подрезки
isomesh new_map, 3ntu_map, 1.0, sele, 2.5, carve=1.5
isomesh new_map, 3ntu_map, 1.5, sele, 2.5, carve=1.5
isomesh new_map, 3ntu_map, 2.0, sele, 2.5, carve=1.5
isomesh new_map, 3ntu_map, 2.5, sele, 2.5, carve=1.5
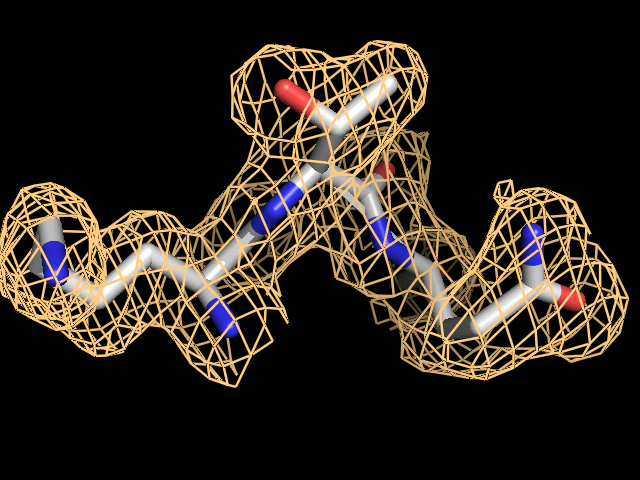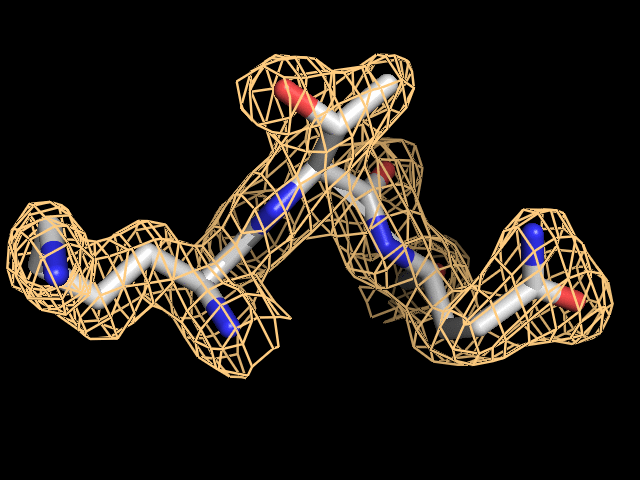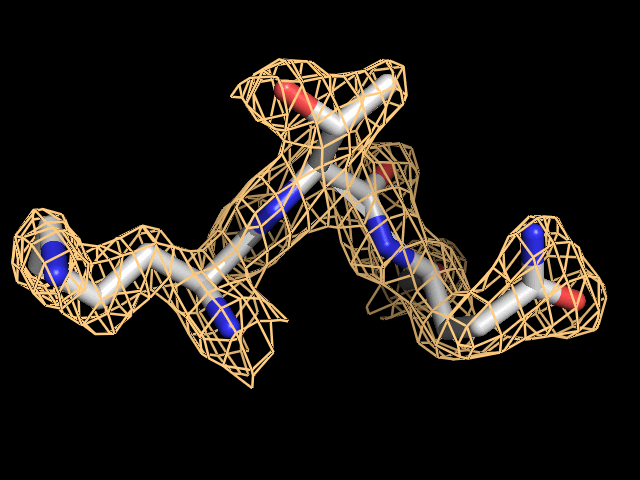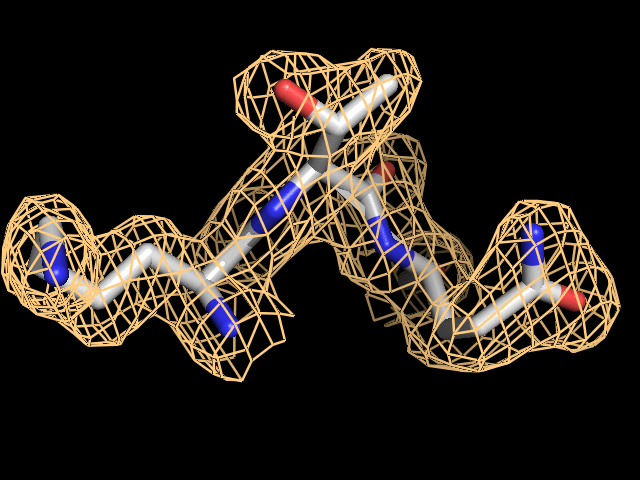
Рис. 2. Электронная плотность вокруг аминокислотных остатков LYS 111, THR 112, GLN 113.Получено с помощью PyMOL и веб-сервиса Gifmaker.me
Заключение
На основании визуализации электронной плотности вокруг полипептидной цепи (рис. 1) и отдельных аминокислотных остатков (рис. 2) можно заключить следующее: разрешение структуры 3NTU не является достаточным для того, чтобы наблюдать отдельные атомы на изображении карты электронной плотности, однако по этой карте виден ход полипептидной цепи и угадывается положение боковых групп аминокислот.