Скрипт:
select all; cartoon on; cpk off; wireframe off; color cpk;
pause;
restrict dna; cartoon off; wireframe on;
pause;
select *.O?'; cpk 200; color red;
select *.OP?; cpk 150; color gold;
select *.N? and dna; cpk 150;
Данная работа посвящена описанию межмолекулярных взаимодействий между ДНК и белком на примере комплекса 1PP7.
Будем рассматривать неполярное и полярное взаимодействия.
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å .
Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å .
Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
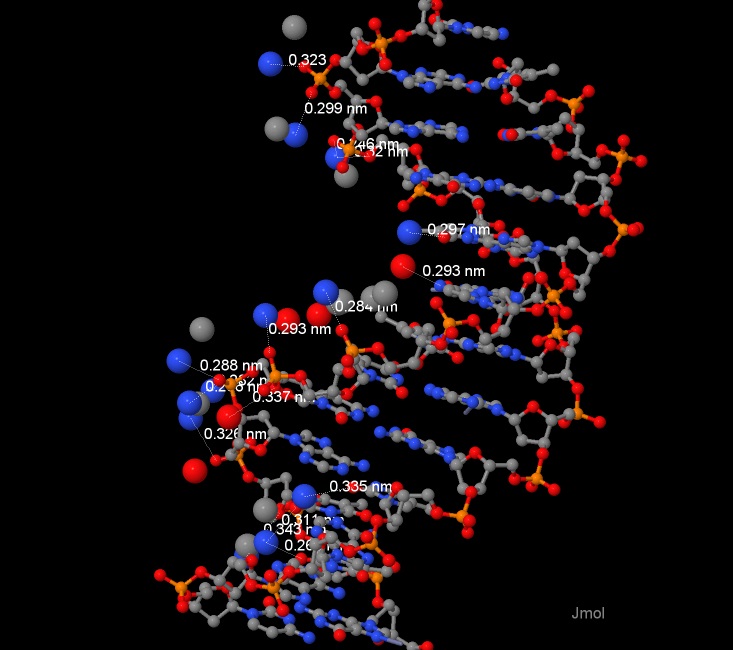
Рис 1. Взаимодействия между полярными атомами ДНК и белка (показаны пунктиром).
 |
 |
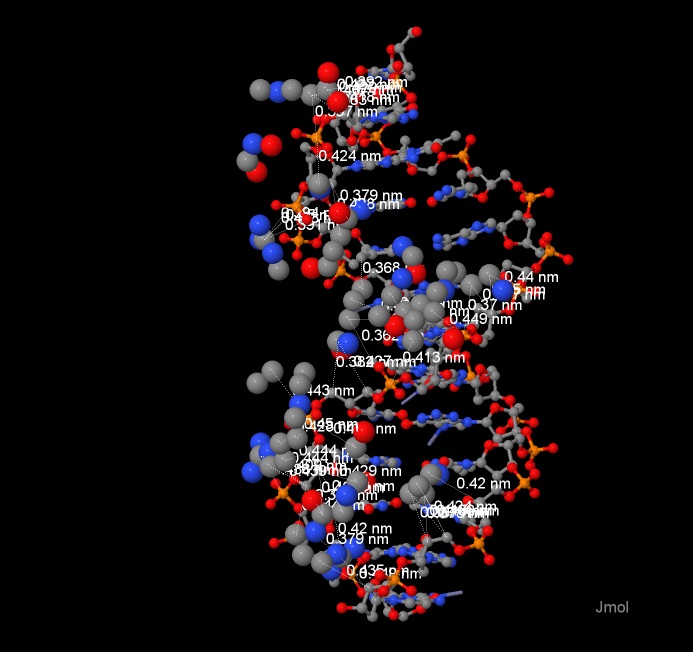
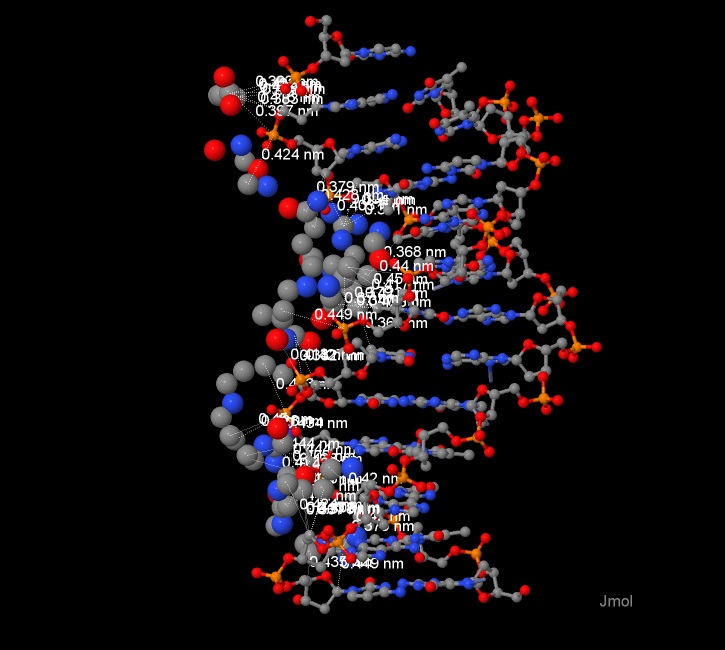
| Рис 2,3. Взаимодействия между неполярными атомами ДНК и белка (показаны пунктиром). |
Таблица 1. Количество контактов разного типа между ДНК и белком.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 3 | 34 | 37 |
| остатками фосфорной кислоты | 9 | 12 | 21 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 6 | 8 |
| остатками азотистых оснований со стороны малой бороздки | 3 | 1 | 4 |
Как видно, неполярных взаимодействий гораздо больше чем полярных. Вероятно это связано с большим числом атомов углерода в белке, чем азота или кислорода.
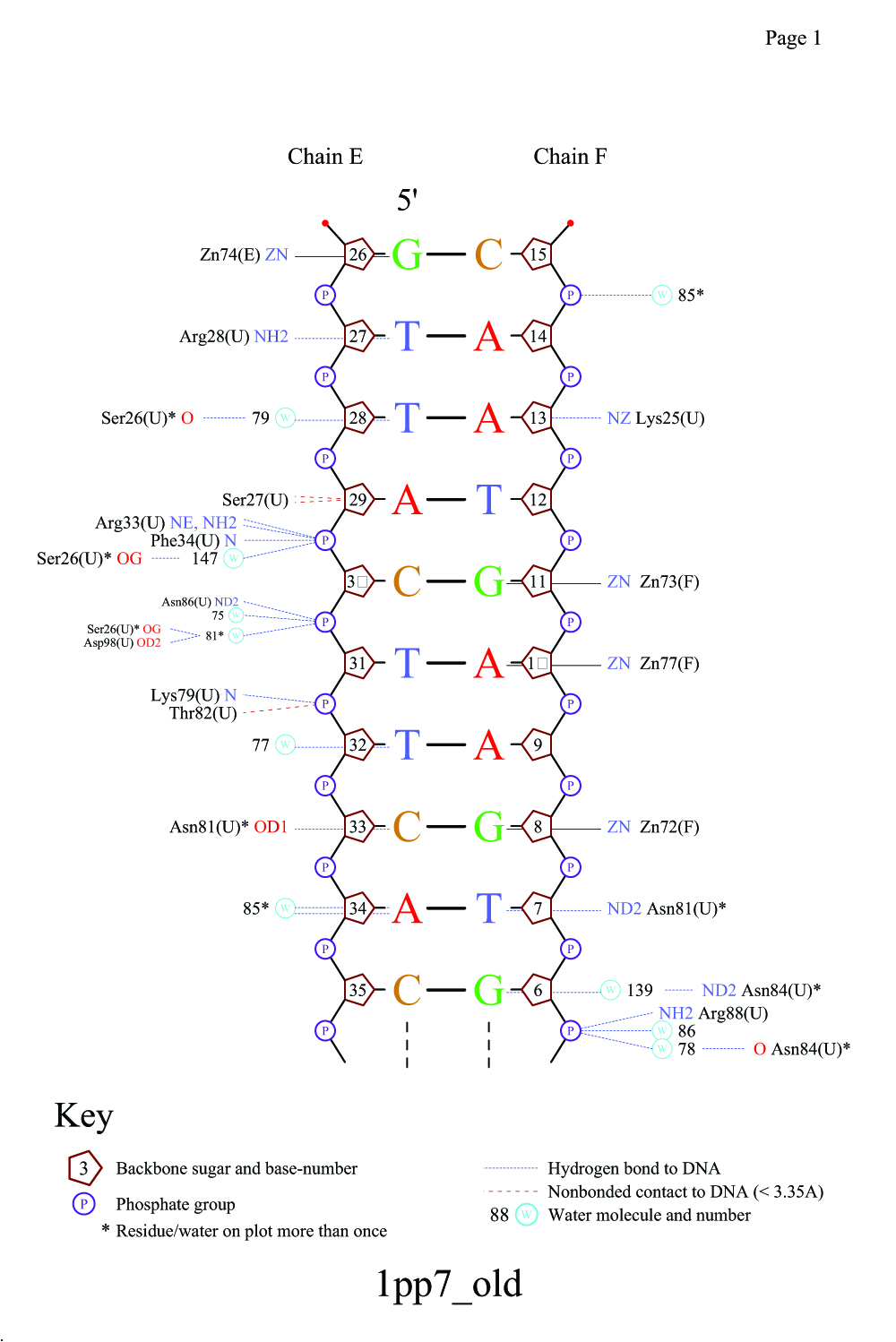
Рис 4. Схема взаимодействий белка и ДНК.
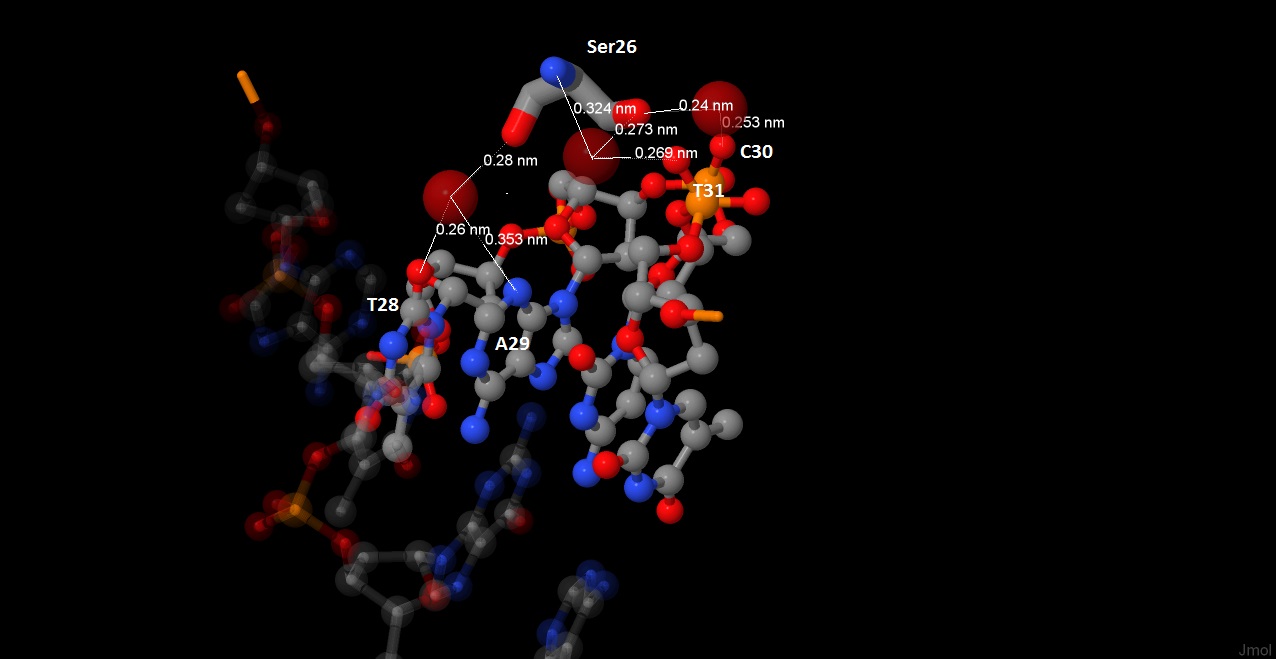
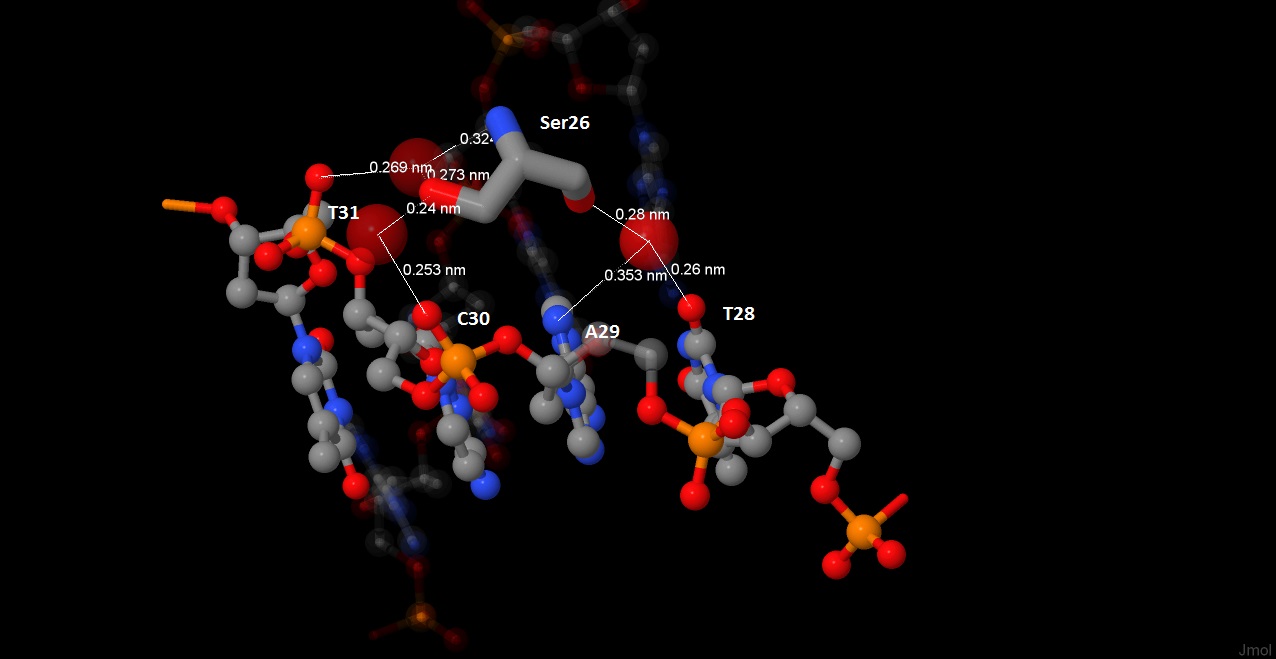
Как видно из схемы, наибольшее число взаимодействий с ДНК имеет Ser26 (с 28, 30 и 31 нуклеотидами). Хотя часть из них опосредована через молекулу воды, все-равно создается жесткая структура координированных связей. Логично предположить, что именно эта аминокислота важна для распознавания необходимой последовательности ДНК.
 |
 |
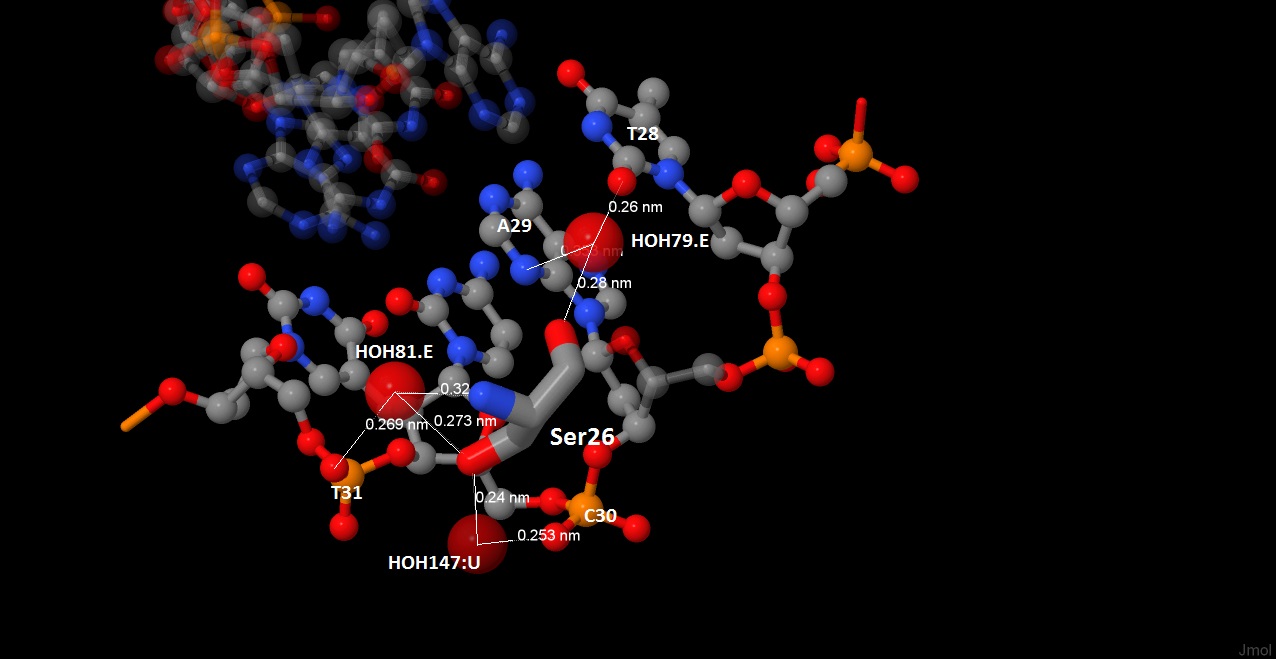 |
Рис 5-8. Взаимодействие Ser26 с ДНК. Линиями показаны минимальные расстояния |
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях.
При помощи данной программы велся поиск повторов для исследуемой тРНК.
На вход программе необходимо подать фаста-последовательность РНК и задать ряд параметров:
штраф за гэп, минимальный вес находок, бонус за пару, штраф за мисмэтч.
При подборе параметров, наилучшим результатом оказался следующий:
fasta: Score 84: 27/27 (100%) matches, 26 gaps
1 gggca-----gg-ttc---ccgagcggccaaaggg-----g---acgg-tctg 35
||||| || ||| || ||| | || || | |||| | |
82 cccgtcccgacctaagcttgg-tcg-c---tt-ccgtatgcggttgccaa-aa 37
Однако (см таблицу 1), даже этот вариант на отражает реальной структуры РНК и правильно находит только 2 стебля из 4.
Structure 2
Folding bases 1 to 86 of fasta
Initial dG = -32.20
10
---- .-UU| GAGCG
GGGCAGG CCC \
CCCGUCC GGG G - Акцепторная и D - петли
ACCA \ --^ AAACC
80 20
30
.-G CUG
ACGGU \ - Антикодоновая петля
UGCCA U
\ - AAA
40
50
.-U G
GGC U - Вариабельная петля
CCG A
\ - U
60
UUC UUC
GCUGG \
CGACC G - Т - петля
--- UAA
70
В таблице 1 сравниваются данные по структуре тРНК, полученные разными способами. Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
5'-1-7-3' |
5'-1-5-3' |
5'-1-7-3' |
D-стебель |
5'-10-12-3' |
0 из 3 реальных |
5'-10-12-3' |
T-стебель |
5'-49-53-3' |
0 из 5 реальных |
5'-49-53-3' |
Антикодоновый стебель |
5'-26-31-3' |
5'-27-30-3' |
5'-27-31-3' |
Общее число канонических пар нуклеотидов |
24 пары |
27 пар |
23 пары |