Командная строка:
getorf seq.fasta -minsize 90 -table 0 -find 1
seq.fasta - название файл с последовательностью нуклеиновой кислоты.
-minsize 90 - задаем минимальную длину orf - 90 нуклеотидов.
-table 0 - выбираем стандартный генетический код.
-find 1 - выбираем нужный тип поиска: orf начинается со старт-кодона и заканчивается стоп-кодоном.
Программа по умолчанию транслирует все найденные orf.
В результате мы получили набор orf:
>Sequence_1 [66 - 155] MQFHPRLPAVLQVCAACDRYASLLPAQRRL >Sequence_2 [56 - 169] MISDAVSSATASSASSLRSMRSVRQSFASSTAALTRWP >Sequence_3 [163 - 432] MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGA >Sequence_4 [218 - 3] (REVERSE SENSE) MLLRCSNCLNVNWKCIRAIWSKPPLSWQKTGVPIACCANLKHCWQSRMKLHRLSSPVTVT WCSQKTILLLSA >Sequence_5 [294 - 1] (REVERSE SENSE) MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGSПоследовательность номер 3 совпадает с последовательностью, аннотированной в записи D89965:
MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGAДоказательство выравниванием:
pr 1 MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQR 50
||||||||||||||||||||||||||||||||||||||||||||||||||
Sequence_3 1 MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQR 50
pr 51 GLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA 90
||||||||||||||||||||||||||||||||||||||||
Sequence_3 51 GLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA 90
Для проверки, была получена последовательность, на которую ссылалась запись D89965 - P0A7B8:>P0A7B8 MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTLFEL FERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPENDL IAIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKAЭта последовательность не имеет ничего общего с указанной orf в записи D89965. Однако, с помощью выравнивания этой последовательности против всех найденных orf, было показано, что она перекрывается с одной из них:
P0A7B8 1 MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGG 50
|||||||||||||||||||||||
Sequence_5 1 ---------------------------MKGNVKKVRRLYNDKVIAGFAGG 23
P0A7B8 51 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 100
||||||||||||||||||||||||||||||||||||||||||||||||||
Sequence_5 24 TADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVAD 73
P0A7B8 101 ETASLIITGNGDVVQPENDLIAIGSGGPYAQAAARALLENTELSAREIAE 150
|||||||||||||||||||||||||
Sequence_5 74 ETASLIITGNGDVVQPENDLIAIGS------------------------- 98
P0A7B8 151 KALDIAGDICIYTNHFHTIEELSYKA 176
Sequence_5 98 -------------------------- 98
Это означает, что 2 идентичные последовательности нуклеиновых кислот относятся к двум очень далеким организмам: крысе и кишечной палочки.
Более того, они кодируют различные белки: у крысы это рецептор серотонина, а у бактерии - АТФ-зависимая субъединица протеазы HslV.
Использованные команды:
entret sw:adh*_* > adh.txt - отбираем из базы данных SwissProt все записи, относящиеся к алкогольдегидрогиназе и записываем их в файл.
infoseq -only -usa > list.txt - экстрагируем из полученного файла названия записей (usa).
grep -f organisms.txt list.txt > onlyneed.txt - Отбираем из всех названий только те, в которых встречаются слова, записанные в файле
organisms.txt
seqret @onlyneed.txt seq.fasta - извлекаем из файла adh.txt только те последоваиельности, названия которых содержатся в файле onlyneed.txt
Ссылка на файл с последовательностями нужных алкогольдегидрогеназ
Ген: SUZ12_HUMAN
Прямой поиск выдал страницу с основной информацией о гене: расположение, координаты, структура, длина, варианты сплайсинга и т.п.

Рис 1. Экзон-интронная структура гена SUZ12_HUMAN.
Также на этой странице расположены ссылки на аннотацию гена и белка.
Если перейти на эти странички мы получаем последовательность гена и белка:
MAPQKHGGGGGGGSGPSAGSGGGGFGGSAAVAAATASGGKSGGGSCGGGGSYSASSSSSAAAAAGAAVLP VKKPKMEHVQADHELFLQAFEKPTQIYRFLRTRNLIAPIFLHRTLTYMSHRNSRTNIKRKTFKVDDMLSK VEKMKGEQESHSLSAHLQLTFTGFFHKNDKPSPNSENEQNSVTLEVLLVKVCHKKRKDVSCPIRQVPTGK KQVPLNPDLNQTKPGNFPSLAVSSNEFEPSNSHMVKSYSLLFRVTRPGRREFNGMINGETNENIDVNEEL PARRKRNREDGEKTFVAQMTVFDKNRRLQLLDGEYEVAMQEMEECPISKKRATWETILDGKRLPPFETFS QGPTLQFTLRWTGETNDKSTAPIAKPLATRNSESLHQENKPGSVKPTQTIAVKESLTTDLQTRKEKDTPN ENRQKLRIFYQFLYNNNTRQQTEARDDLHCPWCTLNCRKLYSLLKHLKLCHSRFIFNYVYHPKGARIDVS INECYDGSYAGNPQDIHRQPGFAFSRNGPVKRTPITHILVCRPKRTKASMSEFLESEDGEVEQQRTYSSG HNRLYFHSDTCLPLRPQEMEVDSEDEKDPEWLREKTITQIEEFSDVNEGEKEVMKLWNLHVMKHGFIADN QMNHACMLFVENYGQKIIKKNLCRNFMLHLVSMHDFNLISIMSIDKAVTKLREMQQKLEKGESASPANEE ITEEQNGTANGFSEINSKEKALETDSVSGVSKQSKKQKL

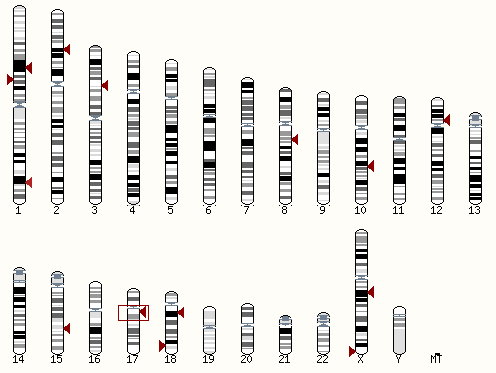
Рис 2. Расположение хитов на кариотипе человека.

Рис 3. Выравнивание хитов против запроса.
Данный ген интересен тем, что имеет 16 экзонов и относится к 17 хромосоме. Соответственно, отдельные экзоны были найдены как отдельные хиты.
Поэтому все лучшие хиты относились, как и следовало ожидать, к 17 хромосоме.
Однако, только к названной хромосоме относится более 35 хороших хитов, с высокой идентичностью и очень низким e-value, так что
вычленение из полученных данных настоящих экзонов - процесс нелегкий.
Если перейти по ссылке ContigView (символ [C] - левый столбец в списке хитов),
то мы попадем на страницу с иллюстрациями расположения
данного хита: вся хромосома, конкретная область и конкретный ген.

Рис 4. Расположение некоторого хита на 17 хромосоме.

Рис 5. Регион, к которому относится данный хит.
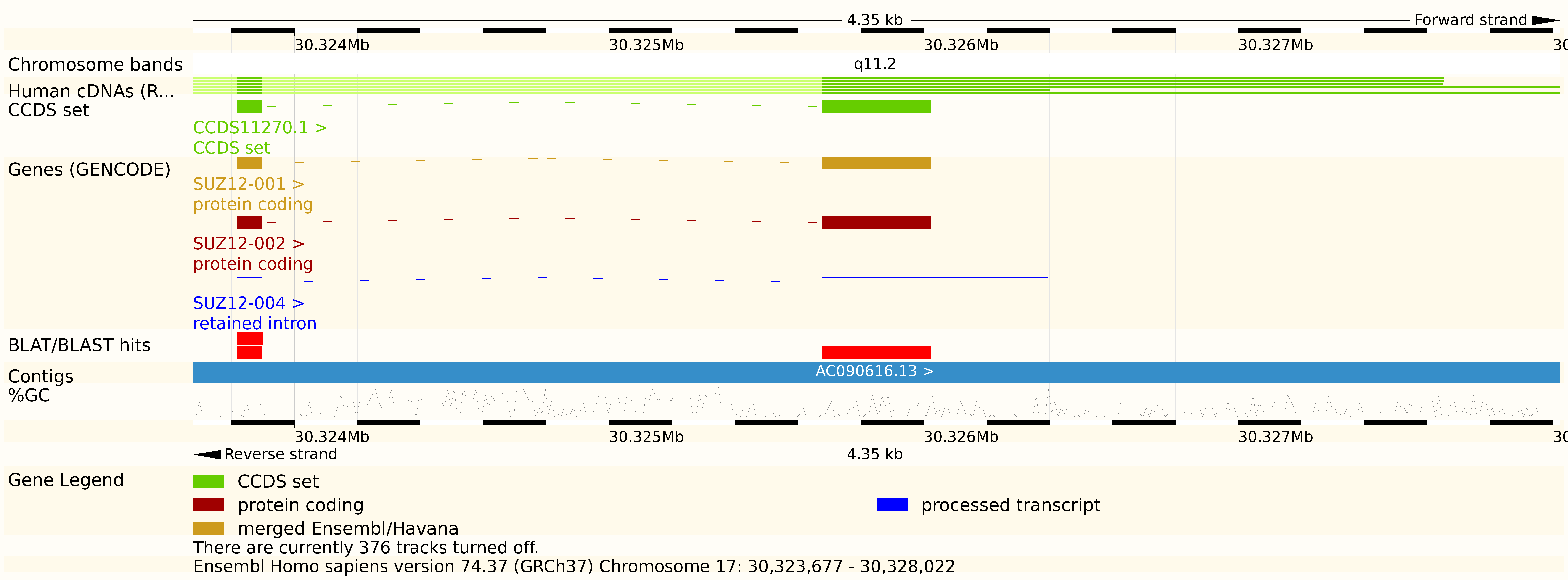
Рис 6. Локус хита.
Все картинки - интерактивные,- можно выбрать любую область, приблизить её, отдалить,
посмотреть процент GC.
Если щелкнуть на соседний значек [G] мы попадем на страничку с последовательностью хита.
Если щелкнуть на [A], мы получим выравнивания хита с запросом.