Отобранные бактерии
| Название | Мнемоника |
| Bacillus subtilis | BACSU |
| Clostridium botulinum | CLOB1 |
| Finegoldia magna | FINM2 |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus delbrueckii | LACDA |
| Lactococcus lactis | LACLM |
| Staphylococcus epidermidis | STAES |
| Streptococcus pneumoniae | STRPN |
Систематика бактерий:
Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia
Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group
Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus
Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus
Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus
Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus
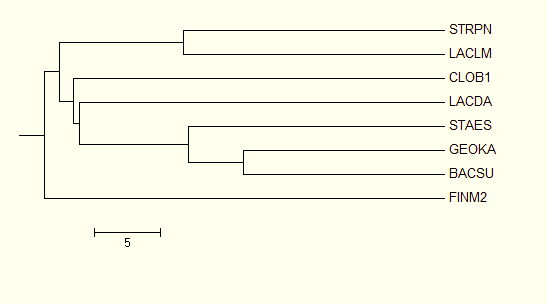
Как видно, дерево 1 отлично соотносится с данными систематики:
При помощи программы JalView было произведено выравнивание (Tcoffee) последовательностей гомологов-шаперонинов всех отобранных бактерий.
По выравниванию были построены деревья 1-4.
 | Дерево 1. Построено алгоритмом Average Distance Using % Identity |
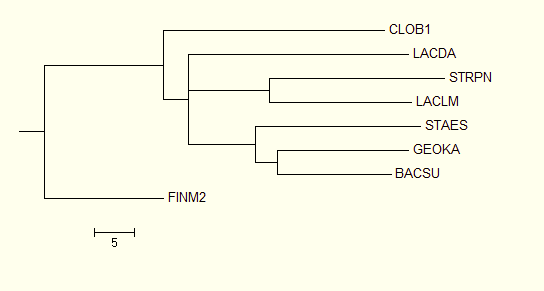
 | Дерево 2. Neighbour Joining Using % Identity |
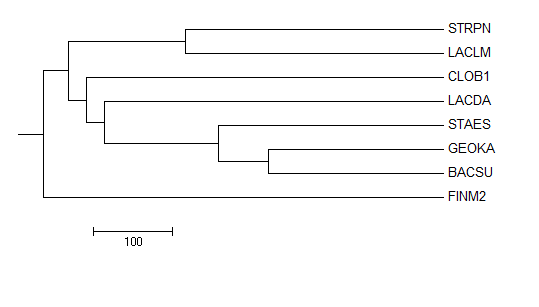
 | Дерево 3. Average Distance Using BLOSUM62 |
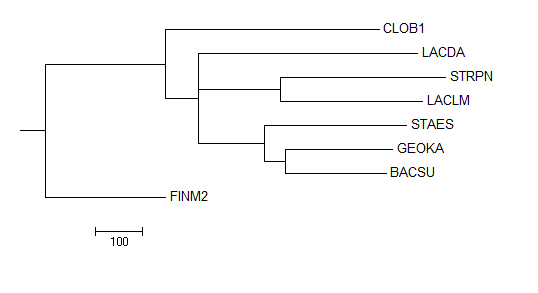
 | Дерево 4. Neighbour Joining Using BLOSUM62 |
Отличия сконструированных деревьев от правильного:
В программе Mega можно провести анализ полученного в Jalview выравнивания методом Maximum Parsimony.
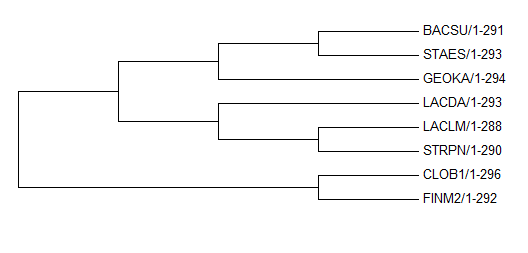
Полученое дерево отличается от правильного тем, что в нем нет ветви {BACSU, GEOKA},которая соответствует семейству Bacillaceae. При этом на обоих деревьях есть ветвь {BACSU, GEOKA, STAES}. Таким образом, листья STAES и GEOKA поменяны местами.