
Для нескольких трансмембранных белков, имеющих разную архитектуру трансмембранных участков, были определен ряд параметров (Таблица 1) при помощи БД (ОМР - Orientations of Proteins in Membranes).
| PDB код | Название белка | Тип (спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1xio | Сенсорный родопсин из Nostoc sp. | спираль | Внутренняя мембрана Грам-отрицательных бактерий | 31.9 ± 1.5 | 22 |
| 2m6b | Человеческий рецептор глицина | Спираль | Эукариотическая плазматическая мембрана | 26.8 ± 0.3 | 19 |
| 4bgn | Потенциал-чувствительный натриевый канал из Caldalkalibacillus thermarum | Спираль | Плазматическая мембрана Грам-положительных бактерий | 27.0 ± 0.3 | 20,5 |
| 1bxw | OMPA из E. coli | Баррель | Внешняя мембрана Грам-отрицательных бактерий | 25.4 ± 1.9 | 9,5 |
| 1uun | Порин MspA из Mycobacterium smegmatis | Баррель | Внешняя мембрана Грам-положительных бактерий | 40.7 ± 2.1 | 9,5 |
| 2jk4 | Человеческий потенциал-зависимый анионный канал | Баррель | Внешняя мембрана митохондрий | 23.4 ± 2.3 | 7,9 |
Таблица 1. Описание трансмембранных белков с известной 3D структурой.
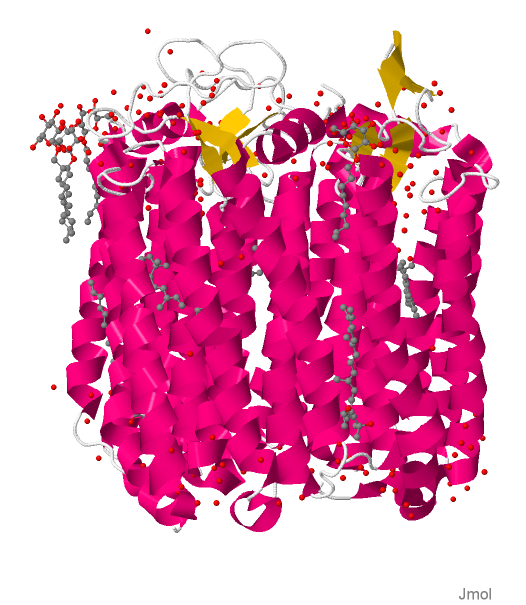
Белок с PDB кодом 3QBG, является трансмембранным белком - галородопсином из археи Natronomonas (Рис 1).

| 
|
| Рис 1А. Структура галородопсина, вид сбоку. n-сторона сверху. Молекулы детергента показаны серым цветом. | Рис 1Б. Структура галородопсина, вид сверху (с n-стороны). |
| PDB код | Название белка | Организм | ТС-код | Тип (спираль, баррель) |
Тип мембраны | Толщина гидрофобной части мембраны в ангстремах | Угол наклона спиралей к нормали | Медиана числа остатков в одном трансмембранном участке |
| 3QBG | Галородопсин | Natronomonas pharaonis | 3.E.1.2.2 | 7 cпиралей | - | 34.0 ± 1.0 | 1.± 1 | 22,3 |
Таблица 2. Информация о белке 3QBG.
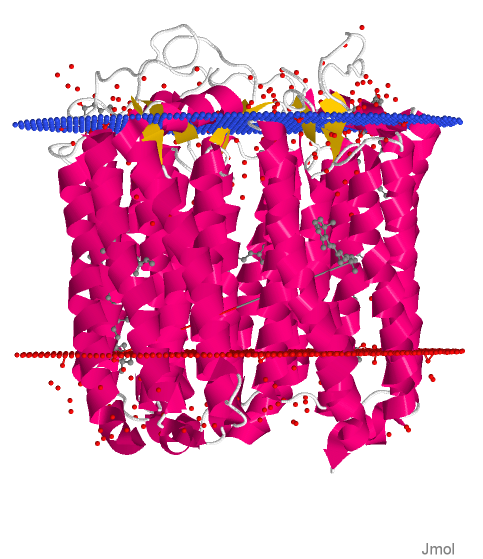
При помощи сервиса РРМ, по PDB структуре 3QBG были получены орентированные изображения галородопсина в мембранном бислое (Рис. 2).
| 
|
| Рис 2А. Структура галородопсина, вид сбоку. n-сторона сверху. Молекулы детергента показаны серым цветом. | Рис 3Б. Структура галородопсина, вид сверху (с n-стороны). Поверхность мембраны показана синим и красным слоями. |
Transmembrane_secondary_structure_segments: A 1(31-55),2(68-89),3(121-138),4(149-173),5(174-195),6(213-234),7(243-264)
При помощи blastp против БД RefSeq, с исключением домена Archaea, была создана репрезентативная выборка гомологов 3QBG.
Последовательности гомологов были выровнены программой Muscle (Выравнивание).
С выравниванием (с последовательностью галородопсина) была ассоциирована пространственная структура. Ориентируясь на структуру, в последовательности
были размечены трансмембранные участки спиралей (выравнивание, аннотация TM_REAL_Halorhodopsin).
Далее, случайно был выбран гомолог галородопсина - опсин из Sordaria macrospora. Для него при помощи сервиса TMHMM были предсказаны
трансмембранные участки, на основании последовательности белка (Таблица 3).
# XP_003349330.1_opsin Length: 307 # XP_003349330.1_opsin Number of predicted TMHs: 7 # XP_003349330.1_opsin Exp number of AAs in TMHs: 147.575 # XP_003349330.1_opsin Exp number, first 60 AAs: 13.23681 # XP_003349330.1_opsin Total prob of N-in: 0.24571 # XP_003349330.1_opsin POSSIBLE N-term signal sequence XP_003349330.1_opsin TMHMM2.0 outside 1 48 XP_003349330.1_opsin TMHMM2.0 TMhelix 49 71 XP_003349330.1_opsin TMHMM2.0 inside 72 77 XP_003349330.1_opsin TMHMM2.0 TMhelix 78 100 XP_003349330.1_opsin TMHMM2.0 outside 101 134 XP_003349330.1_opsin TMHMM2.0 TMhelix 135 152 XP_003349330.1_opsin TMHMM2.0 inside 153 156 XP_003349330.1_opsin TMHMM2.0 TMhelix 157 176 XP_003349330.1_opsin TMHMM2.0 outside 177 185 XP_003349330.1_opsin TMHMM2.0 TMhelix 186 208 XP_003349330.1_opsin TMHMM2.0 inside 209 220 XP_003349330.1_opsin TMHMM2.0 TMhelix 221 243 XP_003349330.1_opsin TMHMM2.0 outside 244 252 XP_003349330.1_opsin TMHMM2.0 TMhelix 253 275 XP_003349330.1_opsin TMHMM2.0 inside 276 307 |
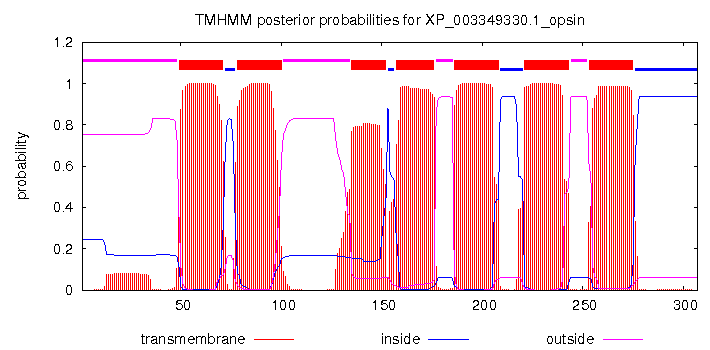 |
| Таблица 3. Предсказание трансмембранных участков сервисом TMHMM для опсина из Sordaria macrospora. | Рис. 3. Графическое представление предсказаний трансмембранных участков для опсина в TMHMM. Спирали соответствуют областям красных пиков. |
Предсказанные трансмембранные участки были размечены на множественном выравнивании (выравнивание, аннотация TM_PREDICTED_opsin).
Как видно, участки спиралей кажутся немного сдвинутыми относительно друг-друга, однако значительно перекрываются и нигде не
перекрываются с соседними выделеными регионами.
Для проверки правильности предсказаний сервисом TMHMM аналогичная операция была проделана с самим галородопсином (Таблица 4).
# WEBSEQUENCE Length: 291 # WEBSEQUENCE Number of predicted TMHs: 7 # WEBSEQUENCE Exp number of AAs in TMHs: 151.9029 # WEBSEQUENCE Exp number, first 60 AAs: 22.15864 # WEBSEQUENCE Total prob of N-in: 0.00027 # WEBSEQUENCE POSSIBLE N-term signal sequence WEBSEQUENCE TMHMM2.0 outside 1 31 WEBSEQUENCE TMHMM2.0 TMhelix 32 54 WEBSEQUENCE TMHMM2.0 inside 55 65 WEBSEQUENCE TMHMM2.0 TMhelix 66 88 WEBSEQUENCE TMHMM2.0 outside 89 119 WEBSEQUENCE TMHMM2.0 TMhelix 120 142 WEBSEQUENCE TMHMM2.0 inside 143 148 WEBSEQUENCE TMHMM2.0 TMhelix 149 171 WEBSEQUENCE TMHMM2.0 outside 172 176 WEBSEQUENCE TMHMM2.0 TMhelix 177 196 WEBSEQUENCE TMHMM2.0 inside 197 215 WEBSEQUENCE TMHMM2.0 TMhelix 216 238 WEBSEQUENCE TMHMM2.0 outside 239 247 WEBSEQUENCE TMHMM2.0 TMhelix 248 267 WEBSEQUENCE TMHMM2.0 inside 268 291 |
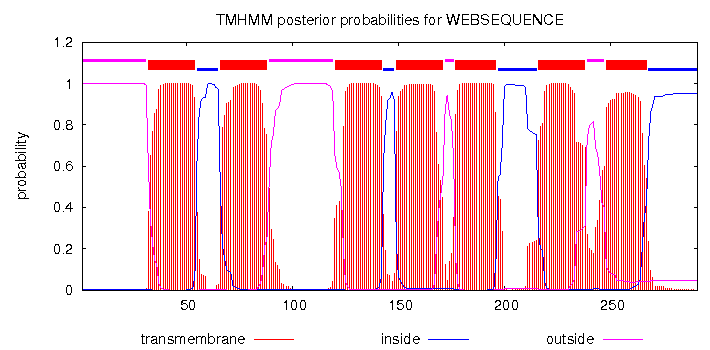 |
| Таблица 4. Предсказание трансмембранных участков сервисом TMHMM для галородопсина 3QBG из Natronomonas pharaonis. | Рис. 4. Графическое представление предсказаний трансмембранных участков для галородопсина в TMHMM. Спирали соответствуют областям красных пиков. |
На множественном выравнивании были отмечены указанные участки (выравнивание, аннотация TM_PREDICTED_Halorhodopsin).
Как видно, участки всех трех разметок хорошо повторяют друг-друга. Более того, нигде не происходит перекрывания соседних трансмембранных
участков. Из этого можно сделать вывод о том, что сервис TMHMM хорошо предсказывает трансмембранные участки.
Далее, выравнивание было раскрашено на гидрофобные аминокислоты с порогом консервативности 34%. Как видно из выравнивания,
консервативные блоки гидрофобных аминокислот отлично совпадают с размеченными трансмембранными спиралями.
Эта закономерность хорошо иллюстрируется пространственной структурой субъединицы А белка, к которой применена таже цветовая схема (Рис. 5).
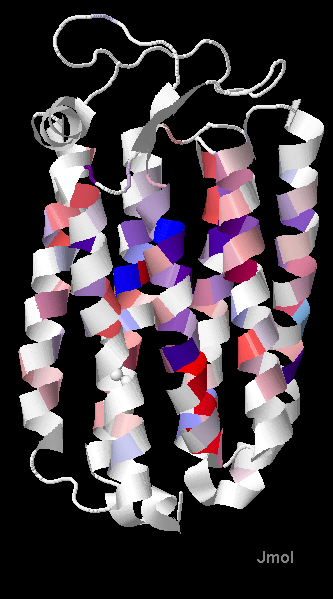
Рис. 5. Структура субъединицы белка 3QBG. n-сторона сверху. Консервативные гидрофильные аминокислоты выделены синим цветом, Консервативные гидрофобные,- красным.
Остальная структура - серая.
Как видно (Рис. 5), консервативные гидрофобные аминокислоты локализуются только в трансмембранной части белка.
Это важно для устойчивости белкового комплекса в гидрофобном окружении липидного бислоя.