Для цепи I из структуры 4TMA (является белком-ингибитором ДНК-гиразы YacG)
был проведен поиск структурных гомологов с помощью сервиса PDBeFold. Поиск проводился
с параметрами по умолчанию.
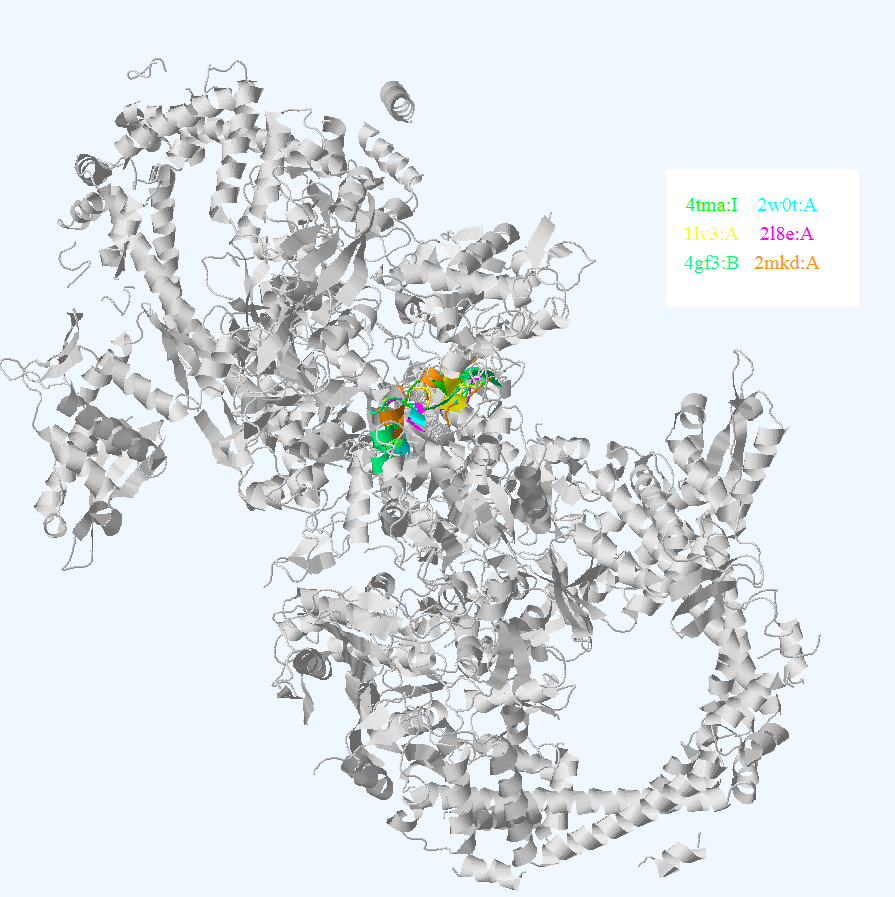
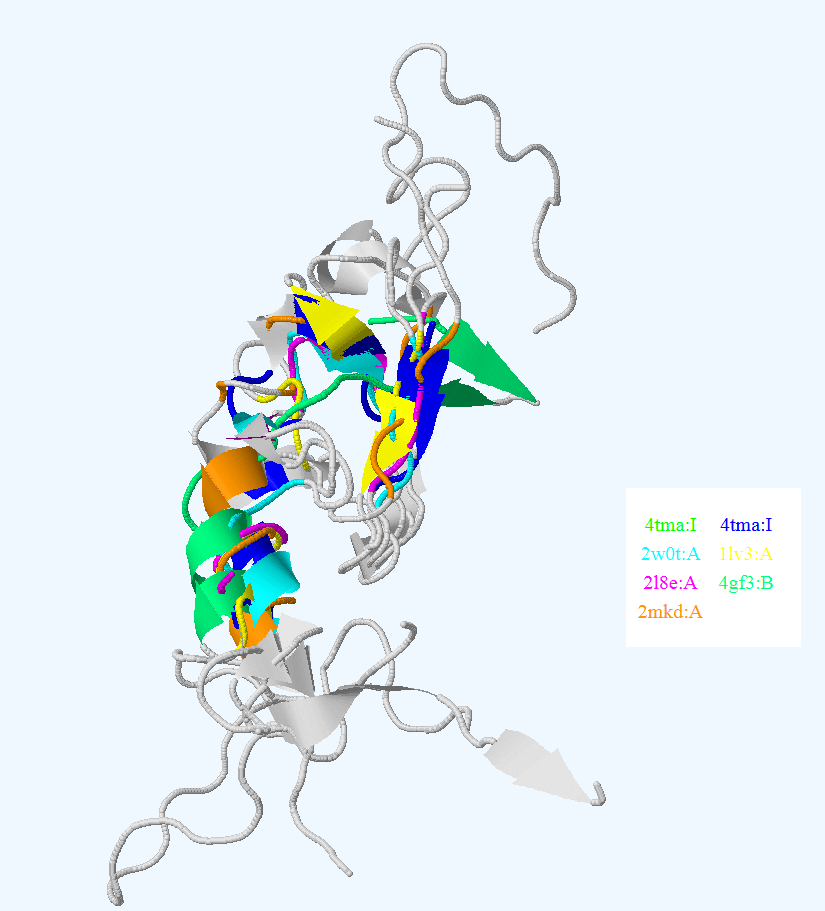
Из списка находок были отобраны 5 гомологов, которые были совмещены (Рис. 1).
 |
 |
| a | б |
Рисунок 1.Совмещение цепи I из структуры 4TMA с ближайшими гомологами. а. Совмещение полных структур; б. Совмещение выровненных участков
Можно сказать, что гомологи довольно хорошо выровнялись друг с другом - общая топология структуры хорошо прослеживается (Рис. 1а). PDB
файл с пространственным выравниванием можно скачать по ссылке.
Для пространственного совмещения было получено соответствующее ему множественное выравнивание последовательностей
(Рис. 2).
Отдельно были получены последовательности, соответствующие совмещенным участкам, которые затем были выровнены в программе JalVeiw
сервисом Muscle (параметры по умолчанию) (Рис. 2); выравнивание можно получить по ссылке.
Интересно, что полученные разными способами множественные выравнивания достаточно сильно отличаются друг от друга. Следует отметить, что выравнивание
Muscle содержит больше консервативных колонок.

Рисунок 2. Сравнение множественных выравниваний, полученных из пространственного совмещения структур и программой Muscle. Оранжевой рамкой выделена консервативная колонка 27 Phe белка YacG
Для проверки выравниваний была выбрана консервативная колонка 27 Phe белка YacG (Рис. 2, выделена рамкой). Данная аминокислота была найдена в нескольких цепях множественного пространственного выравнивания (Рис. 3). Очевидно, что показанные Phe выровнены друг с другом. Можно сделать вывод, что программа Muscle в данном случае сделала лучшее выравнивание.
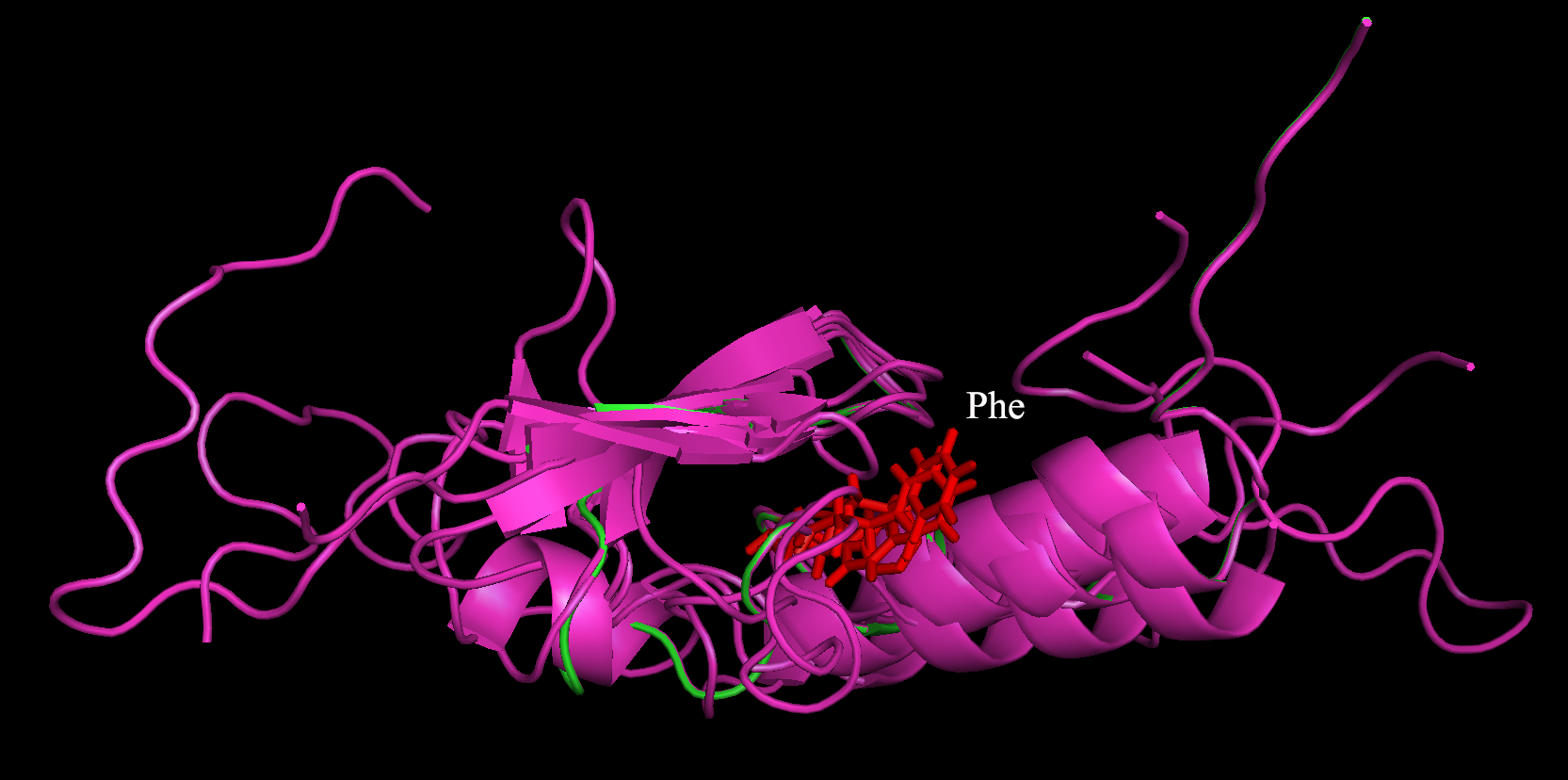
Рисунок 4. Пространственное совмещение гомологов YacG. Зеленым выделена цепь YacG, розовым - все остальные белки. Красным цветом с визуализацией sticks показан консервативный 27 Phe белка YacG и соответствующие ему Phe других цепей.
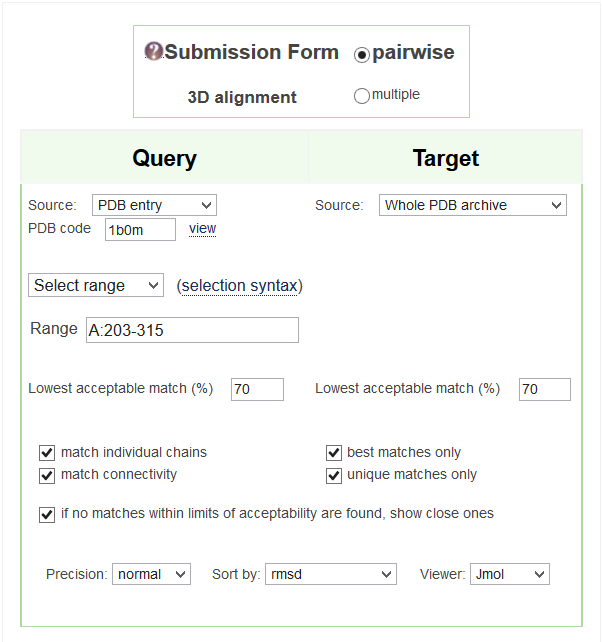
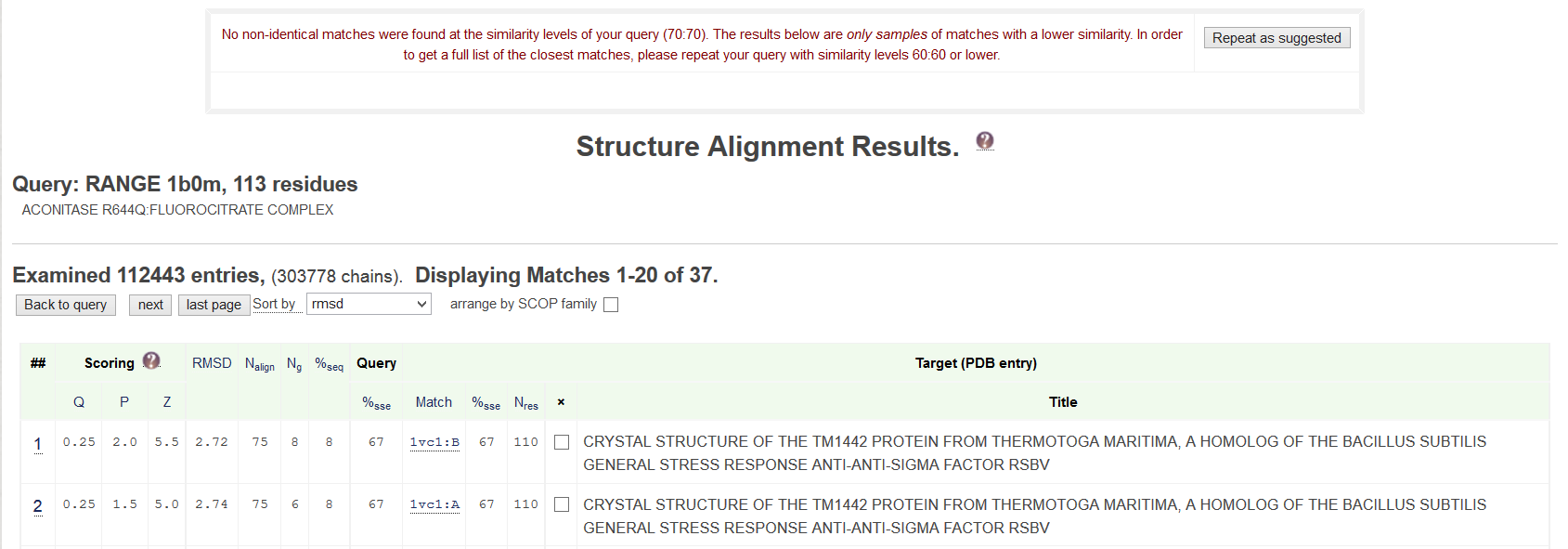
Для поиска был выбран домен 1b0m A:203-315.
При параметрах по умолчанию (Рис. 4а), исходной структуры 1b0m найдено не было (Рис. 4б).
 |
 |
| a | б |
Рисунок 4.Поиск домена с параметрами по умолчанию сервисом PDBeFoldа. Принтскрин запроса; б. Лучшие находки; сортировка по RMSD
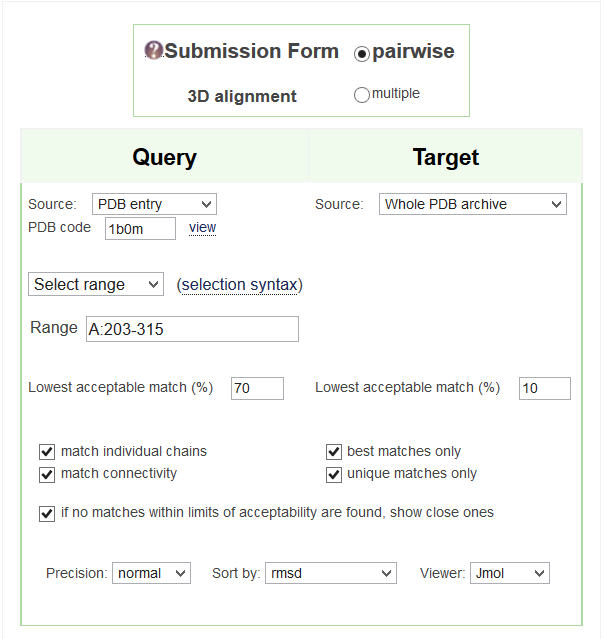
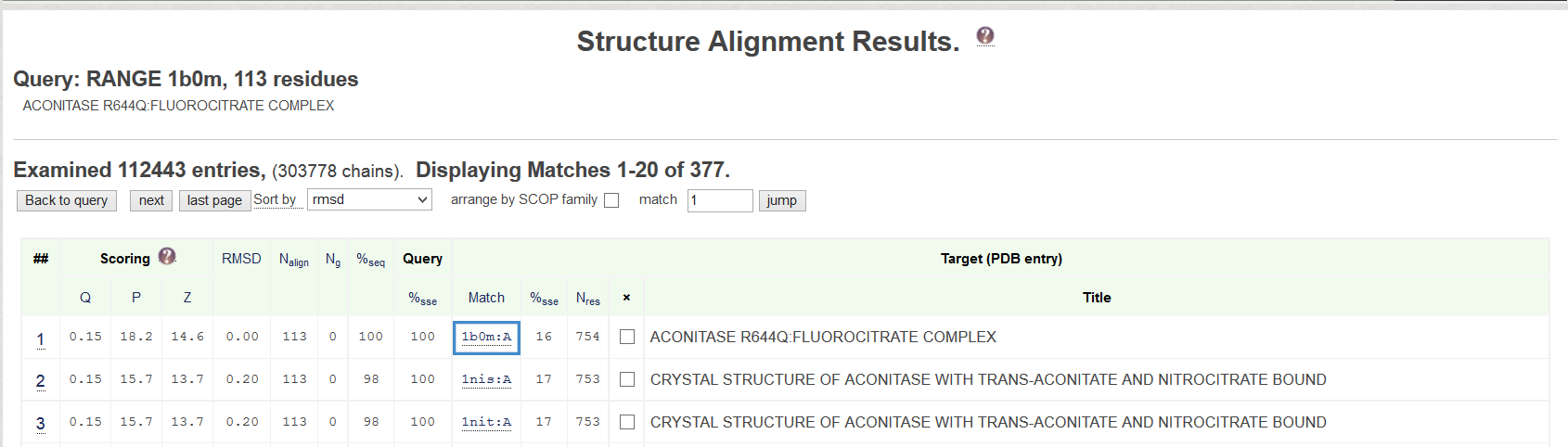
Было замечено, что искомый домен является лишь 14% частью всего белка (112/753 а.к.), что гораздо меньше чем 70% порог перекрывания, установленный по умолчанию. Поэтому, порог перекрывания мишени был снижен до 10% (Рис. 5а). Прием сработал, и исходная струткура 1b0m была обнаружена (Рис. 5б)
 |
 |
| a | б |
Рисунок 5.Поиск домена с порогом перекрывания мишени 10% сервисом PDBeFoldа. Принтскрин запроса; б. Лучшие находки; сортировка по RMSD; исходная структура выделена рамкой среди находок
Используя SCOP, для совмещения были выбраны следующие домены TCR человека (T-cell receptor) структуры 1kgc:
домен α-цепи TCR d:118-206 и домен β-цепи e:119-247.
Для каждой области был получен PDB файл, содержащий только интересующие нас атомы:
1kgc_alpha.pdb и 1kgc_beta.pdb.
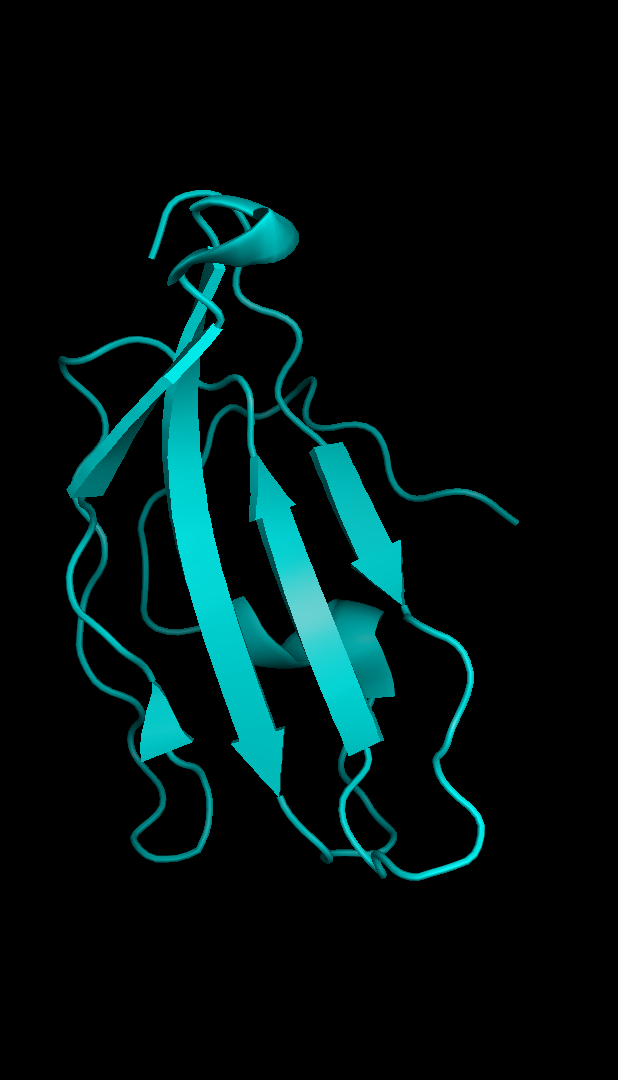
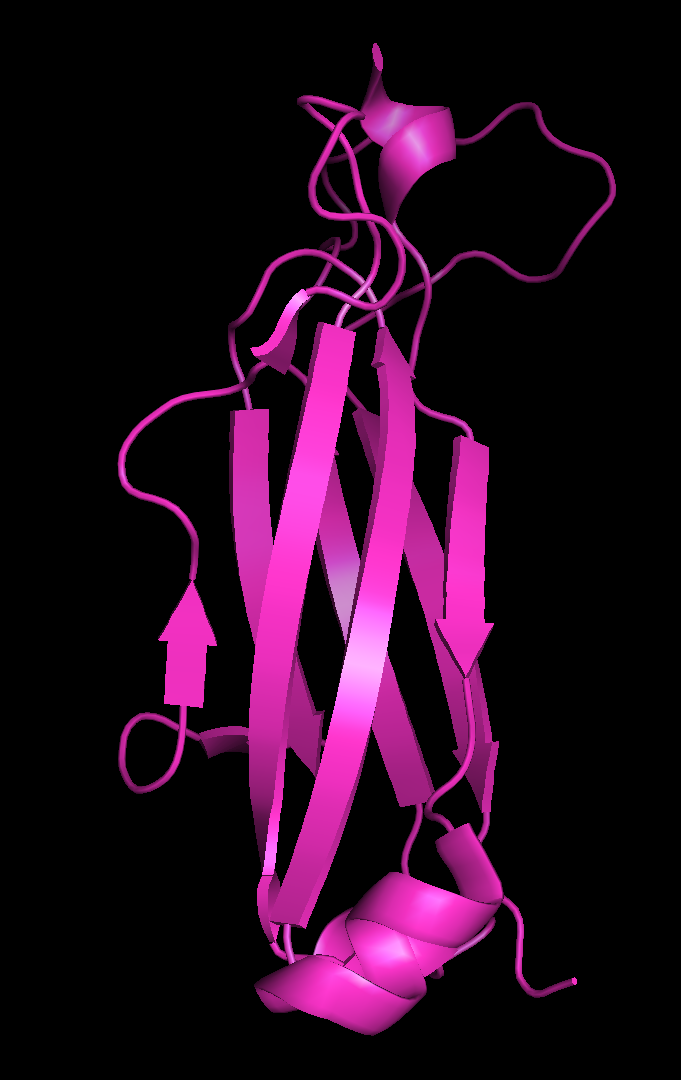
Изображения доменов представлены на Рис. 6.
 |
 |
| a | б |
Рисунок 6.Домены TCR человекаа. Домен α-цепи б. Домен β-цепи
Для совмещения доменов необходимо постоить карту их β-листов, что и было сделано при помощи сервиса SheeP . Карты были выровнены перестановкой строк и столбцов таким образом, чтобы направления тяжей у доменов совпадали (Рис. 7).
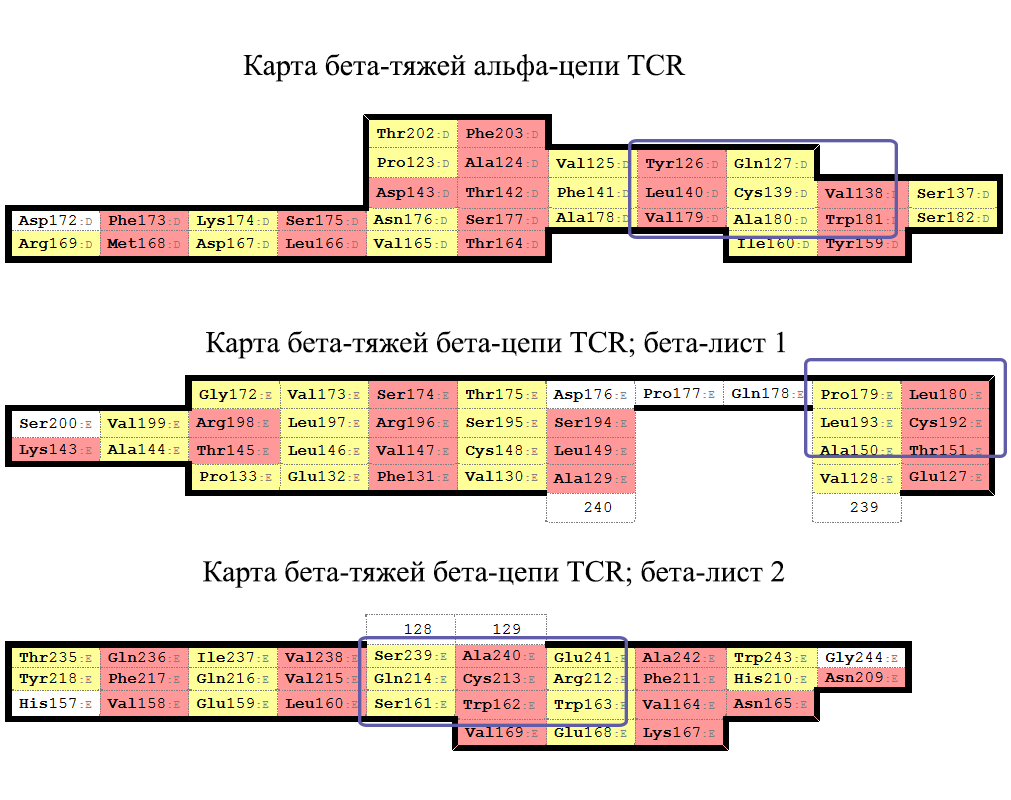
Рисунок 7. Карты β-листов доменов TCR. Синими рамками выделены области, содержащие консервативный цистеин.
Для совмещения двух β-листов необходимо определить соответствующие друг другу аминокислотные остатки. В данном
примере, мы считаем, что остатки цистеина являются высоко консервативными, и пытаемся совместить их и их окружение.
Осталось понять, какой из листов β-цепи соответствует листу цепи α. Из рисунка 7 видно, что цистеин первого листа цепи β
расположен терминально по отношению к своему β-тяжу и вряд ли хорошо совместится с соответствующим остатком из другой цепи.
С другой стороны, Cys второго листа имеет схожее окружение (топологически) с Cys из α-цепи, поэтому именно этот лист был выбран для совмещения.
С помощью команды pair_fit в PyMol были совмещены следующие пары остатков:
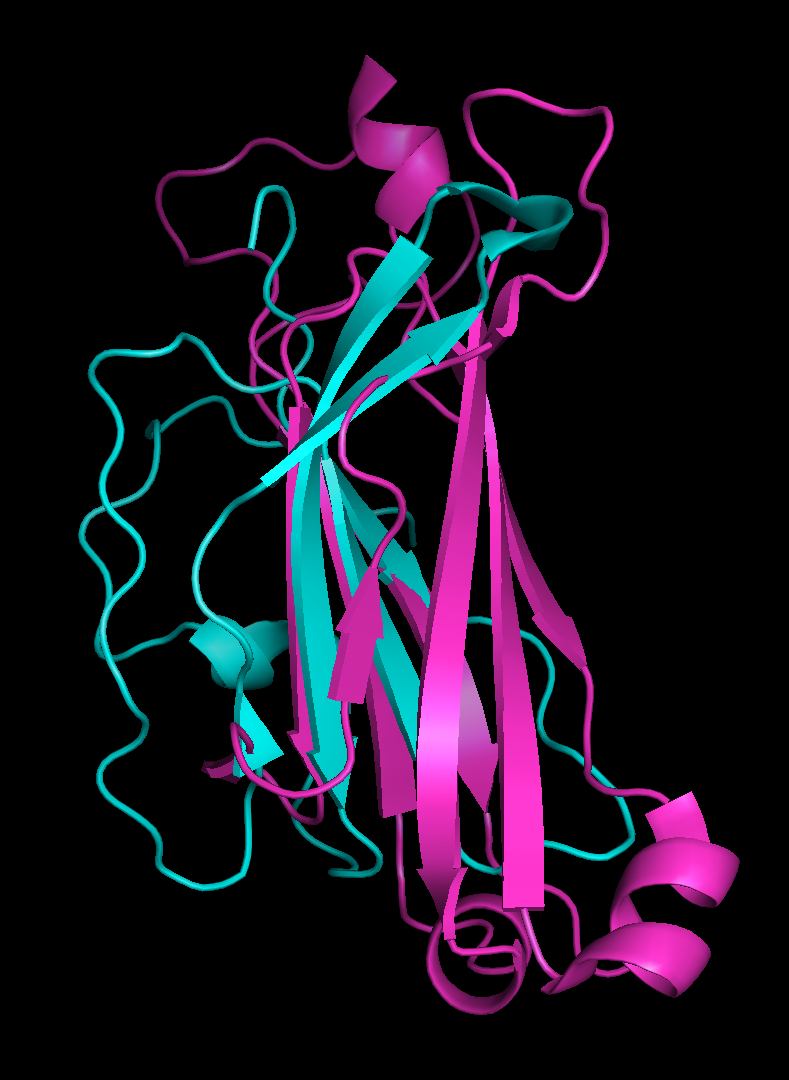
pair_fit alpha and resi 139 and (name CA), beta and resi 213 and (name CA), alpha and resi 140 and (name CA), beta and resi 214 and (name CA), alpha and resi 180 and (name CA), beta and resi 162 and (name CA), alpha and resi 138 and (name CA), beta and resi 212 and (name CA), alpha and resi 127 and (name CA), beta and resi 240 and (name CA),На Рисунке 8 представлены результаты совмещения, следует отметить высокий уровень сходства β-листов.
 |
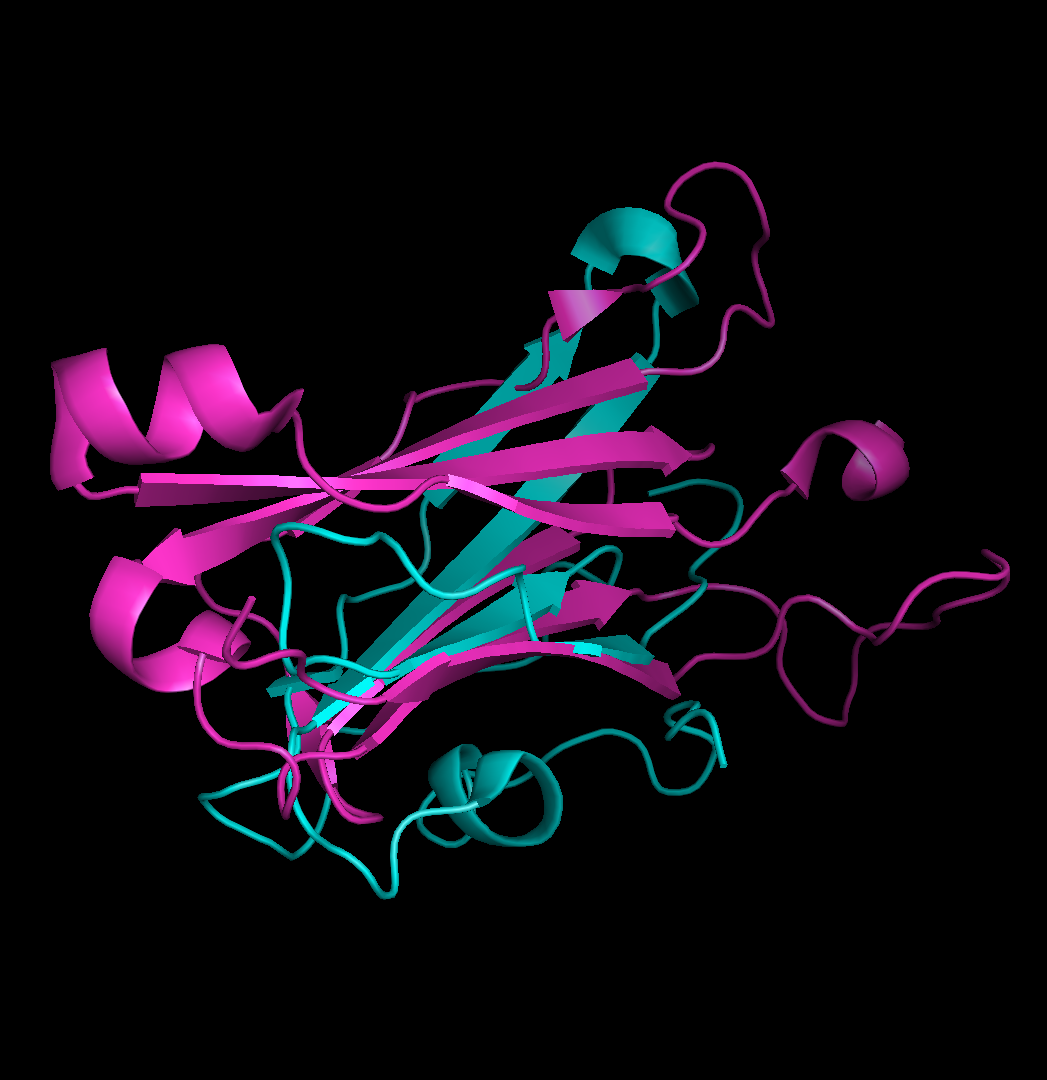 |
Рисунок 8. Пространственное совмещение двух β-листов. Розовым выделена β-цепь, цветом морской волны - α-цепь