Практикум 8
Задание 1
В качестве вариант алгоритма BLAST был взят blastn ввиду того, что исходно почти ничего не известно про эту последовательность, в том числе входит ли в неё белок-кодирующая часть. Выбранная база данных - Nucleotide collection (nr/nt) также из-за недостатка информации об этой последовательности.выдача BLAST. Одинаковый score и E-value наблюдается у первых двух находок из одного надсемейства. Исходная последовательность почти наверняка является гистоном H3, одним из белков нуклеосомы, что подтверждается наименьшим E-value находок с H3.Согласно первым 77 лучшим находкам, исходная хроматограмма вероятно принадлежит представителю надсемейства Gammaroidea
Задание 2
последовательность контига, в котором производился поискИспользовался blastX, поскольку нужно по нуклеотидной последовательности найти функцию соответствующего ей белка по схожим белкам. В качестве базы данных UniProtKB/Swiss-Prot(swissprot), чтобы у найденных белков точно была указана функция и чтобы выдача появилась за меньшее время. Был взят участок контига на месте предполагаемого гена. Также для большей скорости работы Бласта поиск происходил в пределах домена бактерий.
выдача BLASTСогласно функциям 27 из 28 полученных белков, исходный из контига синтезирует лизилфосфатидилглицерол. Это согласуется с тем, что рассматриваемый контиг был взят из Листерии, патогена, для которого важна устойчивость к антимикробным пептидам, которую может обеспечить лизилфосфатидилглицерин.
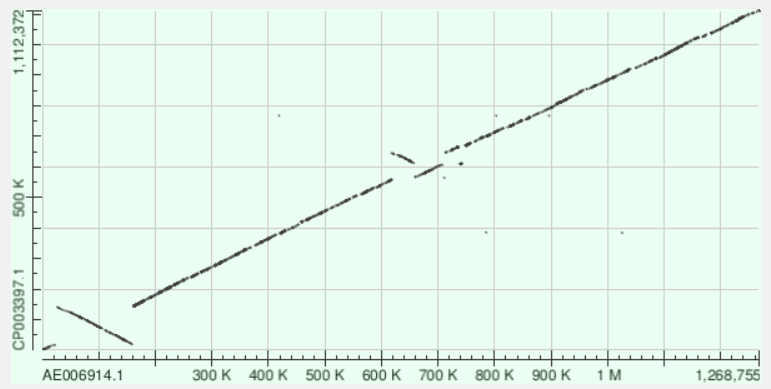
Задание 3
Построена карта локального сходства хромосом Rickettsia conoriiиRickettsia typhi. На фрагменте 20К-160К можно видеть инверсию. Вероятно, на фрагменте 610К-710К произошла транслокация, после инверсия части фрагмента.