Практикум 12
Задание 1. Знакомство с базой данных OPM
| Параметр | Данные |
|---|---|
| Название белка | Amyloid secretion protein FapF |
| Перевод на русский | Белок секреции амилоидов |
| Идентификатор PDB | 5о65 |
| Идентификатор Uniprot | C4IN73_9PSED |
| Толщина гидрофобной части белка в мембране | 25.0 Å |
| В какой мембране находится белок | наружная мембрана грам-отрицательной бактерии |
| Среднее количество остатков в одном β-тяже белка | 9 |
Координаты трансмембранных участков 1( 108- 118), 2( 149- 159), 3( 166- 174), 4( 201- 214), 5( 223- 232), 6( 265- 274), 7( 282- 290), 8( 316- 324), 9( 333- 341),10( 363- 373),11( 381- 388),12( 396- 404) в каждой из 3х субъединиц

Задание 2. DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
В качестве альфа-спирального белка был взят белок лизиса YQFA_ECOLI, бета-листового - белок секреции амилоидов C4IN73_9PSED. В качестве входных данных использовались fasta-файлы: YQFA_ECOLI, C4IN73_9PSED. Предсказания трансмембранных участков представлены на рисунках ниже. Текстовую выдачу программы можно посмотреть по ссылкам:YQFA_ECOLI,C4IN73_9PSED.
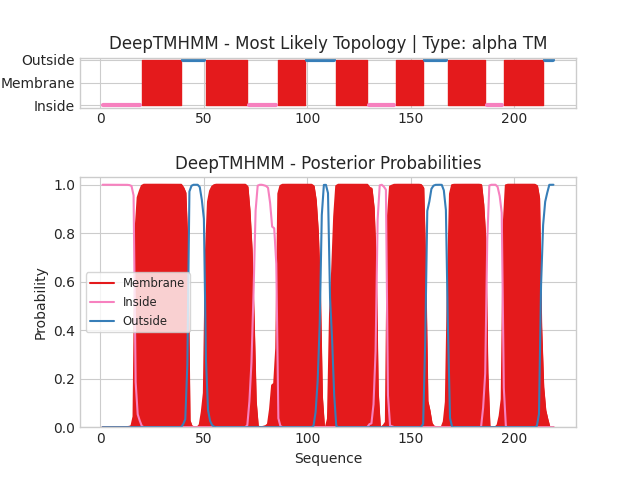
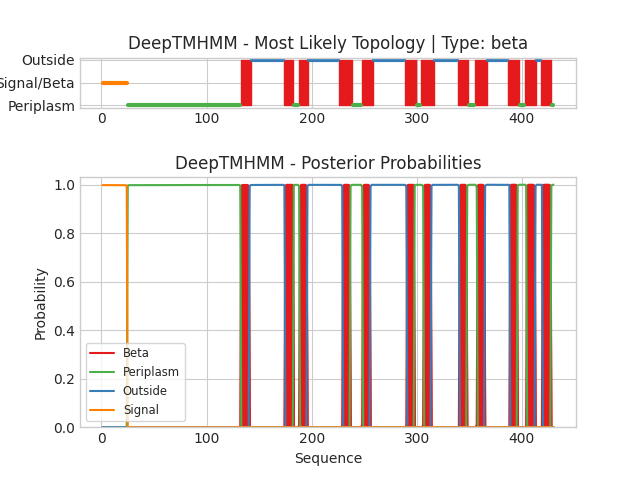
На схеме для YQFA_ECOLI выделены участки структуры, расположенные со стороны цитоплазмы (Inside), с внешней стороны клетки (Outside) и внутри мембраны (Membrane). В нижней части схемы для каждого аминокислотного остатка показана вероятность принадлежности его определенной части структуры. Можно отметить, что N-конец находится в цитоплазме, а С-конец вне клетки. На схеме для C4IN73_9PSED отмечены участки, расположенные с внешней стороны клетки, обращенные в межмембранное пространство (Periplasm), пронизывающие мембрану, а также сигнальная структура (Signal) в виде спирали. N- и С-концы обращены в межмембранное пространство.
Задание 3. PPM: Предсказание положения выданного белка в мембране
Программа PPM получает на вход файл PDB и параметры нашего белка. Он находится в мембране грам-положительной бактерии (информацию о таксономии организма можно найти в Uniprot), N-конец обращен внутрь от мембраны согласно схеме из предыдущего задания, белок пересекает одну мембрану. Небелковые молекулы исключать не нужно, так как на вход подается только белок, предсказанный программой Alfafold.
| Параметр | Данные |
|---|---|
| Название белка | UPF0073 inner membrane protein YqfA |
| Перевод на русский | Внутримембранный белок YqfA |
| PDB-структура | файл |
| Идентификатор Uniprot | YQFA_ECOLI |
| Толщина гидрофобной части белка в мембране | 26.7 ± 1.3 Å |
| В какой мембране находится белок | наружная мембрана грам-отрицательной бактерии |
| Среднее количество остатков в одном β-тяже белка | 15 |
Координаты трансмембранных участков 1( 18- 39), 2( 51- 70), 3( 84- 102), 4( 114- 134), 5( 138- 153), 6( 170- 187), 7( 193- 210)

Задание 4. Сравнение алгоритмов предсказания трансмембранных спиралей
Координаты трансмембранных участков YQFA_ECOLI из предсказания сервера PPM: 1( 18- 39), 2( 51- 70), 3( 84- 102), 4( 114- 134), 5( 138- 153), 6( 170- 187), 7( 193- 210)
Координаты трансмембранных участков YQFA_ECOLI из DeepTMHMM: 1( 17- 42), 2( 51- 74), 3( 86- 106), 4( 111- 133), 5( 139- 157), 6( 168- 186), 7( 195- 212)
Координаты трансмембранных участков C4IN73_9PSED из OPM: 1( 108- 118), 2( 149- 159), 3( 166- 174), 4( 201- 214), 5( 223- 232), 6( 265- 274), 7( 282- 290), 8( 316- 324), 9( 333- 341),10( 363- 373),11( 381- 388),12( 396- 404)
Координаты трансмембранных участков C4IN73_9PSED из DeepTMHMM:1( 133- 140), 2( 175- 182), 3( 189- 195), 4( 230- 236), 5( 249- 255), 6( 291- 297), 7( 307- 313), 8( 341- 347), 9( 358- 364),10( 389- 395),11( 405- 412),12( 420- 427)
Согласно сопоставлению предсказания сервера PPM и DeepTMHMM, хотя есть расхождения на 1-4 остатка, все спирали найдены примерно в одних и тех же частях последовательности. А согласно сопоставлению предсказания из OPM и DeepTMHMM, не смотря на то, что количество предсказанных бета-листов совпало, они находятся совсем в разных местах. Таким образом, DeepTMHMM во втором случае хуже предсказал положение частей белка.
Задание 5. База данных TCDB
Поиск в базе данных TCDB осуществлялся по идентификаторам Uniprot, оба белка,YQFA_ECOLI(P67153) и C4IN73_9PSED(C4IN73), были найдены. В ней можно получить информацию о длине последовательности, молярной массе, принадлежности организму, топологии, субстрате и условиях взаимодействия с ним, количестве трансмембранных элементов. Также тут есть ссылки на другие базы данных, приведены сами последовательности в формате fasta. Переходя по ссылке Predict TMSs, можно получить список координат трансмембранных участков и визуализацию их в последовательности.
Белок YQFA_ECOLI имеет ТС-код 1.C.113.1.10, чтоо отражает его включение в определенные категории трансмембранных белков: 1 - Канал; C - формирующие пору токсины; 113 - семейство гемолизина 3.
Белок C4IN73_9PSED имеет ТС-код 9.B.153.3.18: 9 - Не полностью охарактеризованные транспортные системы; В - предполагаемый белок-транспортер; 153 - семейство предполагаемых бета-бочоночных поринов/Альфа-амилазы.