Практикум 4
| Название | Мнемоника |
|---|---|
| Streptomyces avermitilis | STRAW |
| Thermobifida fusca | THEFY |
| Acidothermus cellulolyticus | ACIC1 |
| Nocardioides sp. | NOCSJ |
| Arthrobacter sp. | ARTS2 |
| Leifsonia xyli | LEIXX |
| Clavibacter michiganensis | CLAMS |
| Bifidobacterium longum | BIFLO |
Задание 1
Найдём среди своих бактерий возможные гомологи белка CLPX_ECOLI с помощью следующего набора команд:
cat ARTS2.fasta BIFLO.fasta CLAMS.fasta ACIC1.fasta LEIXX.fasta NOCSJ.fasta STRAW.fasta
THEFY.fasta > /home/students/y20/suzdal20/public_html/term4/pr4/proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out db
blastp -query P0A6H1.fasta -db db -evalue 0.001 -out clpx_all.txt
Задание 2
Список находок Результат работы алгоритма muscle Дерево, полученное программой MEGA методом Neighbor-joining
На дереве видны такие паралоги: Q82QV8_STRAW и Q82EB8_STRAW, CLPX_CLAMS и B0RHW4_CLAMS, A0K236_ARTS2 и A0JR82_ARTS2. Ортологи: CLPX_LEIXX и CLPX_CLAMS, CLPX_CLAMS и CLPX_ARTS2, RUVB_BIFLO и RUVB_ARTS2. Раскрасим группы ортологов на полученном дереве.
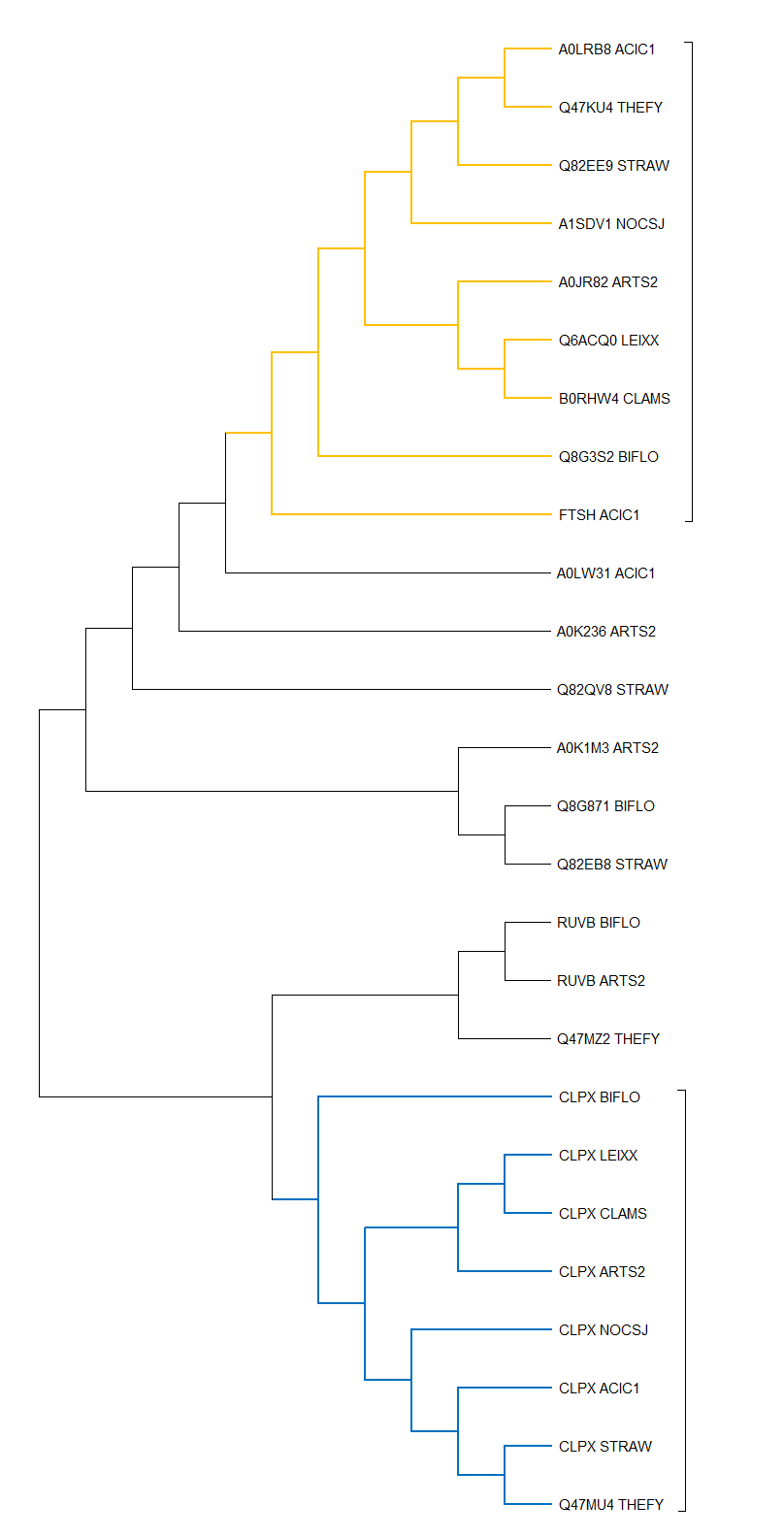
Схлопнем ортологичные группы

Ортологичная группа, окрашенная рыжим, представлена АТФ-зависимой цинковой металлопротеазой FtsH, найденной у всех бактерий. (Заметим, что A0LRB8_ACIC1 и FTSH_ACIC1 хоть и не ортологичны друг другу, поскольку из одного организма, но попарно ортологичны со всеми остальными белками в группе). Данная группа соответствует исходной филогении бактерий по субдеревьям (ACIC1,THEFY) и (ARTS2,(LEIXX,CLAMS)).
Ортологичная синяя группа представлена АТФ-связывающей субъединицей АТФ-зависимой протеазы Clp, найденной у всех бактерий, выбранных для работы. Данная клада значительно отличается от исходной филогении. Совпадает только ветвь (ARTS2,(LEIXX,CLAMS)).