Проект "Эволюция белков митохондриальных рибосом".
I. Поиск бактериальных гомологов белка S10 по нормированному профилю белков эукариотических митохондриальных рибосом
1. Общая характеристика обучающей выборки.
С помощью SRS
из UniProt получили 11 последовательностей рибосомального белка S10 из митохондрий разных
эукариот. Для этого осуществили запрос: [uniprot-AllText:S10*] & [uniprot-Description:28S*].
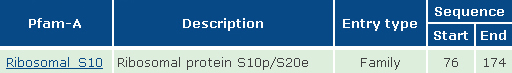
Определили доменную структуру найденных белков на примере последовательности RT10_HUMAN из митохондрий человека:

Таблица. Описание доменной структуры.
Построили выравнивание полных последовательностей найденных белков с помощью программы muscle на kodomo-count и
сравнили с выравниванием из PFAM в GeneDoc:

Исходя из полученных данных (файл Genedoc) и с учетом того, что при извлечении выравнивания
необходимых доменов не были обнаружены 4ре из них, можно сделать вывод, что выравнивание muscle вполне удачно и не требует последующей работы с
программой mafft.
2. Краткое описание и сравнение простого и нормированного профилей.
При помощи команды pfw -m out.ali > out.weighted.ali в выравнивание полных последовательностей добавили веса.
Построили профиль (pfmake -m out.weighted.ali /usr/share/pftools23/blosum45.cmp > myprofile.prf ) с последующей его нормировкой относительно случайной базы
(autoscale -m myprofile.prf > myprofile.scaled.prf).
Оказалось, простой и нормированный профили отличаются значениями SCORE в поле MA (CUT_OFF) и R1/R2 в поле MA (NORMALIZATION), где R1 и R2 - коэффиценты функции нормирования.
3. Обоснование выбора порога.
С помощью программы pfsearch пакета PFTOOLS провели поиск гомологичных белков в альфа-
и гаммапротеобактерий с использованием нормированного варианта профиля.
Таблица. Результаты поиска по двум группам бактерий с разными значениями порога.
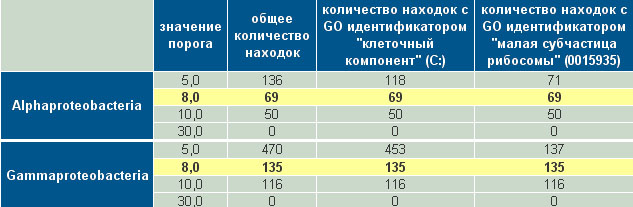
Исходя из данных таблицы, видно, что пороговое значение 8,0 обеспечивает нахождение гомологичных последовательностей, все из которых имеют пометку GO в описании, а значит,
может быть оптимально для поиска по профилю.
4. Распределение нормированных весов находок в протеомах 2-х групп бактерий.
Результат поиска по нормированному профилю с порогом 8,0 в протеоме альфапротеобактерий - alpha8.search, гаммапротебактерий - gamma8.search.
Гистограмма. Распределение весов находок в альфа- и гаммапротеобактериях (Excel-файл).
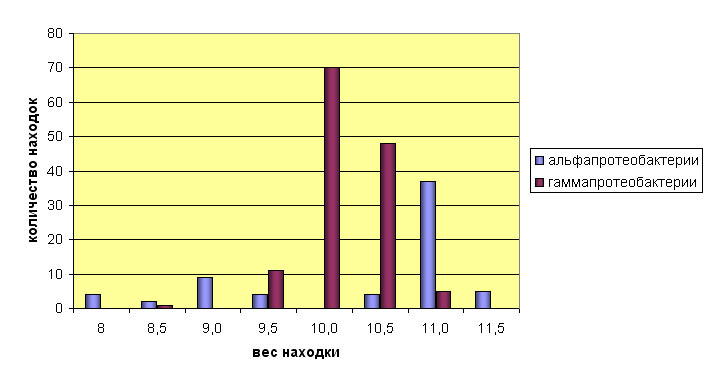
Находок среди гаммапротеобактерий вдвое больше, чем среди гаммапротеобактерий. По этой причине имеет смысл не учитывать абсолютное количество найденных
гомологов, а рассмотреть формы полученных графиков распределения.
Из гистограммы видно, распределение для альфапротеобактерий ближе к нормальному, кроме того, его медиана визуально больше, чем у распределения для гаммапротеобактерий.
Это позволяет сделать промежуточный вывод о том, что последовательности из протеома альфапротеобактерий несколько ближе к исследуемому профилю.
5. Тест Вилкоксона.
С помощью пакета STADIA провели тест Вилкоксона на сравнение распределений весов находок в двух группах бактерий.
Результаты теста говорят о различии между медианами выборок. Для получения дополнительной информации воспользовались функцией "Описательная статистика".
Получили, что медиана альфапротеобактерий правее гаммапротеобактерий. Это свидетельствует о том, что альфапротеобактерии ближе к рибосомальным белкам из митохондрий эукариот.
II. Филогенетический анализ рибосомальных белков S10 из эукариотических митохондрий и 3-х групп бактерий
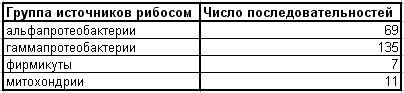
Создали внешнюю группу из 7-ти последовательностей рибосомальных белков из Firmicutes, имеющих то же название, что и заданнй белок.
Также получили файлы с последовательностями из альфа- и гаммапротеобактерий, отобранными ранее с выбранным порогом.
Таблица. Описание выборки.
После посторили объединенное выравнивание митохондриальных белков и белков из рибосом альфапротеобактерий, гаммапротеобактерий и фирмикут.
Методом максимального правдоподобия (phylip proml) было построено филогенетическое дерево:
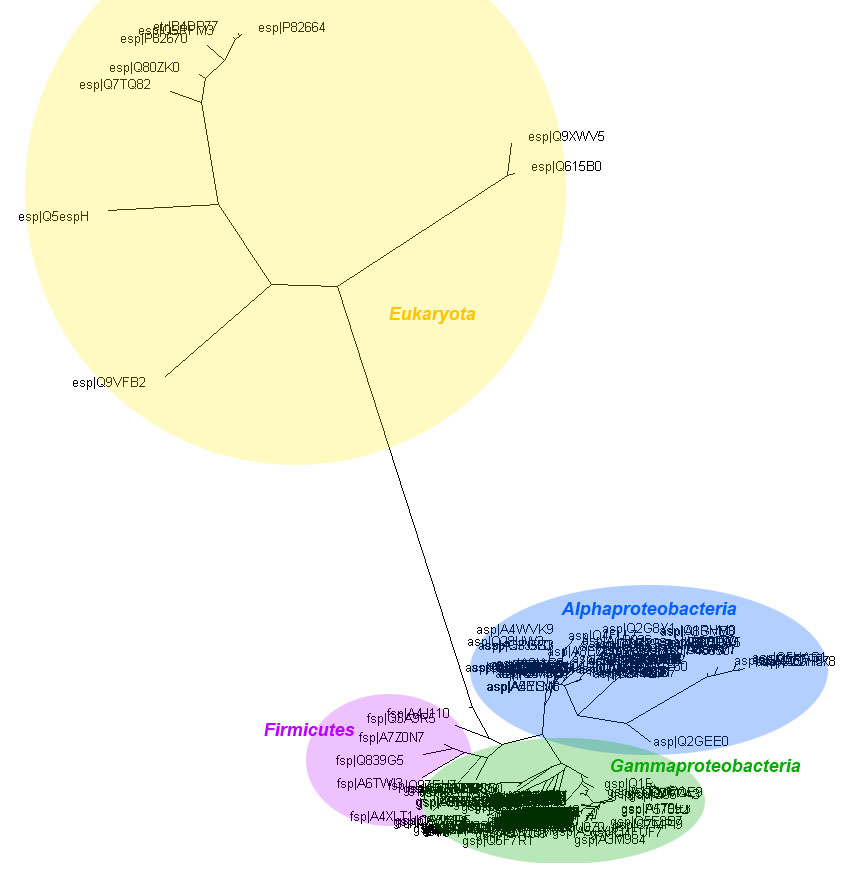
Увеличенный фрагмент для большей ясности:
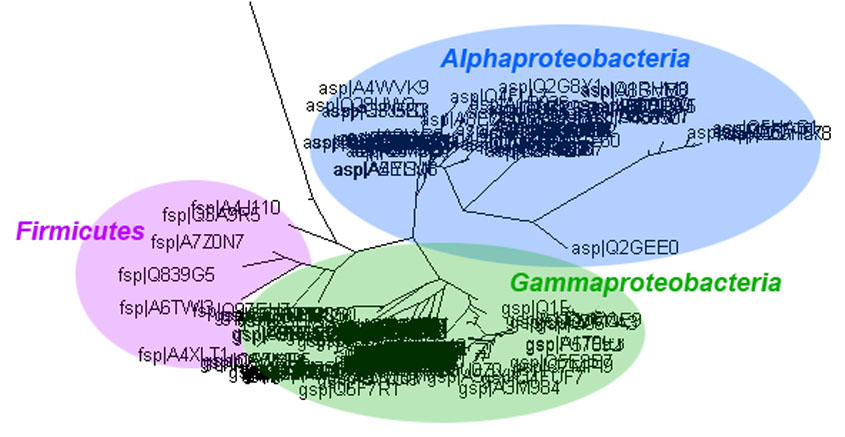
По данному филогенетическому дереву сложно сделать вывод о происхождении рибосомальных белков эукариот.
С помощью команды phylip protdist определелили попарные эволюционные расстояния по JTT и
построили гистограмму распределения попарных расстояний между митохондриальными белками и белками из альфа- и гаммапротеобактерий (Excel-файл):
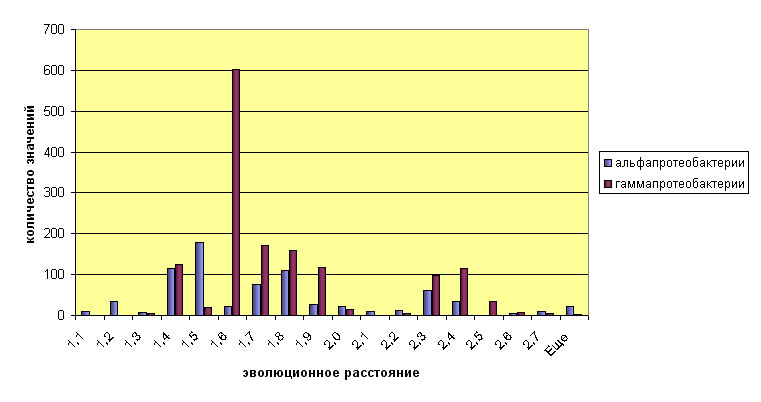
Из гистограммы видно, что медиана распределения в случае альфапротеобактерий меньше, чем гаммапротеобактерий. Это свидетельствует о том, что
попарное эволюционное расстояние наименьшее в случае митохондрий и альфапротеобактерий.
III. Резюме
В результате исследования распределения весов находок в протеоме 2-х групп бактерий и определения попарных эволюционных расстояний с учетом того, что реконструкция филогенетического дерева не дала результатов, можно сделать вывод о том, что рибосомальные белки из альфапротеобактерий ближе к белкам эукариот
.








