Пространственное строение белка дегидратазы (dehydratase) из генома бактерии Chloroflexus aurantiacus, штамм J-10-fl
J-mol — программа, дающая возможность просмотра структуры молекул в трёх измерениях. Она написана на языке Java и поддерживает многие форматы файлов: Protein Data Bank (pdb), MDL Molfile (mol), Crystalographic Informatic File (cif) и так далее.
Protein Data Bank — это банк данных 3-D структур нуклеиновых кислот и белков. Файлы в pdb-формате содержат в себе информацию о белке (состав молекулы, трёхмерная структура), полученную методами рентгеновской кристаллографии или ЯМР-спектроскопии.
Для каждого белка и нуклеиновой кислоты из PDB существует свой идентификатор. Для белка дегидратазы бактерии C. aurantiacus, штамм J-10-fl, ID — 4E3E.
Трёхмерная структура белка представлена на Рисунке 1. PDB-файл с информацией о белке можно скачать здесь.
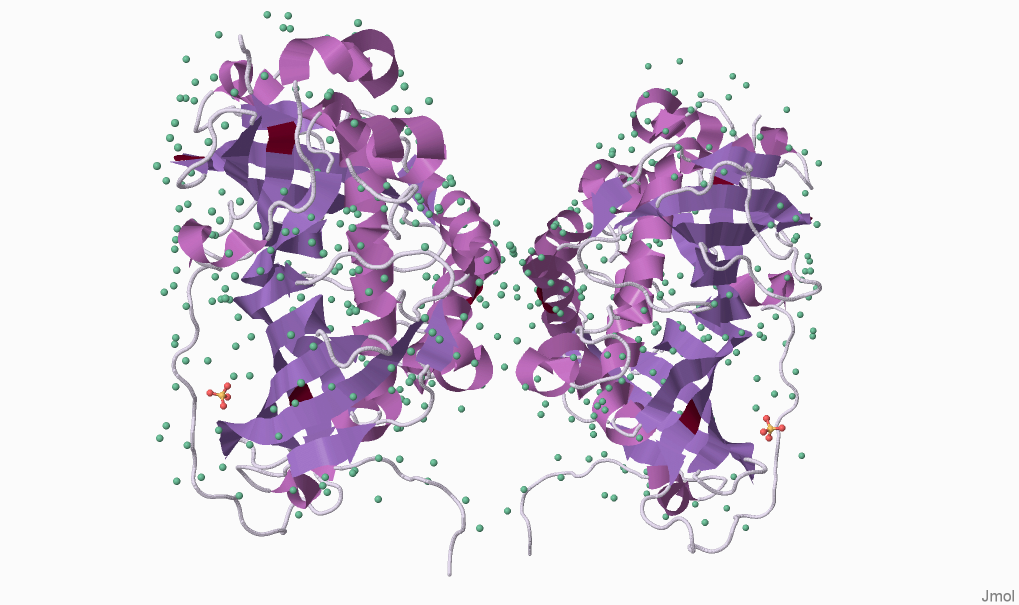
Рисунок 1. Пространственное строение дегидратазы C. aurantiacus, штамм J-10-fl (идентификатор записи PBD 4E3E).
Рисунок получен с помощью программы J-mol.
На рисунке зеленоватым цветом обозначены молекулы воды, сиреневым — α-спирали, фиолетовым — β-листы; участки, не обладающие вторичной структурой — сероватым. Бордовые участки β-листов — селенметионин, жёлтый и красный — атомы серы и кислорода соответственно, входящие в состав сульфат-иона
Для того, чтобы данный белок выглядел именно так, как на приведённом выше изображении, был написан J-mol скрипт. Скачать скрипт можно здесь. Его текст размещён ниже:
load 4E3E.pdb backbone off wireframe off ribbons off cpk off # selects main chain select *.CA cartoons #coloring color [158,113,197] select 118-122 color [230,220,240] select 193-195 color [230,220,240] select 351-353 color [230,220,240] select 320-338 color [230,220,240] select 280-288 color [230,220,240] select 214 color [230,220,240] select 208-211 color [230,220,240] select 201-204 color [230,220,240] select 241-249 color [230,220,240] select 3-10 color [230,220,240] select 151-170 color [230,220,240] select 16-19 color [230,220,240] select 268-270 color [230,220,240] select 176-181 color [230,220,240] select 302-307 color [230,220,240] select 43-44 color [230,220,240] select 134-138 color [230,220,240] select 97-104 color [230,220,240] select 58-64 color [230,220,240] select 29 color [230,220,240] select 23-26 color [230,220,240] select 83-84 color [230,220,240] select 227-229 color [230,220,240] #HOH coloring select hoh cpk 110 color [113,196,157] #SO4 coloring select SO4 backbone 100 wireframe 50 cpk 110 select 500.s color [255,201,102] select 500.o4 color [255,102,102] select 500.o1 color [255,102,102] select 500.o2 color [255,102,102] select 500.o3 color [255,102,102] #two parts select helix color [196,113,194] #mse select mse color [128,0,43] #bgrnd background [249,249,249] center rotate Z 26
Для того, чтобы посмотреть скрипт в действии, необходимо скачать сам скрипу и pdb-файл, а также иметь установленной программу J-mol.
Кроме координат молекул, как было написано ранее, в pdb-файле содержится информация об источнике белка, метода, его структура и использованный метод анализа. Информация, выделенная из данного файла представлена ниже в Таблице 1 и 2.
| Графа | Значение |
| TITLE | crystal structure of putative maoc domain protein dehydratase from chloroflexus aurantiacus j-10-fl |
| COMPD | mol_id: 1
molecule: maoc domain protein dehydratase chain: a, b engineered: yes |
| SOURCE | mol_id: 1
organism_scientific: chloroflexus aurantiacus organism_taxid: 324602 strain: j-10-fl gene: caur_0173 expression_system: escherichia coli expression_system_taxid: 469008 expression_system_strain: bl21(de3)codon+ril expression_system_vector_type: plasmid expression_system_plasmid: bc-psgx3(bc) |
| EXPDTA | x-ray diffraction |
| HETNAM | mse selenomethionine
SO4 sulfate ion |
| FORMUL | mse 8(C5H11NO2Se)
SO4 2(O4 S2-) HOH *480(H2O) |
| Графа | Значение |
| Название | Кристаллическая структура предполагаемого MaoC домена белка дегидратазы |
| Состав |
Молекула: MaoC домен белка дегидратазы
Цепи: a, b |
| Источник | Chloroflexus aurantiacus |
| Метод расшифровки | рентгеноструктурный анализ |
| Малые молекулы и ионы | Mse селенометионин
SO4 сульфат-ион |
| Формулы малых молекул и ионов | Mse 8(C5H11NO2Se)
SO4 2(O4 S2-) HOH *480(H2O) |
Из Таблицы 2 очевидно, что данная пространственная структура белка, который состоит из двух цепей, была получена из бактерии C. aurantiacus методом рентгеноструктурного анализа.
Сравнение строения белка с его биологической единицей
Биологическая единица — это макромалекулярная структура, которая считается функциональной формой молекулы.
У данного белка две биологических единицы. я проанализировала каждый в программе J-mol, и оказалось, что каждый из них представляет собой одну из двух цепочек исходного белка. Строение абсолютно аналогичное.
Ниже на Рисунке 2 приведено изображение этих биологических единиц. Сравнив Рисунок 1 и 2, приходим к выводу об их сходстве.
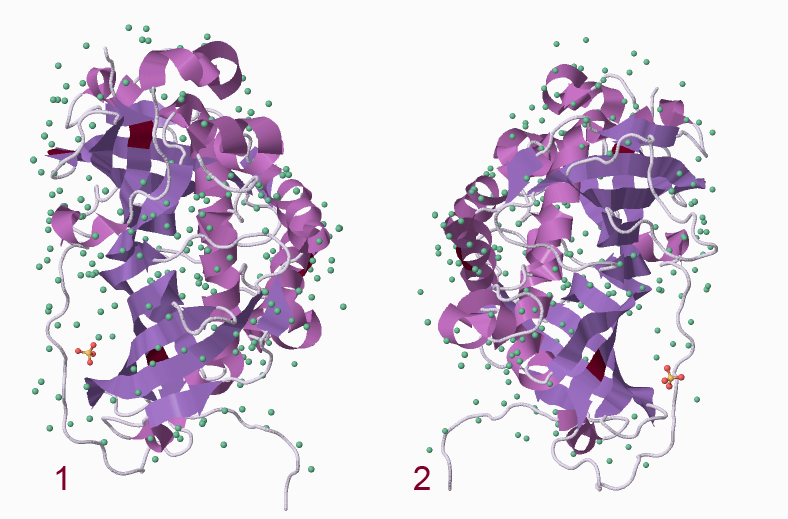
Рисунок 2. Биологические единицы белка дегидратазы.