BLAST
BLAST — это программа, широко используемая в биоинформатике для поиска гомологов для поданной последовательности белка в различных базах данных.
На вход программе подаётся последовательность в формате fasta, выбирается база данных, по которой идёт поиск, а также алгоритм поиска. В данном случае был выбран алгоритм BlastP, который просто сравнивает последовательность белка с другими из базы данных[1]. На выходе получается таблица со списком подходящих последовательностей и выравнивание каждой из них с исходной.
Таблица состоит из 7 колонок: Description, Max score, Total score, Query cover, E-value, Ident, Accession. Ниже приведено краткое пояснение каждого из этих названий колонок.
- Description — полное название белка; в квадратных скобках содержит наименование организма, из которого белок был получен.
- Max score — максимальный вес выравнивания участка найденной последовательности и исходной.
- Total score — общий вес выравнивания. Max score и Total score совпадают, если был всего один участок сходства (matches), по которому производилось выравнивание.
- E-value — ожидаемое число последовательностей с таким же или большим весом при поиске в базе данных случайных последовательностей. Оно показывает, насколько полученное выравнивание случайно.
- Query cover — процент, который составляет гомологичный участок в найденной последовательности от её общей длины.
- Indent — процент совпавших аминокислот.
- Accession — идентификатор белковой последовательности в выбранной для поиска базе данных.
Поиск гомологов белка дегидратазы из организма Chloroflexus aurantiacus штамм J-10-fl (идентификатор YP_001633816.1 в базе RefSeq) в банке Uniprot/SwissProt
В данном задании хорошей находкой считалась находка со следующими параметрами: Identity 40-70%, Сoverage < 100%, E-value < 0.00001, но и не 0.0.
Изначально программа была запущена для поиска гомологов белка дегидратазы, основываясь на банк последовательностей Uniprot/SwissProt. Но в результате было получено только 9 последовательностей, минимальным E-value которых составлял 0.17, что не соответствует упомянутым выше условиям. Результаты поиска приведены на Рисунке 1.
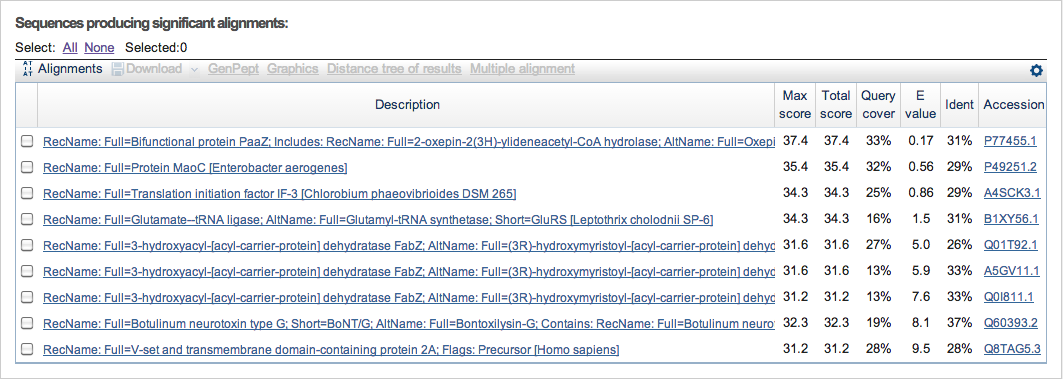
Рисунок 1. Результат поиска гомологов в банке последовательностей Uniprot/SwissProt.
Результат получен с помощью программы BLAST.
Следующие данные приведены для последовательностей, найденных в базе данных Refseq. На Рисунке 2 показана часть результата данного запроса.
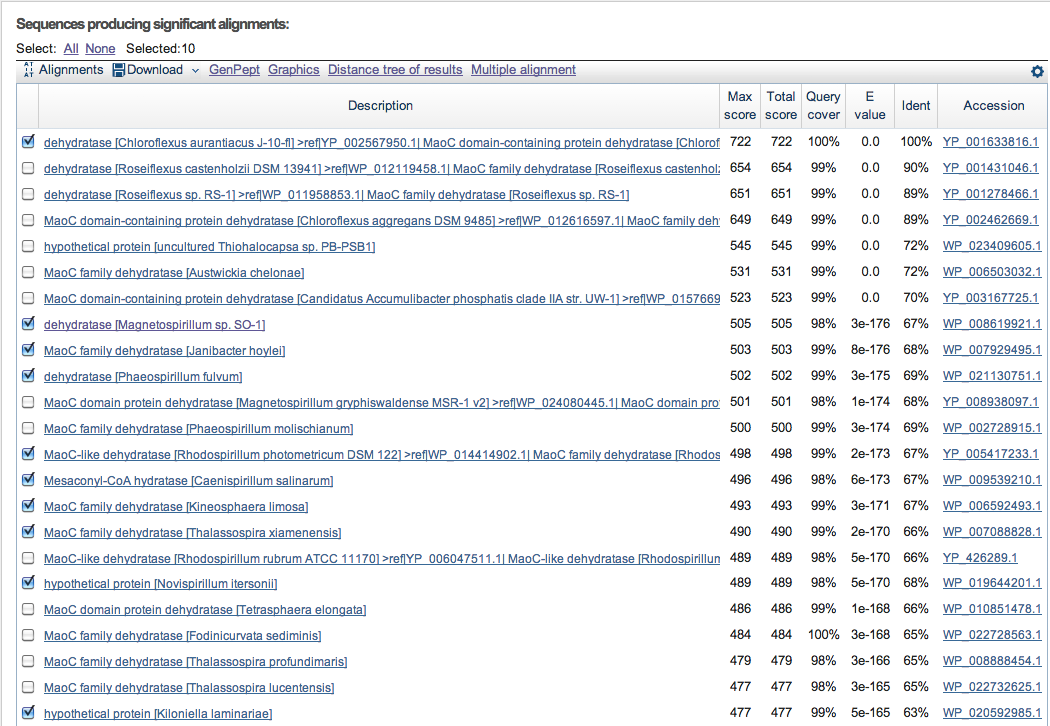
Рисунок 2. Первые 23 (из 100) результата поиска гомологов в банке последовательностей Refseq.
Галочкой отмечены последовательности белков, которые позднее были использованы в выравнивании.
Результат получен с помощью программы BLAST.
В качестве лучшей находки был выбран белок дегидратаза (ID WP_008619921.1 в базе Refseq) из организма бактерии Magnetospirillum sp. штамм SO-1. Это магнитотактическая бактерия-спирилла, обитающая в пресных водоёмах[2]
В Таблице 1 представлены данные для лучшей находки, полученные из выходной таблицы.
| Description | Max score | Total score | Query score | E-value | Indent | Accession | Positives | Gaps |
| dehydratase [Magnetospirillum sp. SO-1] |
505 | 505 | 98% | 3*10-176 | 67% | WP_008619921.1 | 81% | 1% |
Ниже приведено выравнивание последовательности-находки с исходной:
Query 4 KTNPGNFFEDFRLGQTIVHATPRTITEGDVALYTSLYGSRFALTSSTPFAQSLGLER--- 60
KTN GNFFEDFRLGQ I HATPRT+T GDVALYT+LYGSRFA+ SS FA+++GL
Sbjct 3 KTNSGNFFEDFRLGQVISHATPRTVTAGDVALYTALYGSRFAVNSSAEFAKAIGLAAENM 62
Query 61 ---APIDSLLVFHIVFGKTVPDISLNAIANLGYAGGRFGAVVYPGDTLSTTSKVIGLRQN 117
AP+D LL FH+VFGKTVPDISLNA+ANLGYA GRFG +VYPGDT++TTS VIGL++N
Sbjct 63 GHIAPVDDLLAFHVVFGKTVPDISLNAVANLGYAMGRFGEMVYPGDTITTTSTVIGLKEN 122
Query 118 KDGKTGVVYVHSVGVNQWDEVVLEYIRWVMVRKRDPNAPAPETVVPDLPDSVPVTDLTVP 177
+ +TGVVYV SVG NQ E+V++Y+RWVMVRKRD NA E VP+LP +V DL +P
Sbjct 123 SNKQTGVVYVRSVGTNQLHEMVVDYVRWVMVRKRDQNAATSEEKVPELPGAVAAADLVIP 182
Query 178 YTVSAANYNLAHAGSNYLWDDYEVGEKIDHVDGVTIEEAEHMQATRLYQNTARVHFNLHV 237
+ +Y+ AGS +LWDDY VGEKIDHVDG+TIEEAEHM ATRL+QNTA+VHFN +
Sbjct 183 DNLRLGDYDTTLAGSPHLWDDYAVGEKIDHVDGMTIEEAEHMMATRLWQNTAKVHFNQYT 242
Query 238 EREGRFGRRIVYGGHIISLARSLSFNGLANALSIAAINSGRHTNPSFAGDTIYAWSEILA 297
E +GRFGRR++YGGHIIS+AR+LSFNGL NA + AIN GRHTNP+FAGDTI+AW+E+L
Sbjct 243 EGQGRFGRRLIYGGHIISIARALSFNGLGNAFKLVAINGGRHTNPTFAGDTIHAWTEVLE 302
Query 298 KMAIPGRTDIGALRVRTVATKDRPCHDFPYRDAEG-NYDPAVVLDFDYTVLMPRR 351
K+ +PGR D+GALR+R VATK++PC FP + G ++DPAVVLDFDYTVLMPR+
Sbjct 303 KIEVPGRKDVGALRLRLVATKNQPCAGFPLKAENGKDFDPAVVLDFDYTVLMPRK 357
Query — это поданная последовательность, subject — последовательность находки. Исходя из выравнивания, представленного выше, участок сходства в поданной последовательности — 4-351, найденной — 3-357. Участок находки, похожий на query, состоит из 355 аминокислотных остатков, что составляет 99% от общей длины полученной последовательности (от 358).
Карта локального сходства между query и выбранной находкой
Помимо поиска гомологов, с помощью BLAST можно построить карту локального сходства двух выбранных последовательностей. Карта локального сходства белка дегидратазы из организма C. aurantiacus и Magnetospirillum sp. представлена на Рисунке 3.

Рисунок 3. Карта локального сходства.
Ось абцисс соответствует исходной последовательности, ось ординат — лучшей находке.
Результат получен с помощью программы BLAST.
Из Рисунка 3 можно сделать вывод, что последовательности гомологичны на всём своём протяжении, что говорит о гомологии этих последовательностей. Небольшим подъёмам линии соответствуют "гэпы". Вторая линия говорит о том, что фрагмент query (примерно с 186 по 317) также выравнивается с находкой.
Эукариотические гомологи
Для поиска гомологов белка дегидратазы среди эукариот в поле "Organism" был добавлен таксон "eucaryotes (taxid:2759)". В итоге было найдено 13 белков, 3 из которых подходили по E-value под параметры хорошей находки. Это белки из морской диатомовой водоросли (Thalassiosira pseudonana CCMP1335) и клещевины (Ricinus communis). Исходя из названий, это предполагаемые белки.
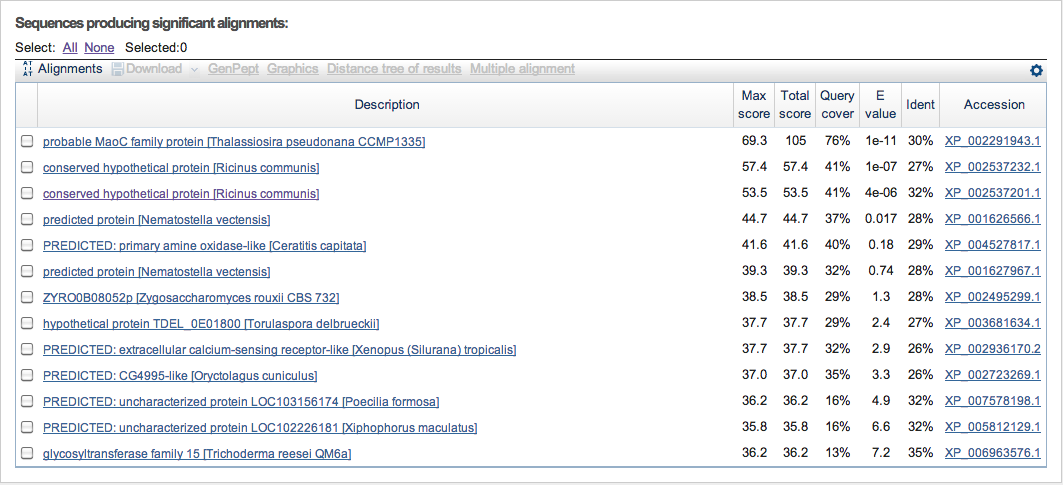
Рисунок 4. Результат поиска гомологов среди эукариот.
Результат получен с помощью программы BLAST.
Множественное "выравнивание" находок
С помощью сервиса COBALT было построено множественное выравнивание 10 найденных последовательностей (включая исходную). На Рисунке 5 приведено его изображение. Скачать выравнивание можно отсюда.

Рисунок 5. Выравнивание гомологов .
Выравнивание получено с помощью сервиса COBALT, изображение — программы JalView. Окрашивание BLOSUM62, Conservation 70%.
Абсолютно консервативных позиций — 139, что составляет 35% от длины всего выравнивания (401); функционально консервативных колонок — 195 (49%).
[1] http://blast.ncbi.nlm.nih.gov/Blast.cgi
[2] Draft Genome Sequence of Magnetospirillum sp. Strain SO-1, a Freshwater Magnetotactic Bacterium Isolated from the Ol'khovka River, Russia.
Grouzdev DS1, Dziuba MV, Sukhacheva MS, Mardanov AV, Beletskiy AV, Kuznetsov BB, Skryabin KG.