PSI-BLAST
PSI-BLAST — один из алгоритмов программы BLAST, используемый для поиска гомологов поданной последовательности белка.
Сначала последовательности находятся по обычному алгоритму BLAST. Потом, основываясь на множественном выравнивании полученных последовательностей, вес которых оказался выше заданного в параметрах, он создаёт позиционно специфическую матрицу весов (PSSM). С помощью этой матрицы находятся уже другие последовательности[1].
Поиск семейства гомологов
Для работы с PSI-BLAST был выбран белок с идентификатором в базе данных Uniprot Q9QCL4. Это P10-белок из борнавируса, кодируемый открытой рамкой считывания.
Первый поиск был произведён в базе последовательностей Refseq. Результат интерации показан на Рисунке 1. Из всех найденных последовательностей, только одна была выше установленного порога E-value (0.005), чего недостаточно для построения PSSM.
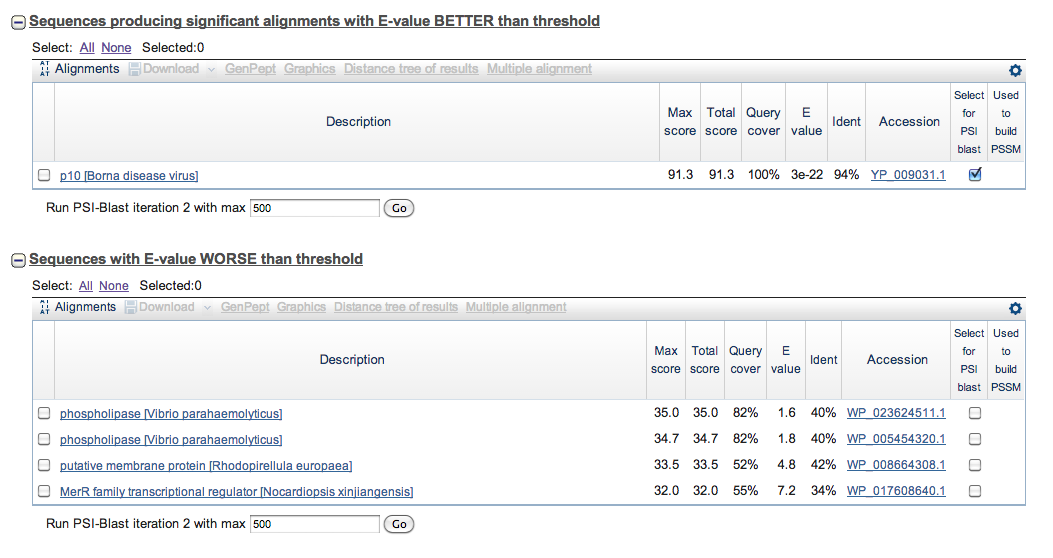
Рисунок 1. Результат поиска гомологов в базе данных Refseq.
Изображение получено с помощью программы BLAST (алгоритм PSI-BLAST).
Поэтому было решено для поиска использовать базу данных nr, которая включает в себя такие базы данных, как SwissProt, PDB, PIR и другие.
В Таблице 1 содержится информация, полученная после каждой проведённой интерации. Для построения PSSM использовались белки, в названиях которых присутствовали "X protein" или "P10 protein", так как белки именно с такими названиями относятся к семейству исходного[2].
| Номер интерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value данной находки | Идентификатор лучшей находки ниже порога | E-value данной находки | 1 | 41 | AFD63129.1 | 3*10-9 | ACG59349.1 | 0.006 |
| 2 | 50 | ADB84604.1 | 0.002 | AFN70789.1 | 0.007 | 3 | 55 | AFN70805.1 | 3*10-5 | BAM72615.1 | 2.3 |
| 4 | 55 | AFN70805.1 | 9*10-8 | BAM72615.1 | 0.47 |
| 5 | 55 | AFN70805.1 | 10-7 | BAM72615.1 | 0.64 |
Из Таблицы 1 видно, что количество находок стабилизировалось после четвёртой интерации. Наибольшая разница между E-value худшей "правильной" находкой и лучшей "неправильной" получена после пятой интерации (примерно 0.64), что говорит о большой вероятности, что "правильные" находки составляют семейство гомологичных белков.
Выравнивание семейства гомологов
Из полученных после пятой интерации последовательностей с помощью сервиса COBALT было построено множественное выравнивание, которое представлено на Рисунке 2. Скачать проект JalView с данным выравниванием можно отсюда.
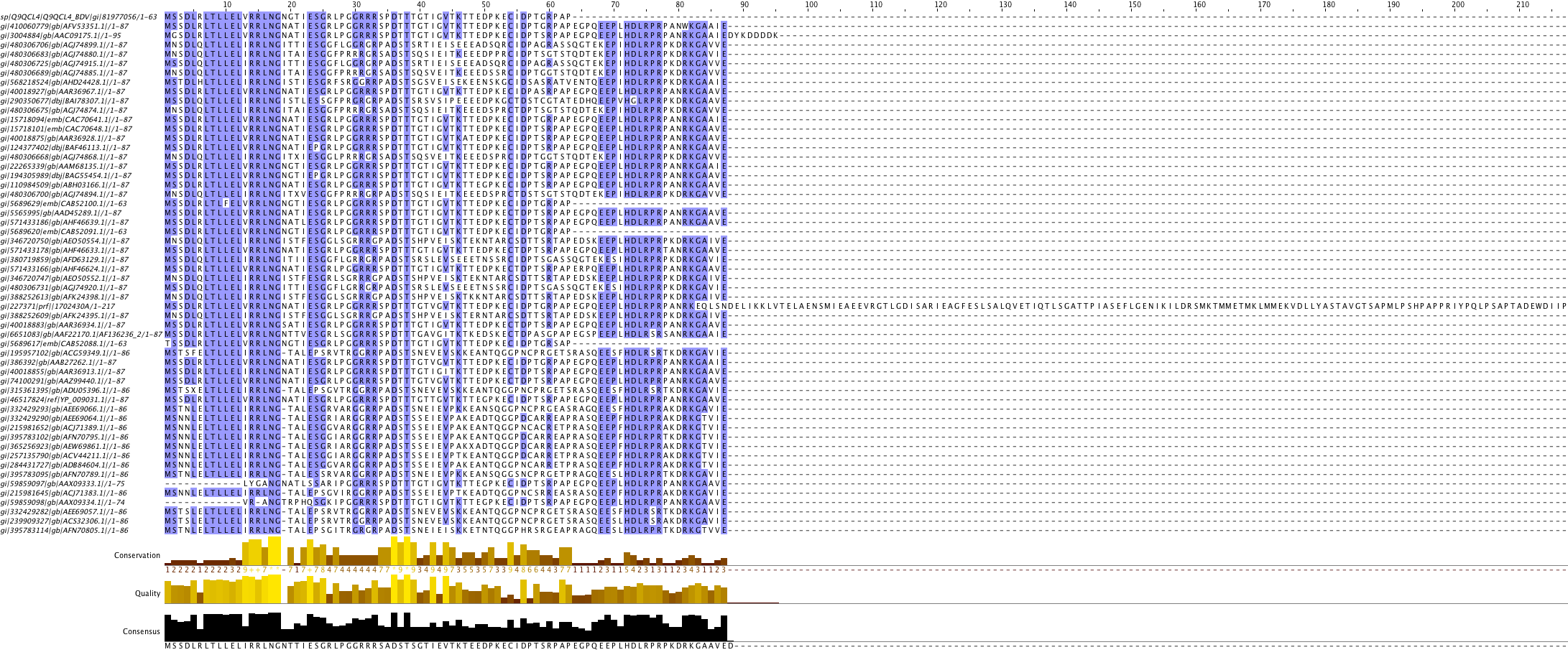
Рисунок 2. Выравнивание семейства гомологов белка P10.
Выравнивание получено с помощью сервиса COBALT, изображение — программы JalView. Окрашивание BLOSUM62, Above Identity Threshold 70%.
[1] Comparative Genomics: Volumes 1 and 2. Chapter 10
Bergman NH, editor.
Totowa (NJ): Humana Press; 2007.