Предсказание генов у прокариот
В данном практикуме гены предсказывались для контига микробиома отдела желудка валлаби (Wallabia bicolor). Использовались программы ORF Finder (getorf) совместно с blastp и GeneMark.
ORF Finder
С помощью программы ORF Finder можно найти открытые рамки считывания для заданной последовательности ДНК. Для нашей последовательности мы использовали генетический код под номером 11 (Bacterial Code). Это значит, что начало трансляции допускается с AUG, GUG, UUG, CUG, AUU. Результат, который выдала программа, представлен на Рисунке 1. Шесть белых полосок изображают исходную последовательность в шести возможных рамках считывания: +1, +2, +3 — прямая цепь; -1, -2, -3 — обратная. Бирюзовые полоски — найденные открытые рамки считывания (ORF) в данной рамке.
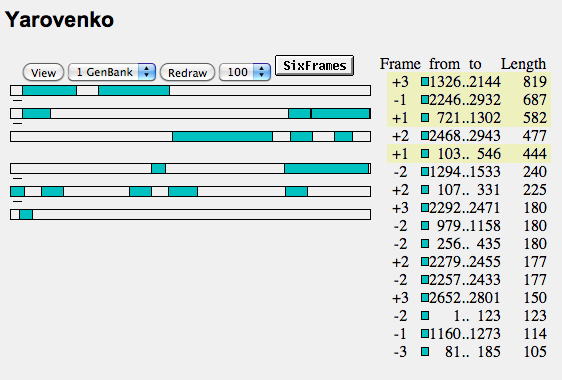
Рисунок 1. Открытые рамки считывания.
Шесть белых полосок изображают исходную последовательность в шести возможных рамках считывания: +1, +2, +3 — прямая цепь; -1, -2, -3 — обратная. Бирюзовые полоски — найденные открытые рамки считывания (ORF) в данной рамке. Жёлтым цветом выделены используемые далее ORF.
Изображения получены с помощью программы ORF Finder.
- Для дальнейшей работы были взяты 4 рамки считывания по следующим критериям:
- Длина рамки не короче 180 нуклеотидов
- Если рамка перекрывается с другой более, чем на половину, то выбирается более длинная
- Длина рамки кратна 3
Для этих ORF был запущен blastp по базе данных Swiss-Prot. В Таблице 1 приведены лучшие результаты поиска — предполагаемые гомологи.
| Начало | Конец | Длина (а.о.) | Цепь | Описание | ID гомолога | Организм | Вес | Покрытие | E-value | Сходство |
| 103 | 546 | 146 | + | гипотетический белок | WP_010455039.1 | Succinivibrionaceae bacterium WG-1 | 119 | 99% | 2x10-30 | 41% |
| 721 | 1302 | 193 | + | транспозаза | WP_013655574.1 | Cellulosilyticum lentocellum | 267 | 100% | 2x10-87 | 69% |
| 1326 | 2144 | 272 | + | интеграза | WP_027439571.1 | Lachnospiraceae bacterium AC2031 | 474 | 100% | 2x10-166 | 82% |
| 2246 | 2932 | 228 | - | АТФ-связывающий кассетный транспортер | WP_015514628.1 | Coprococcus catus | 273 | 98% | 2x10-87 | 59% |
Хорошей находка считалась, если её E-value был меньше 0,001, покрытие не менее 80%. Организмы, представленных в таблице родов, можно встретить в пищевом тракте животных, что весьма логично.
GeneMark
Также гены данного контига были предсказаны с помощью программы GeneMark с эвристическими параметрами 1999 года. Программа выдала данный файл с координатами предсказанных генов, а также pdf-файл с графиком кодируюещего потенциала. График представлен на Рисунке 2. Данные из файла занесены в Таблицу 2.
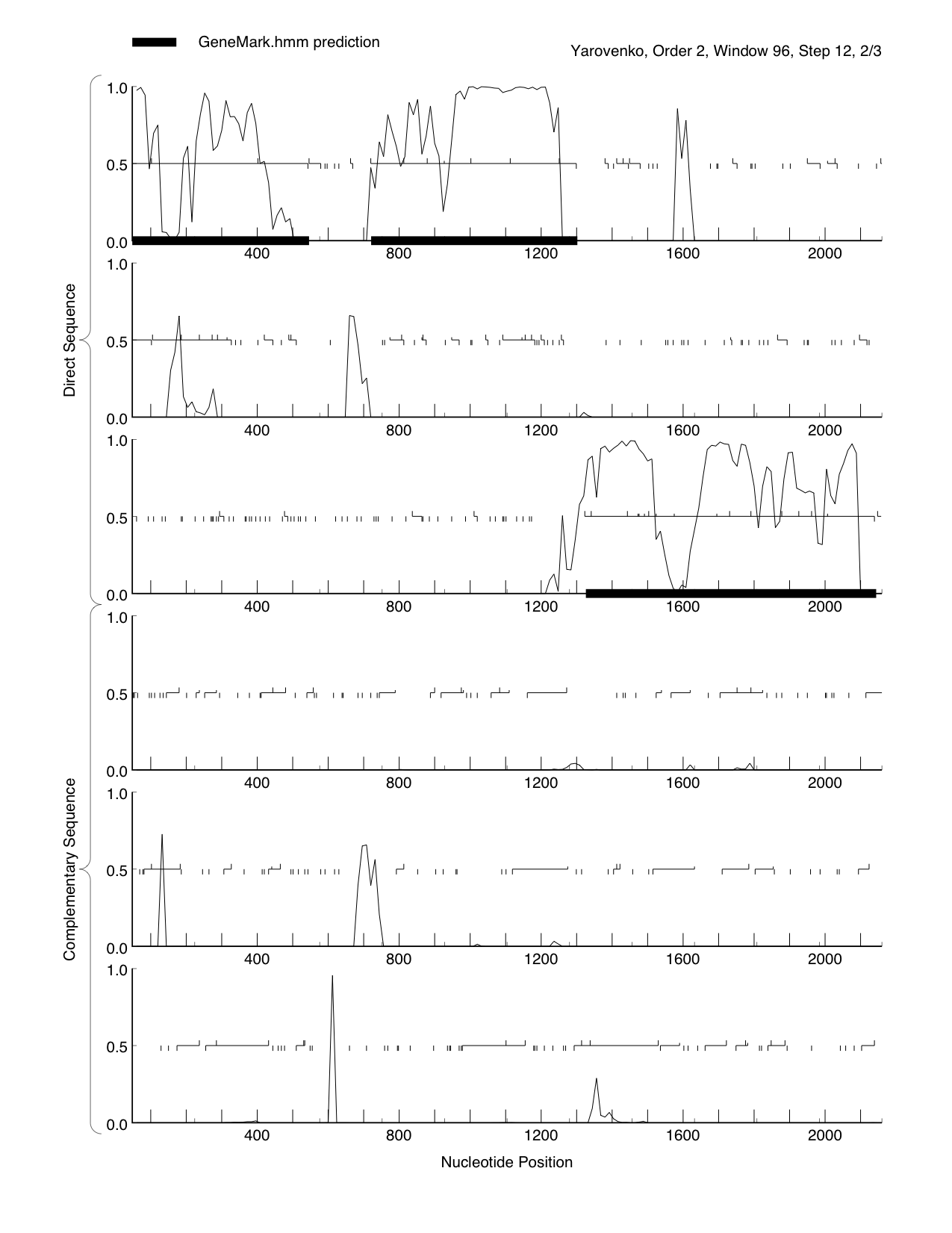
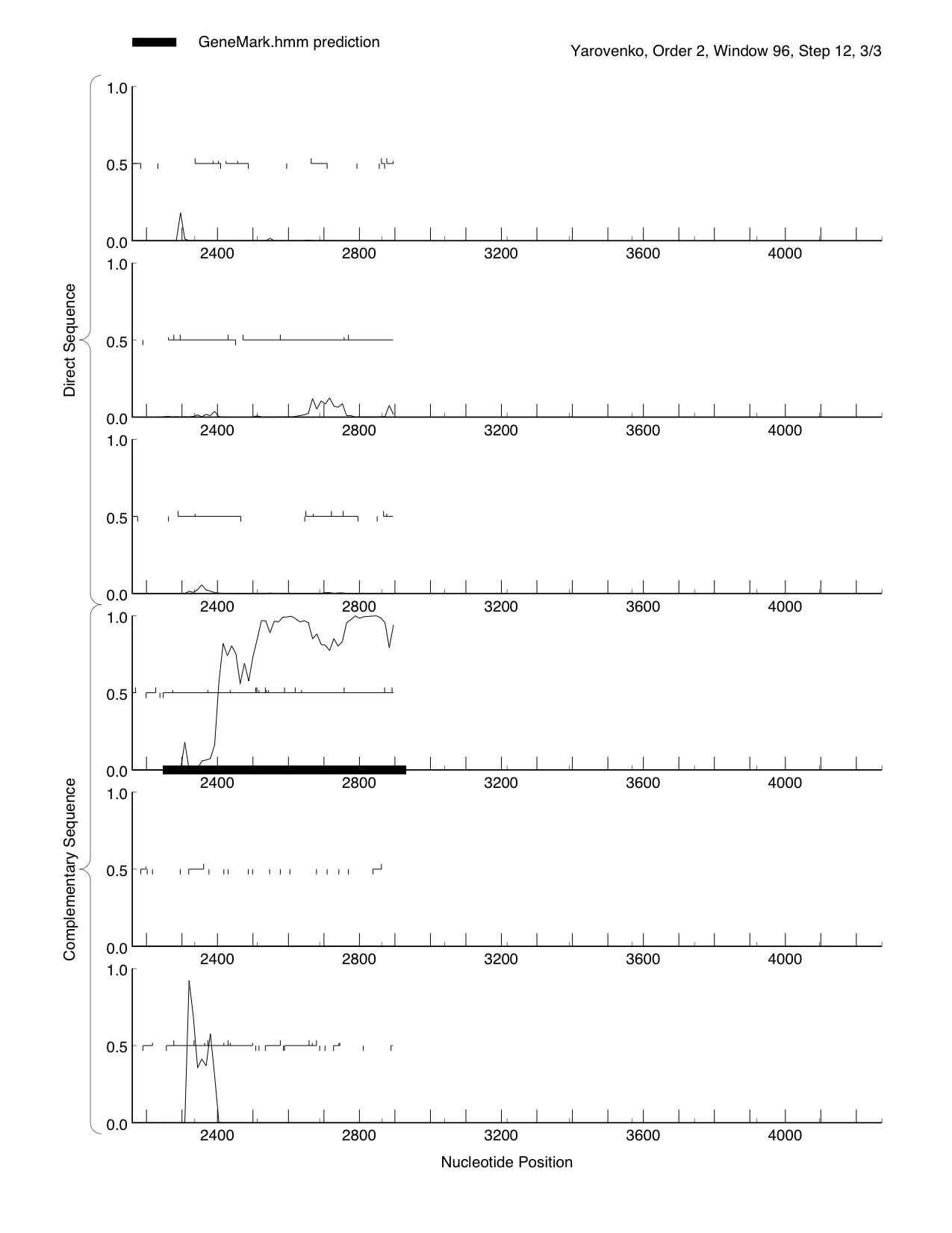
Рисунок 2. График кодирующего потенциала.
Изображение получено с помощью программы Geneark. Эвристические параметры 1999 года (Heuristic parameters, as in 1999 publication).
| Начало | Конец | Длина (а.о.) | Цепь |
| <1 | 546 | 182 | + |
| 721 | 1302 | 193 | + |
| 1326 | 2144 | 272 | + |
| 2246 | 2932 | 228 | - |
Если сравнить результаты, представленные в Таблице 1 и Таблице 2, то видно, что они почти идентичны. Отличаются лишь координаты первого гена. Отчёт программы GeneMark говорит, что ген начинается за пределами контига, ORF Finder и blastp показывает противоположное. Причина такого результата вероятней всего связана с алгоритмом самого blastp, а также с "криворукостью" исследователя. Первый ген выделен в обеих таблицах голубым.
Впоследствии программа GeneMark была запущена с эвристическими параметрами 2010 года, но, увы, результаты никак не поменялись.