Предсказание генов у эукариот
В данном практикуме экзон-интронная структура генов была предсказана для фрагмента ДНК из генома человека (Homo sapiens) и фрагмента ДНК из генома киви (Actinidia chinensis).
Сначала поработаем с фрагментом ДНК из генома человека.
GENSCAN
С помощью онлайн сервиса GENESCAN можно установить экзон-интронную структуру для последовательности ДНК. Программу запускаем с параметрами по умолчанию и на выходе получаем список экзонов с уже определёнными координатами и типами: начальный (Init), внутренний (Intr), конечный (Term). Также в результате встречаются тип PlyA (сайт полиаденилирования) и Prom (промотор). они не являются экзонами, так что мы исключаем их. В Таблицу 1 занесены начало (Begin), конец (End), цепь (S) и тип (Type) всех предсказанных программой экзонов.
| Начало | Конец | Цепь | Тип |
| 2589 | 2447 | - | Конечный |
| 15857 | 15693 | - | Внутренний |
| 16625 | 16562 | - | Внутренний |
| 20390 | 20268 | - | Внутренний |
| 21988 | 21804 | - | Внутренний |
| 23091 | 22933 | - | Внутренний |
| 29533 | 29320 | - | Внутренний |
| 30732 | 30614 | - | Внутренний |
| 43132 | 43069 | - | Конечный |
Genome Browser
Также определить экзон-интронную структуру заданного участка ДНК можно с помощью программы BLAT, которая находится на сайте базы Genome Browser. Выбираем из результатов тот, у которого схожесть с нашей последовательностью 100%, а также имеющий наибольший вес (в нашем случае 40250). Далее можно перейти к просмотру найденного фрагмента ДНК в геномном браузере, нажав на ссылку "browser". Изображение исследуемого участка представлено на Рисунке 1.

Рисунок 1. Исследуемый участок ДНК и соответствующие ему последовательности мРНК.
Чёрными прямоугольниками обозначены экзоны. Фиолетовым цветом выделен участок, где происходит альтернативный сплайсинг. Также в рамочку и жёлтым выделены случаи альтернативных акцепторных сайтов.
Изображения получены с помощью Genome Browser и программы BLAT.
С помощью мРНК мы можем говорить об альтернативном сплайсинге нашего фрагмента. Рассмотрим на Рисунке 1 область, выделенную фиолетовым цветом. У мРНК AK295038 отсутствуют два экзона, которые есть у всех остальных. Значит, эти экзоны — кассетные. В области, выделенной жёлтым цветом мы видим альтернативные акцепторные сайты (последний экзон у трёх групп мРНК различается).
BLASTX
Для дальнейшей работы был предоставлен фрагмент ДНК из генома киви Actinidia chinensis (фрагмент можно загрузить отсюда). Так как геном киви недостаточно хорошо аннотирован, такими инструментов, как GENESCAN или Genome Browser, для данного генома попросту нет. Так что перед нами стояла увлекательнейшая задача: при помощи программы BLASTX проаннотировать этот фрагмент, разметив примерную экзон-интронную структуру генов, а также предсказать их функцию.
BLASTX ищет гомологичные белковые последовательности, которые соответствуют участкам поданного фрагмента ДНК. В нашем случае программа была запущена со следующими параметрами: был исключён (Exclude) поиск по моделям и пробам среды (галочки возле Models (XM/XP) и Uncultured/environmental sample sequences), поиск был ограничен только по белкам растений (Viridiplantae); из поиска был также исключён геном винограда Vitis vinifera.
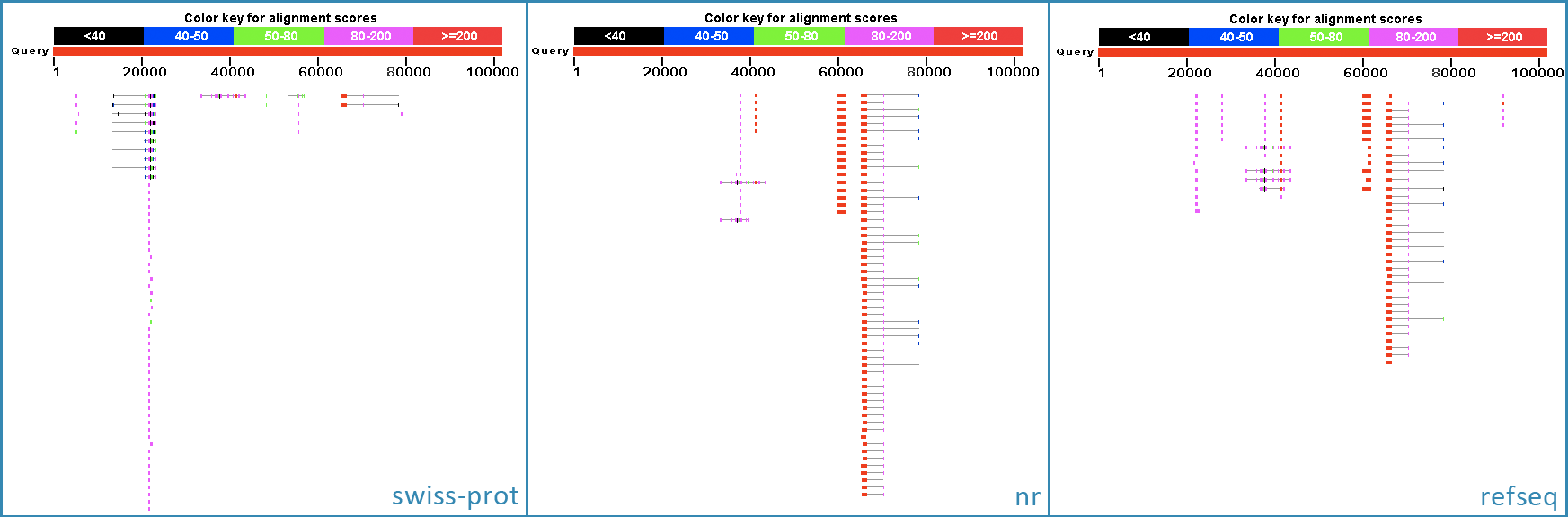
BLASTX запускался несколько раз по различным базам данных (сначала Swiss-Prot, потом RefSeq, nr). Результат поиска представлен на Рисунке 2.
Рисунок 2. Результаты работы программы BLASTX по различным базам данных.
Слева приведён график качества нуклеотидов для чтений из исходного файла, справа — график для уже очищенного файла. При нажатии на картинку открывается изображение большего размера.
Изображения получены с помощью программы BLASTX.
Видно, что по банку Swiss-Prot хорошо разметилось начало фрагмента. Для других, наоборот, только конец. Поэтому для выявления предполагаемых генов и их структур будем пользоваться сразу всеми результатами.
Все полученные результаты были разбиты по группам ("генам") относительно координат участков ДНК, с которыми они выравнялись. Из каждой такой группы выбирался лучший кандидат по следующим критериям: функция белка должна быть известна, E-value << 0.001, самый высокий в своей группе процент сходства (Ident). Далее по этим лучшим гомологам выстраивалась предполагаемая интрон-экзонная структура гена. Предполагаемого экзоны располагались в порядке возрастания координат по белку (Subject start). Их границы уточнялись вручную.
Рассмотрим, например, случай перекрывания первых двух предполагаемых экзонов из предсказанного белка Derlin-2.1 (Рисунок 3). "Экзоны" пересекаются по белковой координате, поэтому, чтобы определить границы наших экзонов, посмотрим на качество выравнивания. Видно, что выравнивание, начиная с 34 белковой координаты значительно лучше у второго экзона, поэтому отдаём кусок 34-43 ему. Аналогичные операции были проделаны ещё с несколькими экзонами. Также экзоны объединялись, если перекрывание по белкам было незначительное (1-3 аминокислоты), а ДНК-координаты были последовательными (шли друг за другом).
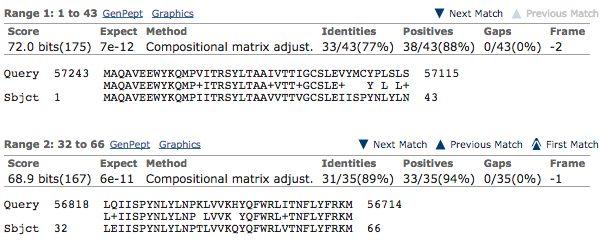
Рисунок 3. Пример перекрывания двух "экзонов" по белку в предсказанном белке Derlin-2.1.
Изображения получены с помощью программы BLASTX.
Итог всей этой утомительной операции представлен в Таблице 2. Особенно отличился ген предполагаемой фосфолипазы A I, у которго аж 16 экзонов! Всего удалось предсказать 6 генов, но их в действительности может быть больше, так как многие кандидаты, которые хорошо выравнялись на наш участок генома, были исключены в виду плохой аннотированности или неясной функции. Также в таблицу не вошли два белка тобамовируса (Tobamovirus multiplication protein 1) из двух разных растений (Nicotiana tabacum и Arabidopsis thaliana).
| Предсказанный глюкоза-6-фосфат транслокатор 1 по белку Arabidopsis thaliana NP_568812.1 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 4244 | 4746 | + |
| 2 | ДНК | 5384 | 5611 | + |
| 3 | ДНК | 5964 | 6182 | + |
| 4 | ДНК | 7096 | 7311 | + |
| Предсказанная бета-глюкозидаза 16 по белку Oryza sativa Japonica Group NP_001053303.1 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 13780 | 13911 | + |
| 2 | ДНК | 20723 | 20797 | + |
| 3 | ДНК | 20905 | 20979 | + |
| 4 | ДНК | 21063 | 21149 | + |
| 5 | ДНК | 21893 | 22150 | + |
| 6 | ДНК | 22281 | 22751 | + |
| 7 | ДНК | 22844 | 22948 | + |
| 8 | ДНК | 23052 | 23135 | + |
| 9 | ДНК | 23450 | 23599 | + |
| Предсказанная фосфолипаза A I по белку Arabidopsis thaliana NP_001185288.1 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 33608 | 33949 | + |
| 2 | ДНК | 36098 | 36307 | + |
| 3 | ДНК | 36602 | 36685 | + |
| 4 | ДНК | 36798 | 36962 | + |
| 5 | ДНК | 37066 | 37287 | + |
| 6 | ДНК | 37379 | 37624 | + |
| 7 | ДНК | 37720 | 37869 | + |
| 8 | ДНК | 37964 | 38227 | + |
| 9 | ДНК | 38316 | 38438 | + |
| 10 | ДНК | 39425 | 39589 | + |
| 11 | ДНК | 39709 | 39897 | + |
| 12 | ДНК | 40019 | 40165 | + |
| 13 | ДНК | 40959 | 41096 | + |
| 14 | ДНК | 41276 | 42040 | + |
| 15 | ДНК | 42157 | 42573 | + |
| 16 | ДНК | 43650 | 44090 | + |
| Предсказанный мульти-пропускной мембранный белок Derlin-2.1 по белку Arabidopsis thaliana NP_193912.3 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 57243 | 57069 | - |
| 2 | ДНК | 56818 | 56714 | - |
| 3 | ДНК | 56009 | 55779 | - |
| 4 | ДНК | 55689 | 55603 | - |
| 5 | ДНК | 53491 | 53342 | - |
| Предсказанный белок, содержащий домен цинковых пальцев MYND-типа, по белку Morus notabilis XM_010103139.1 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 62072 | 60171 | - |
| Предсказанный АТФ-связывающий кассетный транспортер ABCI.8 по белку Morus notabilis XM_010094977.1 |
||||
| № экзона | Координаты | Начало | Конец | Цепь |
| 1 | ДНК | 65421 | 66866 | + |
| 2 | ДНК | 70425 | 70637 | + |