Анализ качества и очистка чтений
Анализ качества чтений и их очистка проводились для файла c ридами генома резуховидки (Arabidopsis thaliana) — Ath_tae_CTTGTA_L003_R1_006.fastq. Контроль качества данных чтений был сделан с помощью программы FastQC. Результат можно посмотреть по ссылке.
Из отчёта программы можно почерпнуть такую важную информацию, как количество последовательностей, их длина, процент нуклеотидов G и C и т.д.
- Для исходного файла с чтениями статистика такова:
- Количество последовательностей: 4000000
- Длина последовательностей: 101
- % GC: 35
Далее был сделан тримминг (т.е. чистка) данных чтений с помощью программы Trimmomatic. Сначала все адаптеры были перенесены в одну папку, а потом помещены в один файл adapter.fa:
cp /P/y13/term3/block4/adapters/* term3/block4/pr12; cat *.fa > adapter.fa
Потом для очистки была использована следующая команда:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 in.fastq out.fastq
ILLUMINACLIP:adapter.fa:2:7:7 TRAILING:20 MINLEN:50
- in.fastq и out.fatsa — названия входного и выходного файла соответственно
- опция ILLUMINACLIP:adapter.fa:2:7:7 вырезает из файла последовательности адаптеров из adapter.fa
- TRAILING:20 удаляет нуклеотиды с качеством ниже 20
- MINLEN:50 убирает прочтения длиной меньше 50 нуклеотидов
"Очищенный" файл пропустили через программу FastQC. Отчёт программы можно увидеть здесь.
- Статистика для нового файла с чтениями:
- Количество последовательностей: 2533835
- Длина последовательностей: 50-101
- % GC: 35
Как видно, изменилось количество чтений (уменьшилось почти в 1,5 раза) и длина ридов. Процент гуанина и цитозина в последовательностях сохранился.
Сравним отдельно несколько графиков из отчётов программы FastQC.
Качество нуклеотидов отражает график Per base sequence quality. График из двух отчётов представлен на Рисунке 1. Как видно из Рисунка 1, качество нуклеотидов немного улучшилось (минимальная оценка качества нескольких нуклеотидов вышла из оранжевой зоны).
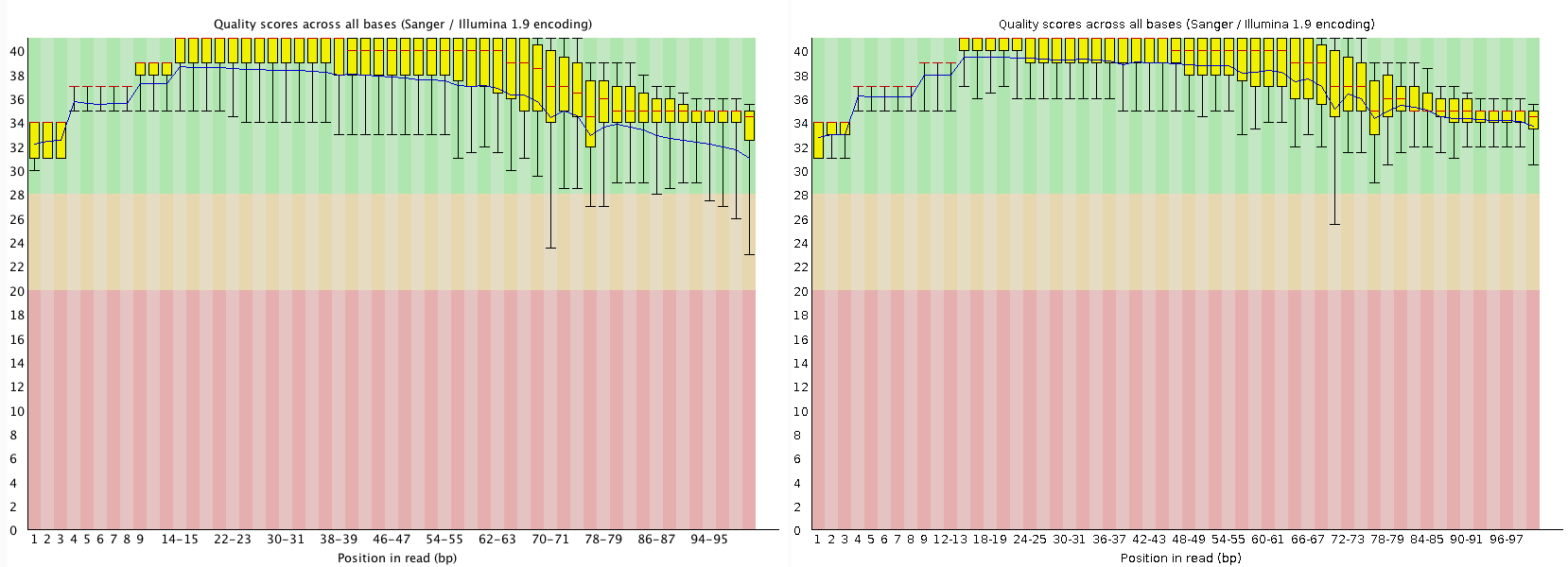
Рисунок 1. Графики "Per base sequence quality".
Слева приведён график качества нуклеотидов для чтений из исходного файла, справа — график для уже очищенного файла.
Изображения получены с помощью программы FastQC.
Графики Per base sequence content, которые показывают частоту встречаемости каждого из четырёх нуклеотидов, абсолютно одинаковые.
Теперь сравним графики "Sequence length distribution", которые показывают распределение длин чтений. Они представлены на Рисунке 2. Мы видим, что до улучшения все последовательности имели длину 101, а после — появились ещё и последовательности длиной 50-100 нукеотидов. Но всё равно последовательностей длины 100-101 значительно больше, чем всех остальных.
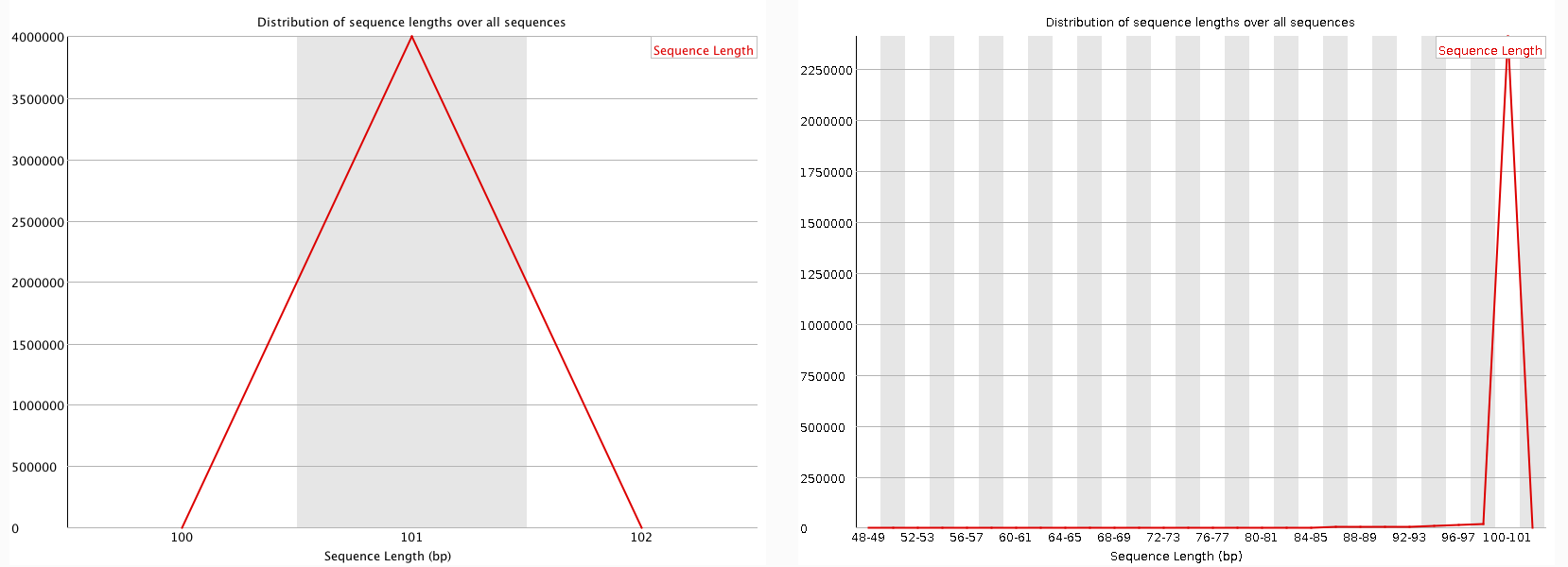
Рисунок 2. Графики "Sequence length distribution".
Слева приведён график качества нуклеотидов для чтений из исходного файла, справа — график для уже очищенного файла.
Изображения получены с помощью программы FastQC.
Таким образом, сравнив "улучшенный" и исходный файл, мы можем предположить, что изначально качество чтений было высоким (отчёты программы FastQC довольно схожи).