Геномное окружение. База данных STRING
В данном задании анализировалось геномное окружение белка дегидратазы (локус гена: Caur_0173) из генома Chloroflexus aurantiacus, штамм J-10-fl (см. предыдущий практикум).
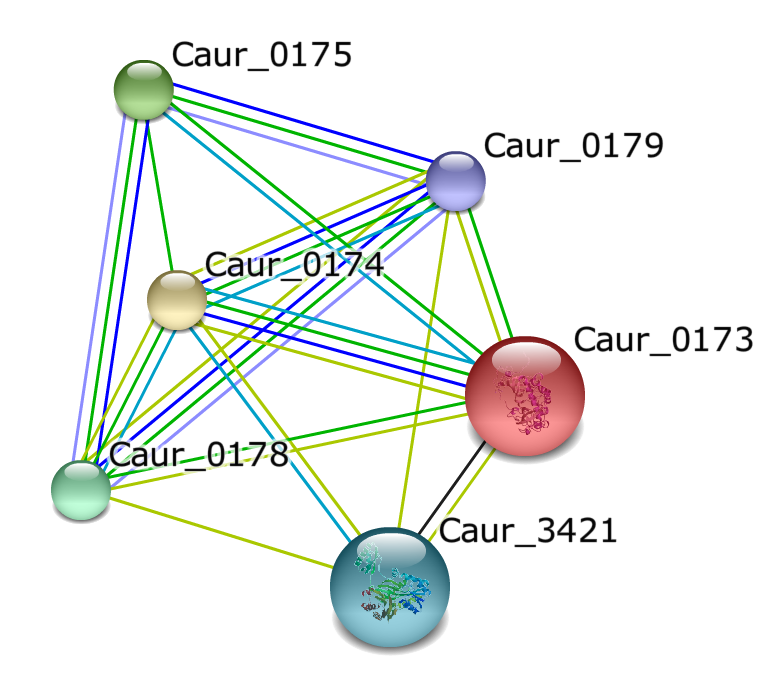
Рисунок получен с помощью БД STRING.
Поиск предполагаемых белков-партнёров осуществлялся в базе данных STRING. На рисунке 1 представлен полученный граф взаимодействия этого белка с найденными партнёрами на первом уровне близости. Ниже объясняется, что обозначают круги (белок и его название) и рёбра (взаимодействия).
- Caur_0173 — дегидратаза
- Caur_0174 — HpcH/HpaI альдолаза
- Caur_0175 — L-карнитин дегидратаза или индуцируемый желчными кислотами белок F
- Caur_0178 — L-карнитин дегидратаза или индуцируемый желчными кислотами белок F
- Caur_3421 — ацетил-КоА карбоксилаза и биотин карбоксилаза
- Caur_0179 — L-карнитин дегидратаза или индуцируемый желчными кислотами белок F
Рёбра зелёного цвета говорят о том, что белки располагаются рядом в геноме, тёмно-синие — о совместной встречаемости белков, черные — о коэкспрессии, голубые — о совместном упоминании белков в базах данных, жёлтые — об упоминании белков в одной статье, светло-голубые — о гомологии.
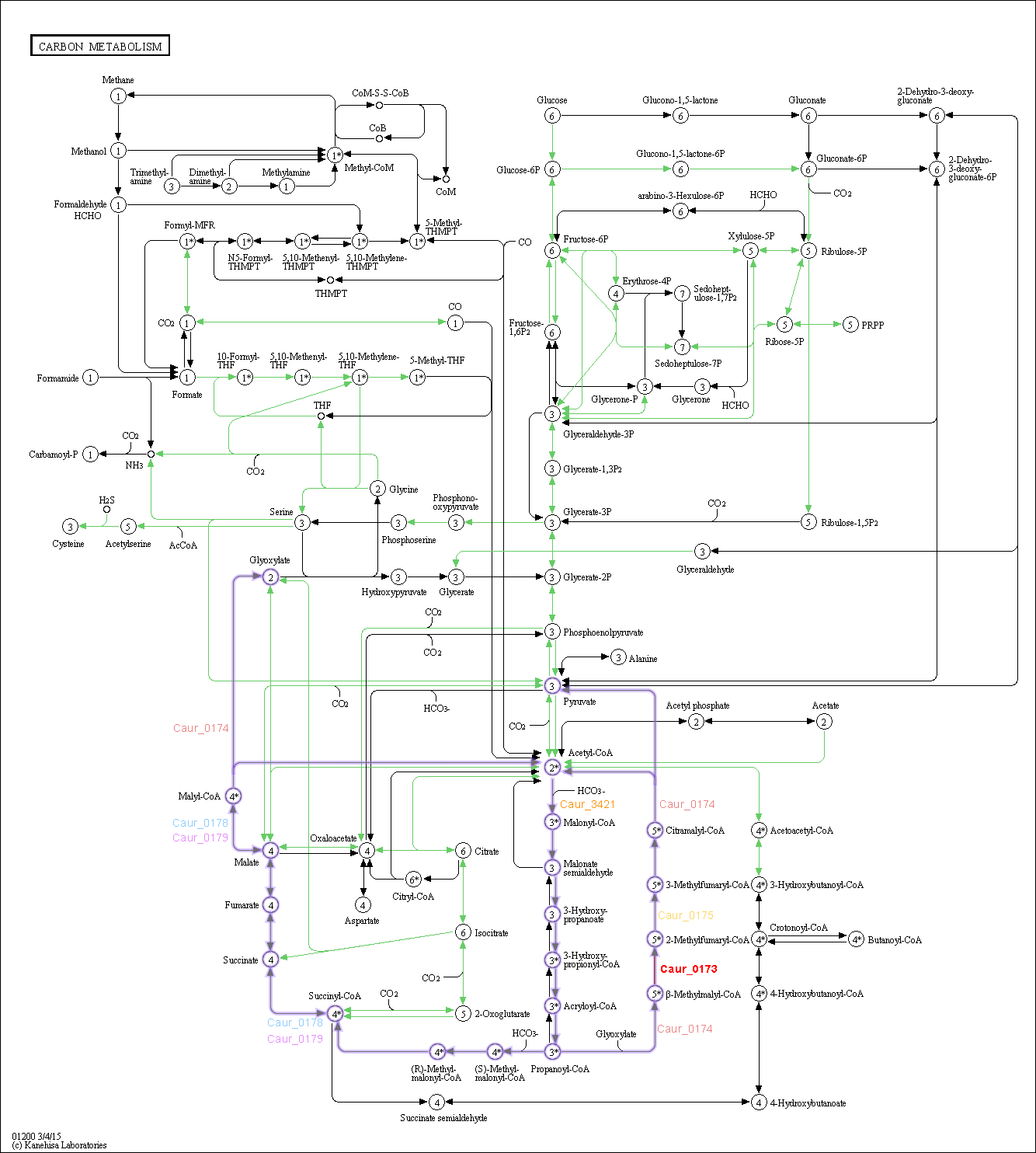
Все найденные белки участвуют в пути фиксации углерода (map01200), а именно в цикле 3-гидроксипропионата[1] (M00376). Схема этого пути представлена ниже на рисунке 2. Причём белки с локусами Caur_0174, Caur_0173 и Caur_0175, как видно на рисунке, катализируют последовательно идущие реакции. В геноме, как было показано ранее, они также расположены рядом с друг другом. Это также показывает и полученный граф (на рисунке 1 рёбра, соединяющие эти белки, зелёного цвета).
Рисунок 2. Фиксация углерода.
Зелёным цветом обозначен полный путь для бактерии Chloroflexus aurantiacus. Фиолетовым цветом выделен цикл 3-гидроксипропионата. Буквами другого цвета отмечен сам белок, а также его предполагаемые функциональные партнёры. При наведении мышки на картинку окрашиваются реакции, которые катализируют эти белки. Нажав на изображение, можно увидеть эту картинку в исходном размере.
Рисунок получен с помощью сервиса KEGG.
Далее было рассмотрено геномное окружение белка дегидратазы. Оно представлено на рисунке 3. Дерево со раскрытыми ветвями, где присутствуют найденные белки, можно посмотреть здесь. Ниже находятся обозначения.
- белки, ортологичные Caur_0173 — дегидратазе
- белки, ортологичные Caur_0174 — HpcH/HpaI альдолазе
- белки, ортологичные Caur_0175 — L-карнитин дегидратазе или индуцируемому желчными кислотами белку F
- белки, ортологичные Caur_0178 — L-карнитин дегидратазе или индуцируемому желчными кислотами белку F
- белки, ортологичные Caur_3421 — ацетил-КоА карбоксилазе и биотин карбоксилазе
- белки, ортологичные Caur_0179 — L-карнитин дегидратазе или индуцируемому желчными кислотами белку F
- другие белки, входящие в оперон, но с недостаточным подтверждением

Рисунок 3. Геномное окружение Caur_0173 (NC_010175.1).
Рисунок получен с помощью БД STRING.
Из рисунка 3 видно, что Caur_0179, Caur_0178, Caur_0175, Caur_0174, Caur_0173 находятся в одном опероне (филум Chloroflexi). Это соответствует ранее полученным данным. У протеобактерий есть оперон примерно с теми же белками. Все найденные белки присутствуют у актинобактерий. Также эти белки встречаются у автотрофных архей, что весьма логично. Например, у Halobacterium salinarum и несколько других галобактерий так же способны фиксировать углерод через биосинтез пирувата [2, с.145]. А для бактерии Sulfolobus metallicus была показана ацетил-КоА активность[3].
Далее было построено дерево совместной встречаемости данного белка, которое представлено на рисунке 4. Чем интенсивнее цвет квадратика, тем более гамологичен белок тому, что указан сверху. Если не у всех организмов из токсина есть похожий белок, то угол квадратика "откусан".
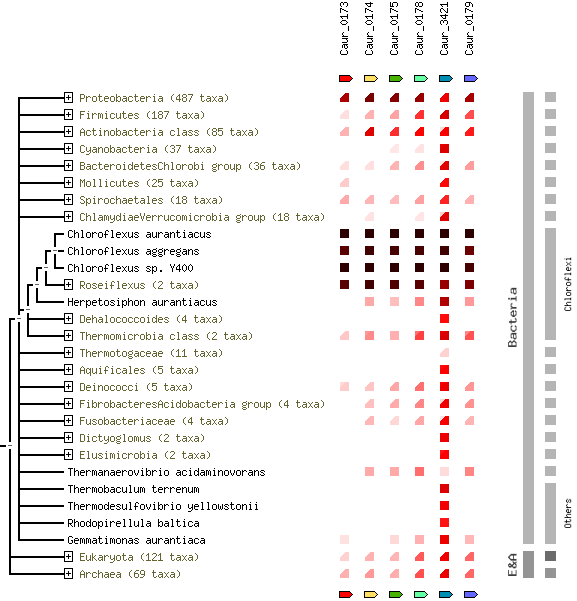
Рисунок 4. Совместная встречаемость Caur_0173 (NC_010175.1) и других найденных белков.
Фиолетовым цветом обозначается нетривиальная ветвь, образующая кладу из белков одного ортологического ряда. Зелёным — заинтересовавшие меня по длине тривиальные ветки.
Рисунок получен с помощью программы Mega5.1. Дерево построено по алгоритму Neighbour-Joining в программе Mega5.1.
По рисунку 4 видно, что белок с локусом Caur_3421 (ацетил-КоА карбоксилаза и биотин карбоксилаза) встречается почти во всех организмах. Сама по себе эта лигаза по сравнению с другими белками участвует в большем количестве метаболических путей (Caur_3421), в том числе в биосинтезе и метаболизме жирных кислот.
[1] D. S. Kim and Y. Hahn, “Human-specific protein isoforms produced by novel splice sites in the human genome after the human-chimpanzee divergence.,” BMC Bioinformatics, vol. 13, no. 1, p. 299, 2012.
[2] A. Oren, The Order Halobacteriales. 2006.
[3]C. Menendez, Z. Bauer, H. Huber, N. Gad’on, K.-O. Stetter, and G. Fuchs, “Presence of Acetyl Coenzyme A (CoA) Carboxylase and Propionyl-CoA Carboxylase in Autotrophic Crenarchaeota and Indication for Operation of a 3-Hydroxypropionate Cycle in Autotrophic Carbon Fixation ,” J. Bacteriol. , vol. 181 , no. 4 , pp. 1088–1098, Feb. 1999.