» Семестры » Восьмой семестр » Анализ молекулярной динамики биологических молекул в GROMACS
Анализ молекулярной динамики биологических молекул в GROMACS¶
- Силовое поле, используемое при построении топологии: lipid.itp (ffgmxnb.itp)
- Заряд системы: 0 из-за амфифильности молекулы.
- Размер и форма ячейки: 6.26x4.443x5.778, параллелепипед
- Минимизация энергии
- Алгоритм минимизации энергии: integrator = steep
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: Cut-off
- Модель, которой описывался растворитель: spc
- Утряска растворителя
- Число шагов: 1000
- Длина шага: 0.0002 ps
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: Cut-off
- Алгоритмы термостата и баростата: Tcoupl = V-rescale, Pcoupl = no ;Berendsen (anisotropic)
- Основной расчёт МД
- Длина траектории: 50000
- Число шагов: 10000000
- Длина шага: 0.005
- Алгоритм интегратора: md
- Алгоритм расчёта электростатики и Ван-дер-Ваальсовых взаимодействий: pme и Cut-off
- Алгоритмы термостата и баростата: Tcoupl = v-rescale, Pcoupl = Berendsen (semiisotropic)
Начнём с визуального анализа движений молекул:
%%bash
trjconv -f b_md.xtc -s b_md.tpr -o b_pbc_1.pdb -skip 20 -pbc mol
3 модель организована на подобии миуеллы, а уже на 30 модели 64 DPPC образовали что-то похожее на бислой:
MODEL 4 (t=1500 ps):
 MODEL 30 (t=14500 ps):
MODEL 30 (t=14500 ps):
Определим площадь, занимаемую одним липидом. Для этого получим размеры ячейки из траектории:
%%bash
g_traj -f b_md.xtc -s b_md.tpr -ob box_1.xvg
Для подсчёта площади определим, какая ось является нормалью к поверхности бислоя.
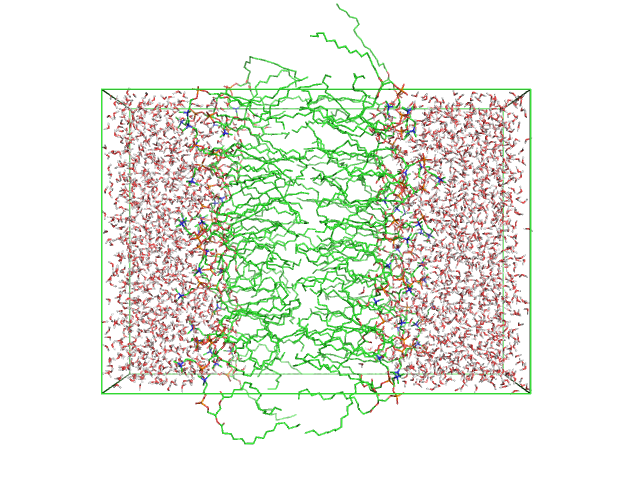 Видно, что это ось Х.
Видно, что это ось Х.
Построим зависимость площади по соотвествующим осям (Y и Z) от времени и нормируем это значание на один липид в слое.
with open("box_1.xvg","r") as box:
mod_time = []
surface = []
for line in box:
line = line.strip().split()
mod_time = mod_time+[int(line[0])]
surface = surface+[float(line[2])*float(line[3])/32]
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
x=np.array(mod_time)
y=np.array(surface)
#Plot it
plt.plot(x, y, "b-",alpha=0.5)
plt.ylabel('Surface area/32, nm^2')
plt.xlabel('Time, ps')
plt.show()
surface[-1]
Из графика видно, что значение площади уменьшается со временем, пока не достигает примерно 0.64 нм2.
Определим изменение гидрофобной и гидрофильной поверхности в ходе самосборки:
%%bash
g_sas -f b_md.xtc -s b_md.tpr -o sas_b.xvg
Построим зависимость изменения гидрофобной и гидрофильной поверхностей, доступных растворителю от времени:
with open("sas_b.xvg","r") as box:
mod_time_2 = []
hb_sas = []
hf_sas = []
for line in box:
line = line.strip().split()
mod_time_2 = mod_time_2+[int(line[0])]
hb_sas = hb_sas+[float(line[1])]
hf_sas = hf_sas+[float(line[2])]
[mod_time_2[x] for x in range(len(hf_sas)) if hf_sas[x]<hb_sas[x]]
x=np.array(mod_time_2)
y_1=np.array(hb_sas)
y_2=np.array(hf_sas)
plt.plot(x, y_1, "r-", alpha = 0.5, label="Hydrophobic")
plt.plot(x, y_2,"b-", alpha=0.5, label="Hydrophylic")
plt.ylabel('Solvent Accessible Surface, nm^2')
plt.xlabel('Time, ps')
plt.legend()
plt.show()
По графику видно, что после 150 ps, величина доступной растворителю гидрофобной поверхности становится меньше гидрофильной поверхности. За счет уменьшения гидрофобной поверхности происходит сборка липидов в бислой.
Традиционной мерой оценки фазового состояния амфифильных молекул является мера порядка. Для анализа понадобится специальный индекс файл.
Запустите сам анализ, где X - это ось, которая является нормалью к поверхности бислоя, для конца и начала траектории:
%%bash
g_order -s b_md -f b_md.xtc -o ord_end.xvg -n sn1.ndx -b 45000 -d X
g_order -s b_md -f b_md.xtc -o ord_start.xvg -n sn1.ndx -e 5000 -d X
Построим зависимость:
with open("ord_start.xvg","r") as order:
mol_num = []
s_start = []
for line in order:
line = line.strip().split()
mol_num = mol_num+[int(line[0])]
s_start = s_start+[float(line[1])]
with open("ord_end.xvg","r") as order:
s_end = []
for line in order:
line = line.strip().split()
s_end = s_end+[float(line[1])]
%pylab inline
pylab.rcParams['figure.figsize'] = (15, 5)
x=np.array(mol_num)
y_1=np.array(s_start)
y_2=np.array(s_end)
plt.subplot(1,2,1)
plt.plot(x, y_1, "ro", alpha = 0.5, label="start")
plt.ylabel('S')
plt.xlabel('DSSP atom number')
plt.legend()
plt.subplot(1,2,2)
plt.plot(x, y_2,"bo", alpha=0.5, label="end")
plt.ylabel('S')
plt.xlabel('DSSP atom number')
plt.legend()
plt.tight_layout()
plt.show()
Видно, что к концу моделирования значения S снизились на порядок, что говорит о более упорядоченном состоянии DPPC в конце. На двух графиках видно, что атомы на конце липида (жирный хвост) более подвижны, чем гидрофильная головка.
Дата последнего обновления: 21.05.17
© Светлана Яровенко