Обзор генома Lactobacillus crispatus ST1
1. Введение
Lactobacillus crispatus по большинству признаков является типичным представителем рода Lactobacillus. Морфология: грам-положительные неподвижные бациллы. Экология: мезофильные, факультативные анаэробы [1], одна из составляющих ротовой, кишечной и вагинальной микрофлоры [2]. Метаболизм: молочнокислое брожение [2].
Является H2O2-продуцирующей бактерией. Это, наряду с закислением среды в ходе брожения, позволяет использовать её как пробиотик для нормализации вагинальной микрофлоры [2] (в частности, показано [3], что в половых путях L. crispatus подавляет адгезию патогенной птичьей E. Coli; проводились исследования препарата, содержащего L. crispatus для лечения и профилактики гарднереллёза (бактериального вагиноза)). L. crispatus ST1 была выделена из куриного зоба. [3]
Геном содержит 2,043,161 п.н., кодирует 1834 белка [3] для примерно 1700 из которых известны функции. По другим данным – 2003 белка [4].
В данной работе проводится общий анализ генома с целью нахождения закономерностей расположения генов.
2. Материалы и методы
Для анализа использовались данные (хромосомная таблица) из базы NCBI Genomе [1], которые обрабатывались методами Microsoft Excel. Ниже перечислены использованные функции:
2.1 Получение данных
Для последующей обработки данные переводились в формат плоской таблицы (представлена на листе genes) при помощи функции ВПР. Строки типа gene сопоставлялись со строками типа CDS или RNA по идентификатору locus_tag, после чего были отсортированы по координате start.
2.2 Общая статистика
Для получения общей статистики генов различного типа применялась функция СЧЁТЕСЛИМН с условиями на тип гена (РНК, транслируемый, псевдоген и т.д.), цепь (+, -), молекулу (хромосома или плазмида).
2.3 Статистика типов белков
Для нахождения белков различных типов применялась функция СЧЁТЕСЛИМН с текстовой маской в качестве одного из условий.
2.4 Рибосомальные РНК и белки
Названия рибосомальных РНК и белков были получены посредством применения к исходным данным фильтрации по текстовому фильтру. Для рибосомальных РНК использовался фильтр по полю class со значением rRNA, для рибосомальных белков – фильтр по полю name: содержит ribosomal protein, не содержит ase (для удаления белков типа 50S ribosomal protein L11 methyltransferase) и ручной проверкой для удаления белков типа ABC-F type ribosomal protection protein.
2.5 Транспортные РНК
class со значением tRNA. Количество тРНК для каждой из аминокислот подсчитывалось функцией СЧЁТЕСЛИ. Значения долей аминокислот в белках были получены из данных о последовательностях белков банка Uniplot [], посчитаны с использованием скрипта, написанного на Python.
2.6 Гистограмма длин белков
Для построения гистограммы длин белков применялась функция СЧЁТЕСЛИМН с условием на число относительно конкретной величины, функции МАКС и МИН для определения границ наблюдений, функция ТЕКСТ и операции с текстом, такие как &, для автоматической генерации подписей «карманов» гистограммы по имеющемуся столбцу граничных значений.
Ширина «карманов» диаграммы выбиралась такой, чтобы огибающая была сравнительно гладкой.
2.7 Гистограмма межгенных промежутков
Помимо описанного в пункте 2.4, для нахождения расстояния до предыдущего гена на той же цепи применялся следующий алгоритм:
-
Формирование массива значений посредством применения ЕСЛИ к диапазону данных от начала до данной ячейки (возвращает номер строки при помощи функции СТРОКА, если цепь совпадает, 0 иначе), для работы функций массива необходим выход из ячейки не Enter, а Ctrl+Shift+Enter
-
Применение к полученному массиву функции МАКС, возвращающей номер строки предыдущего гена на той же цепи
-
Использование функции ДВССЫЛ, преобразующей текст в формулу, и результата предыдущего этапа для адресации к предыдущему гену и нахождения расстояния от него.
Отрицательные значения промежутков соответствуют перекрывающимся генам.
2.8 Кластеры и квазиопероны
Для нахождения кластеров использовались данные о промежутках из пункта 2.6. Кластером считалась группа последовательных генов на одной цепи с промежутками меньше фиксированного. Квазиопероны определялись схожим обрезом, но с необходимым условием: эти последовательные гены должны все быть белоккодирующими.
Для удобства восприятия использовалось условное форматирование: ячейки с названиями генов, относящихся к одному кластеру/квазиоперону залиты одним цветом, цвета чередуются.
2.9 Случайность распределения белков
Проверялась гипотеза о случайном распределении генов с равной вероятностью по цепям и по рамкам считывания.
2.9.1 По цепям
Использовались данные пункта 2.2, для определения значимости рассчитывалось p-значение (p-value) с использование функции БИНОМ.РАСП. Эта функция выдаёт вероятность P того, что из n испытаний с фиксированной вероятностью успеха p будет наблюдаться k или (если параметр «интегральная» = ИСТИНА) больше успехов по формуле
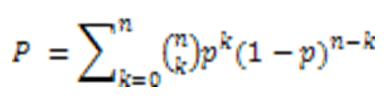
В данном случае «успех»–попадание на цепь, содержащую меньшее количество генов. Поскольку для нас одинаково значимы ситуации, когда минимум генов наблюдается на + и на – цепи, в случае неравенства этих количеств, результат функции умножается на два (т.е. складывается с точно таким же результатом для другой цепи).
2.9.2 По рамкам считывания
Применялись функции СЧЁТЕСЛИМН и ОСТАТ для получения данных о количествах белков в шести рамках считывания (по три на каждой цепи), уровень значимости определялся при помощи функции ХИ2.ТЕСТ. Данная функция рассчитывает вероятность того, что для независимо распределённых по r*c (r строй, c столбцов) классам величин A c ожидаемыми значениями E значение критерия χ2, вычисляемого по формуле
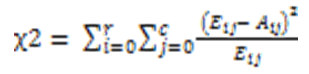
будет не меньше полученного значения критерия для имеющихся данных. Вероятность вычисляется с использованием количества степеней свободы распределения данных, равному (r-1)(c-1), если r>1 и c>1; c-1, если r=1 и c>1; r-1, если c=1 и r>1; при r=1 и с=1 распределение лишено смысла. Таким образом, значения, выдаваемые функцией для несоответствующим ожидаемым данных близки к 0, а для соответствующих ожидаемым – к 1. В качестве ожидаемого в данном случае используется равномерное распределение (среднее арифметическое в каждом классе).
3. Результаты и обсуждение
В хромосомной таблице представлены хромосома, содержащая 2422 гена и плазмида CP026504.1, содержащая 19 генов.
3.1 Статистика
3.1.1 По цепям
В таблице ниже приведены данные о распределении генов различных типов по + и – цепи, а p-значение для гипотезы о распределении с равной вероятностью. Распределение по всей хромосоме в сумме и в плазмиде также графически представлено на рис. 1.
| хромосома | всего | + цепь | - цепь | p-value | случайно |
|---|---|---|---|---|---|
| транслируемые | 2176 | 1075 | 1101 | 0,59 | да |
| псевдогены | 157 | 84 | 73 | 0,42 | да |
| всего (белки) | 2333 | 1159 | 1174 | 0,77 | да |
| тРНК | 71 | 49 | 22 | 0,00 | нет |
| рРНК | 15 | 12 | 3 | 0,04 | нет |
| тмРНК | 1 | 0 | 1 | 1,00 | да |
| некод. РНК | 2 | 1 | 1 | 0,75 | да |
| всего РНК | 89 | 62 | 27 | 0,00 | нет |
| всего | 2422 | 1221 | 1201 | 0,70 | да |
Как можно видеть, неслучайным является распределение тРНК и рРНК, они преобладают на + цепи. Объяснение этого факта обсуждается в разделе 3.5.
| плазмида | всего | + цепь | - цепь | p-value | случайно |
|---|---|---|---|---|---|
| транслируемые | 14 | 4 | 10 | 0,18 | да |
| псевдогены | 5 | 1 | 4 | 0,38 | да |
| всего (белки) | 19 | 5 | 14 | 0,06 | да |
| всего РНК | 0 | 0 | 0 |
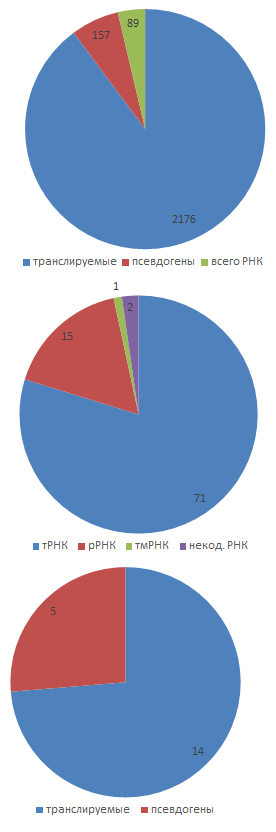
В плазмиде не содержится генов РНК, распределение по цепям можно считать находящимся в пределах случайного. Данная плазмида, вероятно, транспозонного происхождения, она несёт несколько генов транспозаз, резольвазу (сайт-специфическую рекомбиназу, свойственную транспозонам) [5], а также гены, необходимые для собственной репликации. Единственный ген плазмиды, который может быть полезен для бактерии – filamentation induced by cAMP protein fic. Fic-домен известен во многих бактериальных и даже человеческих белках, участвующих в посттрансляционной модификации. Цели этой модификации нередко представляют собой часть системы токсин-антинтоксин или же просто секретируемые токсины [6].
3.1.2 По рамкам считывания
Распределение белков по рамкам считывания определялось только для хромосомы, поскольку в плазмиде белков слишком мало для достоверного расчёта.
| цепь | + | - | ||||
|---|---|---|---|---|---|---|
| рамка | 1 | 2 | 0 | 1 | 2 | 0 |
| число | 425,00 | 394,00 | 402,00 | 429,00 | 371,00 | 401,00 |
| ожидаемое | 407,00 | 407,00 | 407,00 | 400,33 | 400,33 | 400,33 |
| хи2 тест | 0,53 | 0,12 | ||||
| ожидаемое | 403,67 | 403,67 | 403,67 | 403,67 | 403,67 | 403,67 |
| хи2 тест | 0,35 | |||||
Таким образом, можно утверждать, что распределение генов по рамкам считывания независимое.
3.2 Гипотетические белки
Гипотетические белки составляют 472 из 2176 белок-кодирующего гена, т.е. 21,7%. Эта достаточно большая доля свидетельствует о недостаточной изученности данного генома.
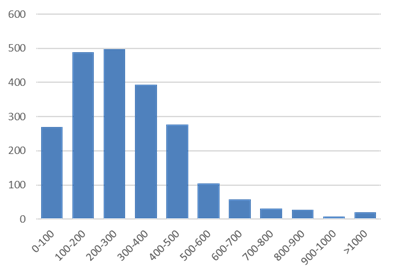
3.3 Гистограмма белков по длинам
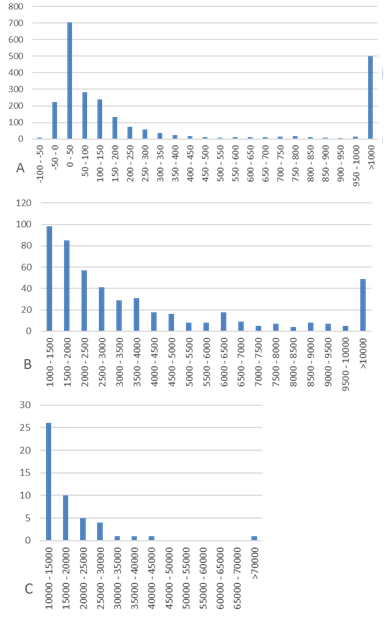
Гистограмма приведена на рис.2
В отличие от типичного для бактерий случая, на диаграмме не наблюдается один пик вместо двух, соответствующих белкам одно- и двудоменной структуры. Это может свидетельствовать о том, что у L.crispatus доля двудоменных белков мала.
3.4 Межгенные промежутки, кластеры, опероны
3.4.1 Гистограмма межгенных промежутков
Основная часть гистограммы (до промежутка в 1000 п.н.) представлена на рис. 3 (разбита на участки с разным масштабом).
Подобный вид гистограммы показывает, что оперонная структура у L.crispatus плохо выражена. В противном случае следовало бы ожидать двух пиков: характерное расстояние между генами внутри оперона и между двумя оперонами. Поскольку регуляторные участки расположены в основном между оперонами, соответствующее расстояние должно значительно превосходить внутриоперонное.
Поскольку полученные результаты не позволяют найти характерное расстояние между генами внутри оперона для L.crispatus, в дальнейшем (при поиске предположительных оперонов) используется полученная для других бактерий граница в 100 п.н.
3.4.2 Кластеры
При границе в 100 п.н. в хромосоме выделяется 1200 кластеров. Несколько наиболее крупных, но не являющихся при этом оперонами, представлены в таблице ниже.
Псевдогены встречаются поодиночке и не собираются в кластеры.
3.4.3 Квазиопероны
Несмотря на обсуждавшуюся выше проблему непредставленности квазиоперонов на гистограмме межгенных промежутков, была предпринята попытка их выделить и проанализировать. При границе в 100 п.н. в хромосоме выделяется 1110 квазиоперонов (ненамного меньше, чем кластеров, поскольку «разбивающие» кластеры псевдогены и гены РНК составляют лишь небольшую часть (10%) генома). Ниже приведены несколько наиболее крупных из тех, чьи гены, если судить по названию, имеют сходную функцию и участвуют в одном процессе.
| Номер | Количество генов | Состав/функция |
|---|---|---|
| 720 | 38 | Фаг (только часть квазиоперона) |
| 163 | 29 | Рибосомные белки |
| 484 | 27 | Фаг |
| 599 | 19 | Фаг (только часть квазиоперона |
| 677 | 14 | Клеточное деление (только часть квазиоперона) |
| 690 | 13 | АТФ-синтаза |
| 676 | 11 | Гены, работающие с аминок-тами и тРНК |
| 620 | 8 | Трансляция |
| 420 | 7 | Fe-S кластер |
| 443 | 7 | Цитратлиаза (только часть квазиоперона) |
| 520 | 7 | Система рестрикции-модификации |
Два наиболее крупных квазиоперона (№786 (38 генов); №658 (33 гена)) содержат исключительно гипотетические белки. Квазиопероны 720, 484, 599 содержат гипотетические и вирусоподобные белки и предположительно являются интегрировавшимся в геном бактериофагами.
Далеко не для всех крупных квазиоперонов удаётся предположить функцию, часто входящие в них гены совершенно различны и находятся на столь небольшом расстоянии просто по случайности.
Несомненно, среди найденных 1200 присутствует ещё множество достоверных (т.е. содержащие схожие по функции гены) квазиоперонов меньшего размера, но их поиск затруднён большим количеством недостоверных случайных сближений, ведь, чем меньше количество генов в квазиопероне, тем больше вероятность получить похожее положение случайно для несвязанных друг с другом генов. При этом уменьшение границы значительно увеличивает число квазиоперонов, затрудняя поиск.
При этом многие очевидные единства, где подряд на расстоянии чуть большем 100 идут, например, субъединицы одного комплекса, данным методом отнесены к нескольким последовательным группам. Однако повышение границы кажется нецелесообразным, поскольку оно значительно повышает количество случайных сближений несвязанных генов.
Таким образом, данный метод выдаёт «сырые данные», с которыми можно работать дальше, анализируя на более глубоком уровне.
3.4.4 Перекрытия
Рассматривались перекрывания только с предыдущим геном. Результаты представлены в таблице, число – общее число перекрытий, доля – доля перекрытий от общего числа промежутков в данной категории:
| Перекрытий | Всего | На + цепи | На - цепи |
|---|---|---|---|
| Число | 326 | 161 | 165 |
| Доля | 13,46% | 13,19% | 13,74% |
Как можно видеть, процент перекрывающихся генов очень близок на обеих цепях. Среди перекрывающихся генов встречаются гены с совершенно разными функциями.
Если посмотреть на долю перекрывающихся генов в некторых квазиоперонах и кластерах (рассматривались лишь категории, содержащие хотя бы 10 генов), наблюдается следующее:
| Категория | Число | Доля |
|---|---|---|
| оперон 720 (фаг) | 15 | 39,47% |
| оперон 484 (фаг) | 12 | 44,44% |
| оперон 599 (фаг) | 10 | 52,63% |
| оперон 481 (гипотетические белки) | 8 | 61,54% |
| оперон 690 (АТФ-синтаза) | 1 | 7,69% |
| оперон 677 (клеточное деление) | 4 | 28,57% |
| оперон (работают с ак и тРНК) | 3 | 27,27% |
| кластер (тРНК) | 0 | 0,00% |
| кластер (тРНК) | 0 | 0,00% |
| все РНК | 3 | 3,37% |
| все гипотетические | 75 | 15,89% |
В квазиоперонах процент перекрытий варьируется довольно чильно, что неудивительно, учитывая, что в небольшой группе велика вероятность выбросов.
В фагах процент перекрытий гораздо выше среднего, что неудивительно, ведь стратегия перекрывания генов часто используется вирусами для уменьшения размеров генома. Столь высокий процент перекрытий не может быть объяснён высокой долей гипотетических белков в этих оперонах, как можно подумать, глядя на процент перекрываний в 481 опероне, поскольку для всех гипотетических белков в среднем он значительно ниже. Возможно, это указывает, что 481 оперон также имеет вирусное происхождение.
Интересно, что в кластерах, кодирующих тРНК, перекрытия не встречаются вовсе. Это верно не только в пределах этого кластера: из 72 генов тРНК лишь один перекрывается с предыдущим, и то тот ген лишь гипотетический, в случае рРНК перекрываются 2 из 15 генов, оба с гипотетическими белками. Возможно, это значит, что гипотетические белки не являются реальными или на самом деле заканчиваются раньше. Причиной подобного свойства генов некодирующей РНК может быть тот факт,
3.5 Рибосомальные РНК и белки
В геноме сдержится 15 генов рРНК: по пять генов 5S, 16S и 23S рРНК (ribosomal RNA). По расположению они концентрируются в три основные группы: кластеры 42-45, кластеры 241-246 (на + цепи) и кластер 960 (на - цепи). Помимо рРНК эти кластеры содержат гены тРНК, а кластер 245 содержит белки, необходимые для функционирования рибосомы: РНКазу, участвующую в созревании рРНК, транслоказу (вероятно, участвующую в переносе ионов Mg2+, необходимых для работы рибосомы), несколько РНК-связывающих белков и гликозил-трансфераз неизвестной функции. Небольшое количество кластеров, в которые собраны рРНК и приводит к неравномерности распределения этих генов по цепям, отмеченному в пункте 3.1.1.
Внутри тРНК-рРНК кластеров рРНК встречаются в виде двух подкластеров с одинаковым составом и одинаковым порядком генов: 16S rRNA, hypothetical protein, 23S rRNA, 5S rRNA или 16S rRNA, tRNA-Ile, tRNA-Ala, 23S rRNA, 5S rRNA. Вероятно, наблюдаемое положение рРНК в геноме получилось в результате дупликации одного предкового кластера с произошедшей в процессе мутацией, результат которой впоследствии также дуплицировался.
В геноме содержится 56 рибосомальных белков: 20 белков малой субъединицы (30S ribosomal protein S1 – S21 кроме S14 и два type Z 30S ribosomal protein S14.), 33 белка большой субъединицы (50S ribosomal protein L1 - L36, кроме L8, L25, L26, L31 но с L7/12, type B 50S ribosomal protein L31 и двумя L33).
Примерно половина (27 из 56) из них располагаются в одном квазиопероне (№163), остальные (29 из 56) по одному-два распределены по разным местам генома.
3.6 Транспортные РНК
Геном в геноме содержатся 71 ген тРНК. Количество тРНК и доля относительно всех тРНК для каждой из аминокислот показано в табл. 8 (также см. диаграмму рис.4), также показана доля соответствующей аминокислоты во всех белках.
| Аминокислота | Количество тРНК | Доля тРНК | Доля в белках |
|---|---|---|---|
| Ala | 2 | 2,8% | 7,4% |
| Arg | 5 | 7,0% | 3,9% |
| Asn | 4 | 5,6% | 5,5% |
| Asp | 4 | 5,6% | 6,0% |
| Cys | 1 | 1,4% | 0,6% |
| Gln | 5 | 7,0% | 4,1% |
| Glu | 3 | 4,2% | 5,6% |
| Gly | 6 | 8,5% | 6,4% |
| His | 2 | 2,8% | 2,1% |
| Ile | 3 | 4,2% | 7,5% |
| Leu | 5 | 7,0% | 9,4% |
| Lys | 3 | 4,2% | 7,9% |
| Met | 4 | 5,6% | 2,7% |
| Phe | 2 | 2,8% | 4,4% |
| Pro | 3 | 4,2% | 3,5% |
| Ser | 6 | 8,5% | 5,7% |
| Thr | 4 | 5,6% | 5,6% |
| Trp | 3 | 4,2% | 1,1% |
| Tyr | 3 | 4,2% | 3,9% |
| Val | 3 | 4,2% | 6,8% |
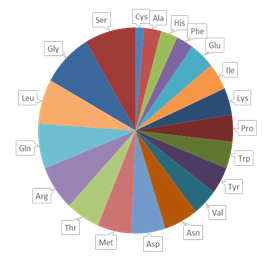
На диаграмме рис.7 показано соотношение долей тРНК и аминокислоты в белках для каждой из аминокислот:
Примерно в половине случаев доли схожи, в оставшихся наблюдаются отклонения. Значительно отличаются доли для аланина, аргинина, глутамина, изолейцина, лизина, метионина, серина, триптофана, валина. В случае метионина это можно объяснить его ролью при трансляции: метионин кодируется старт-кодоном, поэтому его тРНК необходимо много, но при созревании белка часто вырезается.
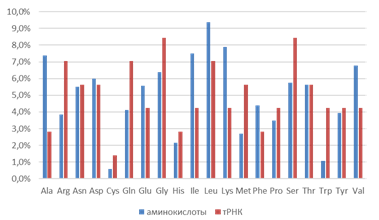
Также это может быть следствием того, что каждый белок в рассмотренных данных представлен один раз, соответственно, эти доли не отражают реальные доли аминокислот, необходимых бактерии.
В геноме тРНК распределены следующим образом:
| Число | Доля | Положение |
|---|---|---|
| 42 | 59% | В кластере с рРНК |
| 9 | 13% | В собственном кластере или с нерибосомальными генами |
| 20 | 28% | Одиночные |
В отличие от рРНК, никакой закономерности в расположении, которая могла бы указывать на дупликацию части изначального кластера, выделить не удаётся.
4. Заключение
В ходе данной работы были выявлены следующие особенности распределения генов:
- рРНК образуют три крупных кластера, содержащих также тРНК и некоторые связанные с трансляцией белки.
- Гены РНК никогда не перекрываются с соседними.
- Найденные квазиопероны вирусного происхождения отличаются от остального генома по частоте перекрытий генов.
Остальные результаты нуждаются в дальнейшем анализе.
5. Сопроводительные материалы
Скрипт для подсчёта долей аминокислот в белках
6. Источники
[1] The National Center for Biotechnology Information (NCBI) Organism overview Lactibacillus crispatus (просмотрено 15.12.2019)
[2] May A. D. Antonio and Sharon L. Hillier, DNA Fingerprinting of Lactobacillus crispatus Strain CTV-05 by Repetitive Element Sequence-Based PCR Analysis in a Pilot Study of Vaginal Colonization. J Clin Microbiol. 2003 May; 41(5)
[3] Ojala T, Kuparinen V, Koskinen JP, et al (2010) Genome sequence of Lactobacillus crispatus ST1, J Bacteriol. 2010 Jul;192(13):3547-8.
[4] База данных Uniplot, страница протеома (просмотрено 15.12.2019)
[5] В.З. Тарантул, Толковый биотехнологический словарь. (2009)
[6] Veyron S, Peyroche G, Cherfils J (2018) FIC proteins: from bacteria to humans and back again. Pathog Dis. 2018 Mar 1;76(2).