Алгоритм BLAST, матрицы BLOSUM, карты локального сходства
Отличия между двумя выравниваниями
Были взяты множественное выравнивание (fasta-файл) гомологов белков с мнемоникой QUEC из 10 практикума, сделанное программой muscle, из которых удалили все последовательности, кроме первой и последней (fasta-файл), и оптимальное глобальное выравнивание этих двух последовательностей, найденное программой needle (fasta-файл). Некоторые наблюдаемые различия (мнемоника функции из названия последовательности опущена, оставлена только мнемоника организма):
- Muscle: ECOLI-H39 сопоставлен METMA-A38; needle: ECOLI-H39 сопоставлен METMA-G39

По матрице Blosum62 оба эти сопоставления имеют одинаковую цену -2. Но вариант сопоставления muscle представляется более убедительным, поскольку ECOLI-H39 является концом консервативного блока (столбцы 40-45), и вставка в METMA нуклеотида G39 после этого блока более вероятна, чем вставка A38 внутрь блока.
- Muscle: ECOLI-V81 сопоставлен METMA-V80; needle: ECOLI-V81 сопоставлен METMA-S82.

Muscle даёт более убедительный результат. На самом деле это не точечное различие, а сдвиг сразу шести нуклеотидов (DSIPVP, выделены красным): из области, присутствующей у всех последовательностей множественного выравнивания в область, где у большинства из них наблюдается индель, и гомология конкретных двух последовательносей в этом участк менее вероятна, чем их гомология на участке, общем для всех последовательностей. Хотя непосредственно для валина сопоставление с валином (4) лучше, чем с серином (-2), в сумме оба варианта расположения этого шестиаминокислотного фрагмента весят одинаково (7 + штраф за индель длины 2), поэтому для программы needle они равнозначны.
- Muscle: ECOLI-G196 сопоставлен METMA-I201; needle: ECOLI-G196 сопоставлен METMA-P202.

Множественное выравнивание здесь не даёт однозначного преимущества, поскольку любая из вставок разрывает консервативный блок (столбцы 234-250) - или же, если начинать этот блок с 238 (где консервативность полная), то любой вариант вставки не влияет. По значению в матрице сопоставление G-I (-4) хуже, чем G-P (-2), таким образом, здесь вариант из needle предпочтительнее.
Множественное выравнивание в среднем лучше парного, поскольку учитывает при выборе другие последовательности, а значит, с меньшей вероятностью разрывает гэпами консервативные (соответственно, вероятно, функциональные) блоки, если этого можно избежать.
Карта локального сходства
При помощи алгоритма BLAST были выровнены последовательности двух белков: Cystic fibrosis transmembrane conductance regulator (ID CFTR_DANRE; AC Q1LX78) и ATP-binding cassette sub-family B member 6, mitochondrial (ID ABCB6_HUMAN ; AC Q9NP58).
Получилась следующая диаграмма выравнивания:
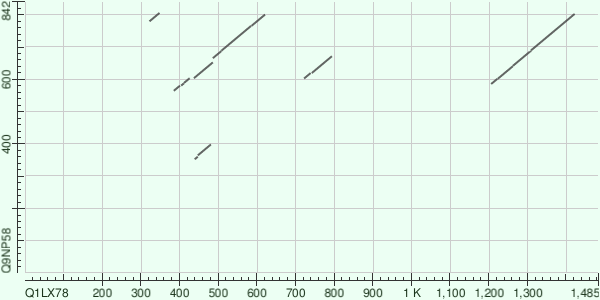
На ней можно видеть, что большая часть обоих белков не имеет схожих участков, однако один длинный фрагмент в Q9NP58 соответствует двум фрагментам Q1LX78 одновременно. Также присутствует несколько меньших по размерам участков сходства (все они дублируют какую-то часть одного из крупных участков на одном из белков).
Лучшее локальное выравнивание из найденных:
Query 565 FDLLKEETEVKDLPGA---GPLRFQKGRIEFENVHFSYADGR-----------ETLQDVS 610
+DL E E++D+ + GP + RI+ EN + +G L+D+S
Sbjct 386 YDLSITELELQDVTASWDEGPGELLE-RIKQENKANGHHNGDAGLFFTNLYVAPVLKDIS 444
Query 611 FTVMPGQTLALVGPSGAGKSTILRLLFRFYDISSGCIRIDGQDISQVTQASLRSHIGVVP 670
+ G+ LA+ G G+GKS++L + SSG IR G+ I
Sbjct 445 LKLKKGEMLAVTGSMGSGKSSLLMTILGELVPSSGKIRHSGR-------------ISYSS 491
Query 671 QDTVLFNDTIADNIRYGRVTAGNDEVEAAAQAAGIHDAIMAFPEGYRTQVGERGLKLSGG 730
Q + TI DNI +G +T ++ +A + + + A PE +T + E GL LSGG
Sbjct 492 QTAWIMPGTIRDNILFG-LTYDEYRYKSVVKACQLEEDLAALPEKDKTPMAEGGLNLSGG 550
Query 731 EKQRVAIARTILKAPGIILLDEATSALDTSNERAI-QASLAKVCANRTTIVVAHRLSTVV 789
+K RVA+AR + + + LLD + LD + E+ I L K+ A++T I+V +++ +
Sbjct 551 QKARVALARAVYRDADLYLLDAPFTHLDIATEKEIFDKCLCKLMASKTRILVTNKIEHLK 610
Query 790 NADQILVIKDG 800
AD+IL++ +G
Sbjct 611 RADKILLLHNG 621
С выравниванием, выданным для этих последовательностей прграммой water оно даже не пересекается (по первой последовательности, т.е. query, CFTR_DANRE):
CFTR_DANRE 801 LDIYTRRLSDSTYDMTGILEEENIEACLTDEIDEIEETFETTKWNTYVR- 849
|.::...|...:|.:....|::::|. .::....:.:.|..:.|
ABCB6_HUMAN 202 LGLWAPGLRPQSYTLQVHEEDQDVER------SQVRSAAQQSTWRDFGRK 245
CFTR_DANRE 850 --------YVSNNKSLLYVLIFILFIAAIEIAGSV-AGIF------LITD 884
:...:.:|..|::..|.:..:|.|.:| ..|| |:|:
ABCB6_HUMAN 246 LRLLSGYLWPRGSPALQLVVLICLGLMGLERALNVLVPIFYRNIVNLLTE 295
CFTR_DANRE 885 EL-WREEHQRSEPNMTKHSNASSSGQTYAITVTPTSSYYILYI------- 926
:. |. ::..|.||..::.::
ABCB6_HUMAN 296 KAPWN-----------------------SLAWTVTSYVFLKFLQGGGTGS 322
CFTR_DANRE 927 --YVATSESLLAMGFFRGLPFV--HTTITISKKLHQKMLHAVLSAPMSVL 972
:|:...:.| :.|...|. ...:.|...||:..|...|.
ABCB6_HUMAN 323 TGFVSNLRTFL---WIRVQQFTSRRVELLIFSHLHELSLRWHLG------ 363
CFTR_DANRE 973 NTMKTGRIMNRFTKDMATIDDMLPLLMFDFV-QLTVVVVGCILVVSIVRP 1021
.:||.::....:..:::..:|..|:|:.: .|..:::|.|........
ABCB6_HUMAN 364 --RRTGEVLRIADRGTSSVTGLLSYLVFNVIPTLADIIIGIIYFSMFFNA 411
CFTR_DANRE 1022 YIFLAATPLAIIFIVMRKYF------------LRTGQQLKQLETEARSPI 1059
:..| |:|:.|..|. .|.....::..|.||:
ABCB6_HUMAN 412 WFGL------IVFLCMSLYLTLTIVVTEWRTKFRRAMNTQENATRARA-- 453
CFTR_DANRE 1060 FSHLIMSLKGLWTIR-----AFERQAYFEALF-HKTLNTHTATWFLYLS- 1102
:.||....|:: ::|.:.|.||:. ::.|...::...:.|:
ABCB6_HUMAN 454 ----VDSLLNFETVKYYNAESYEVERYREAIIKYQGLEWKSSASLVLLNQ 499
CFTR_DANRE 1103 ----TLRWFLFRADILFVFFFT-----LAAWIAVGTNQDKPGEIGIIICL 1143
.:...|....:|..:|.| :..::..|| .||.|
ABCB6_HUMAN 500 TQNLVIGLGLLAGSLLCAYFVTEQKLQVGDYVLFGT---------YIIQL 540
CFTR_DANRE 1144 AMLI--LGTFQWCVATS-IAVDGMMRSVDRVFKFIDLPSETPKPDKGKDS 1190
.|.: .||:...:.|: |.::.|...:....:..|||...|...:
ABCB6_HUMAN 541 YMPLNWFGTYYRMIQTNFIDMENMFDLLKEETEVKDLPGAGPLRFQ---- 586
CFTR_DANRE 1191 DLIIENVDAQADSSWPHRGQIEVRNLTVKYTEAGHAVLKNLSFSAEGRQR 1240
:|:||..|:...|.: |...|:::||:....|.
ABCB6_HUMAN 587 -----------------KGRIEFENVHFSYAD-GRETLQDVSFTVMPGQT 618
CFTR_DANRE 1241 VGILGRTGSGKSSLFNALLKLV-YTDGEISIDGVNWNKMPLQKWRKAFGV 1289
:.::|.:|:|||::...|.:.. .:.|.|.|||.:.:::.....|...||
ABCB6_HUMAN 619 LALVGPSGAGKSTILRLLFRFYDISSGCIRIDGQDISQVTQASLRSHIGV 668
CFTR_DANRE 1290 VPQKVFIFTGPLRMNLDPYG--CHSDEELWRVAEEVGLKTVIEQFPDKLD 1337
|||...:|...:..|: .|| ...::|:...|:..|:...|..||:...
ABCB6_HUMAN 669 VPQDTVLFNDTIADNI-RYGRVTAGNDEVEAAAQAAGIHDAIMAFPEGYR 717
CFTR_DANRE 1338 FQLEYGGYVLSNGHKQLICLARSILSGARILLLDEPSAHLDPVTIKVLKK 1387
.|:...|..||.|.||.:.:||:||....|:||||.::.||....:.::.
ABCB6_HUMAN 718 TQVGERGLKLSGGEKQRVAIARTILKAPGIILLDEATSALDTSNERAIQA 767
CFTR_DANRE 1388 TLRQSFSTCTILLSEHKVEPLLECQSFLMMDKG 1420
:|.:..:..|.::..|::..::.....|::..|
ABCB6_HUMAN 768 SLAKVCANRTTIVVAHRLSTVVNADQILVIKDG 800
Однако частью этого выравнивания является второе по весу (не сильно отличающееся: 95.5 bits, в то время как лучшее найденное 99.8 bits):
Query 1207 HRGQIEVRNLTVKYTEAGHAVLKNLSFSAEGRQRVGILGRTGSGKSSLFNALLKLV-YTD 1265
+G+IE N+ Y + G L+++SF+ Q + ++G +G+GKS++ L + +
Sbjct 586 QKGRIEFENVHFSYAD-GRETLQDVSFTVMPGQTLALVGPSGAGKSTILRLLFRFYDISS 644
Query 1266 GEISIDGVNWNKMPLQKWRKAFGVVPQKVFIFTGPLRMNLDPYG--CHSDEELWRVAEEV 1323
G I IDG + +++ R GVVPQ +F + N+ YG ++E+ A+
Sbjct 645 GCIRIDGQDISQVTQASLRSHIGVVPQDTVLFNDTIADNIR-YGRVTAGNDEVEAAAQAA 703
Query 1324 GLKTVIEQFPDKLDFQLEYGGYVLSNGHKQLICLARSILSGARILLLDEPSAHLDPVTIK 1383
G+ I FP+ Q+ G LS G KQ + +AR+IL I+LLDE ++ LD +
Sbjct 704 GIHDAIMAFPEGYRTQVGERGLKLSGGEKQRVAIARTILKAPGIILLDEATSALDTSNER 763
Query 1384 VLKKTLRQSFSTCTILLSEHKVEPLLECQSFLMMDKGQV 1422
++ +L + + T ++ H++ ++ L++ G +
Sbjct 764 AIQASLAKVCANRTTIVVAHRLSTVVNADQILVIKDGCI 802
В этих выравниваниях есть несколько отличий. Так, Q586 ABCB6_HUMAN(Sbjct) соответствует H1207 CFTR_DANRE(Query) в выравнивании BLAST и K1186 в выравнивании water, D1306 CFTR_DANRE соответствует R685 ABCB6_HUMAN в выравнивании BLAST и ничему в выравнивании water.
Этот фрагмент (CFTR_DANRE 1207-1422, ABCB6_HUMAN 586-802) в обоих белках примерно совпадает с размеченным доменом ABC transporter (позиции: CFTR_DANRE 1211-1444 и ABCB6_HUMAN 590-824). Причём лучшее из BLAST'вских выравниваний (CFTR_DANRE 386-621 и ABCB6_HUMAN 565-800) представляет этот же домен (позиция CFTR_DANRE 424-645). Если нанести размеченные домены на диаграмму выравнивания, можно видеть яснее, что домен ABC transporter и является тем фрагментом, присутствующим дважды в CFTR_DANRE и один раз в ABCB6_HUMAN, который был виден на карте локального сходства. Меньшие участки, заметные на ней, скорее всего выровнялись по случайным причинам и эволюционного смысла это выравнивание не несёт.
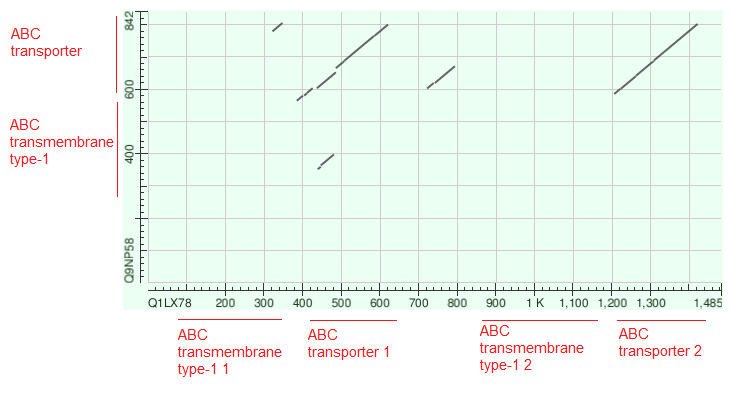
Для выравнивания CFTR_DANRE 440-481, ABCB6_HUMAN 353-398 это дополнительно подтверждается следующим соображением: раз этот фрагмент на CFTR_DANRE является частью размеченного домена, было бы логичным видеть его, и, соответственно, локальное выравнивание его с ABCB6_HUMAN 353-398, и во втором таком же домене, однако, этого не происходит.
Выравнивание CFTR_DANRE 323-348, ABCB6_HUMAN 780-850 просто покрывает очень короткий участок, и вероятность случайного совпадения высока.
Про выравнивание CFTR_DANRE 723-794, ABCB6_HUMAN 603-671 непонятно. С одной стороны, сравнение того, какие именно аминокислоты выровнялись с соответствующим фрагментам, показывает, что совпадения, если они есть, сравнительно часто именно по консервативным (т.е. общим и для двух других выравниваний)позициям - если бы сходство было случайным, совпадения обнаруживались бы одинаково часто для консервативных и неконсервативных позиций.

С другой стороны, в полях FT для CFTR_DANRE этот участок попадает внутрь TOPO_DOM "Cytoplasmic" и REGION "Intrinsically disordered R region" (то есть участок без выраженой вторичной структуры, и в соответствующих полях для него она, действительно, не указана). Однако соответствующий участок ABCB6_HUMAN 603-671, согласно полям FT, вторичную структуру имеет.
Ещё кажется странным, что выравнивание не показывает гомологии участков, размеченных как ABC transmembrane type-1, хотя от них можно было бы ожидать сходство.