Alignment & Pfam
Описание домена из Pfam
Для описания был взят один из доменов хеликаз (точная функция непосредственно данного домена ещё не определена).
| ID | Helicase_C |
|---|---|
| AC | PF00271 |
| Всего находок (full) | 191324 |
| Seed | 421 |
| Доменных архитектур | 3120 |
| 3D структур (различных белков) | 95 |
Часто встречающимя в одной архитектуре с данным доменом являются, например:
- DEAD
- SNF2_N
- HA2
Функция - расплетание цепей нуклиновых кислот, белки с этим доменом участвуют в различных процессах, связанных с метаболизмом РНК: транскрипция, сплайсинг, трансляция, образование рибосом, транспорт ядро-цитоплазма, разрушение РНК. Примеры архитектур: X7F901_9RHOB, B7KI22_CYAP7, W7UXI0_RUMFL, A0A1E5L758_9BACL, W9YGA6_9EURO
Встречается в различных белках, вовлечённых в процессы регуляции транскрипции, репарации, рекомбинации, деконденсации хроматина. Примеры архитектур: W4GTV6_9STRA, L0EI52_THECK, Z5XZF8_9GAMM, A0A139AA03_GONPR
Реже, чем предыдущие два. Про данный домен предполагается участие в связывании РНК, он встречается в различных РНК-хеликазах. Примеры архитектур: W9YGA6_9EURO, W7HR86_9PEZI, X5DIP8_9BACT, A0A1M7Y2I5_9DELT
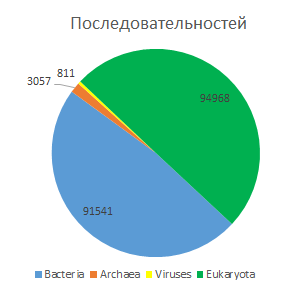
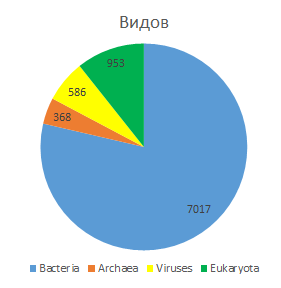
По доменам жизни данный домен распределяется следующим образом:
| sequenses | species | |
| Bacteria | 91541 | 7017 |
| Archaea | 3057 | 368 |
| Viruses | 811 | 586 |
| Eukaryota | 94968 | 953 |
HMM-профиль данного домена создан Sun Aug 5 18:44:25 2018, имеет длину в 111 позиций. Ниже для оживления отчёта показана его визуализация:

Анализ выравнивания из Pfam
Для сравнения последовательностей был выбран порядок архей Thermoplasmatales, содержащий 97 последовательностей из 17 видов
Скачать проект JalviewДалее этот набор был "прорежен" до 13 последовательностей, их можно наблюдать в окне ниже:

В этом выравнивании были найдены следующие примеры блоков:
- Консервативный вертикальный блок
- Консервативный невертикальный блок (не все последовательности)
- Негомологичный вертикальный блок
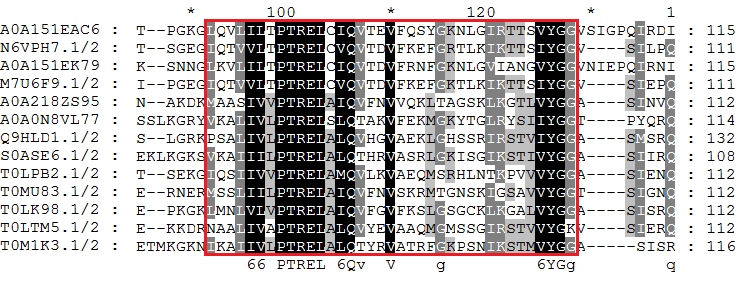

Представлены два варианта раскраски, чтобы показать, где начинается участок, возможно, гомологичный уже у всех последовательностей.

Подобных блоков в выравнивании мало, поскольку в ходе "прореживания" удалялись именно несхожие последовательности. К тому же, часто даже если в целом в блоке последовательности сильно различаются, некоторые последовательности всё равно имеют участки, выглядящие как гомологичные друг другу. Поэтому был взят участок с конца, но одна последовательность оказалась короче. Однако, поскольку мы демонстрируем именно негомологичные участки, наличие гэпов, в отличие от консервативных блоков, не является запрещённым.
Представленность домена в Uniprot
В Uniprot данный домен встречатеся в 636 652 белках (Swissprot - 2 550). Таблицы находок (по отдельным группам, поскольку иначе объём был слишком большой для скачивания из Uniprot): Eukaryota, Archaea, Proteobacteria, Actinobacteria, остальные бактерии, Viruses
Из таблицы можно видеть, что домену Helicase_C в Pfam соответствует в PROSITE домен HELICASE_CTER.
С доменной архитектурой A0A1M7Y2I5_9DELT: DEAD, Helicase_C, HA2, OB_NTP_bind, DUF3418:

в Uniprot находится 7921 белков, Pfam даёт число 1566.
Сначала показалось странным, что, на самом деле, в Uniprot не нашлось ни одного белка с точно такой же архитектурой: все ячейки, содержащие каждый из требуемых доменов имеют порядок PF00270;PF11898;PF04408;PF00271;PF07717, что соотвествтует DEAD, DUF3418, HA2, Helicase_C, OB_NTP_bind. Однако более пристальный взгляд показывает, что в Uniprot cross-references на Pfam всегда упорядочены по алфавиту по id, и их порядок не отражает их реальный порядок в последовательности. А поскольку в полях FT DOMAIN отмечены иные домены, не взаимно однозначно сопоставимые с доменами Pfam, получается, что различить две архитектуры, состоящие из одних и тех же доменов, расположенных в разном порядке при помощи Uniprot не удастся.
И, раз поиск всё равно вёлся отдельно по таксонам, мне показалось интересным включить в отчёт и таблицу с распределением находок по ним (слева). Можно сделать вывод, что данная архитектура характерна для бактерий, причём в двух наиболее крупных круппах (протеобактерии и актинобактерии) встречается заметно чаще, чем в среднем по остальным.
Справа же представлено общее распределение домена по организмам в сравнении с Pfam. Видно, что Pfam сильно отстаёт в добавлении новых записей.
| Таксон | Всего | С архитектурой |
|---|---|---|
| Eukaryota | 196 765 | 4 |
| Proteobacteria | 183 914 | 5 162 |
| Actinobacteria | 67 771 | 2 596 |
| Other bacteria | 165 883 | 159 |
| Archaea | 13 254 | 0 |
| Viruses | 8 122 | 0 |
| Pfam | Uniprot | |
|---|---|---|
| Eukaryota | 94 968 | 196 766 |
| Bacteria | 9 1541 | 417 568 |
| Archaea | 3 057 | 13 284 |
| Viruses | 811 | 8 122 |