Филогенетические деревья-4
1. Составление списка гомологичных белков, включающих паралоги
Поиском blastp по протеомам отобранных бактерий гомологов последовательности CLPX_ECOLI с ограничением на e-value равным 0.001 дал следующий результат:
Score E
Sequences producing significant alignments: (Bits) Value
sp|Q8ZC66|CLPX_YERPE ATP-dependent Clp protease ATP-binding sub... 805 0.0
sp|A4XTZ6|CLPX_PSEMY ATP-dependent Clp protease ATP-binding sub... 658 0.0
sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding sub... 642 0.0
sp|Q1BH84|CLPX_BURCA ATP-dependent Clp protease ATP-binding sub... 621 0.0
sp|Q5P160|CLPX_AROAE ATP-dependent Clp protease ATP-binding sub... 619 0.0
sp|A4SXD7|CLPX_POLAQ ATP-dependent Clp protease ATP-binding sub... 613 0.0
sp|Q92QQ2|CLPX_RHIME ATP-dependent Clp protease ATP-binding sub... 596 0.0
sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding sub... 557 0.0
sp|Q8ZJJ5|HSLU_YERPE ATP-dependent protease ATPase subunit HslU... 95.1 2e-21
sp|Q5P503|HSLU_AROAE ATP-dependent protease ATPase subunit HslU... 93.6 1e-20
sp|A4XPN6|HSLU_PSEMY ATP-dependent protease ATPase subunit HslU... 93.2 1e-20
sp|Q92TA7|HSLU_RHIME ATP-dependent protease ATPase subunit HslU... 92.8 2e-20
tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit Hs... 86.7 2e-18
sp|Q1BSM8|HSLU_BURCA ATP-dependent protease ATPase subunit HslU... 82.4 5e-17
tr|A0A2S9PH39|A0A2S9PH39_YERPE Putative magnesium chelatase fam... 46.2 2e-05
tr|A0A5P8YCE6|A0A5P8YCE6_YERPE Cell division protein OS=Yersini... 45.8 3e-05
tr|Q92M98|Q92M98_RHIME ATP-dependent zinc metalloprotease FtsH ... 45.4 4e-05
tr|A0A0H2XMS5|A0A0H2XMS5_BURCA ATP-dependent zinc metalloprotea... 43.9 1e-04
tr|Q3SJR4|Q3SJR4_THIDA ATP-dependent zinc metalloprotease FtsH ... 43.5 2e-04
tr|A4SXL5|A4SXL5_POLAQ ATP-dependent zinc metalloprotease FtsH ... 43.5 2e-04
sp|Q9JUB0|RUVB_NEIMA Holliday junction ATP-dependent DNA helica... 42.7 2e-04
Полные описания последовательностей можно посмотреть в fasta-файле.
2. Реконструкция и визуализация
Белки были выровнены программой muscle с параметрами по умолчанию на kodomo, по полученному выравниванию построено дерево методом Neighbor-Joining.
Скобочная формула получившегося дерева:
(((((((CLPX_PSEMY,CLPX_THIDA),CLPX_YERPE),(CLPX_POLAQ,(CLPX_BURCA,CLPX_AROAE))),CLPX_RHIME),CLPX_NEIMA),(HSLU_RHIME,((HSLU_YERPE,HSLU_PSEMY),(HSLU_BURCA,(HSLU_AROAE,Q3SFW1_THIDA))))),(RUVB_NEIMA,(A0A5P8YCE6_YERPE,(Q92M98_RHIME,(Q3SJR4_THIDA,(A0A0H2XMS5_BURCA,A4SXL5_POLAQ))))),A0A2S9PH39_YERPE);
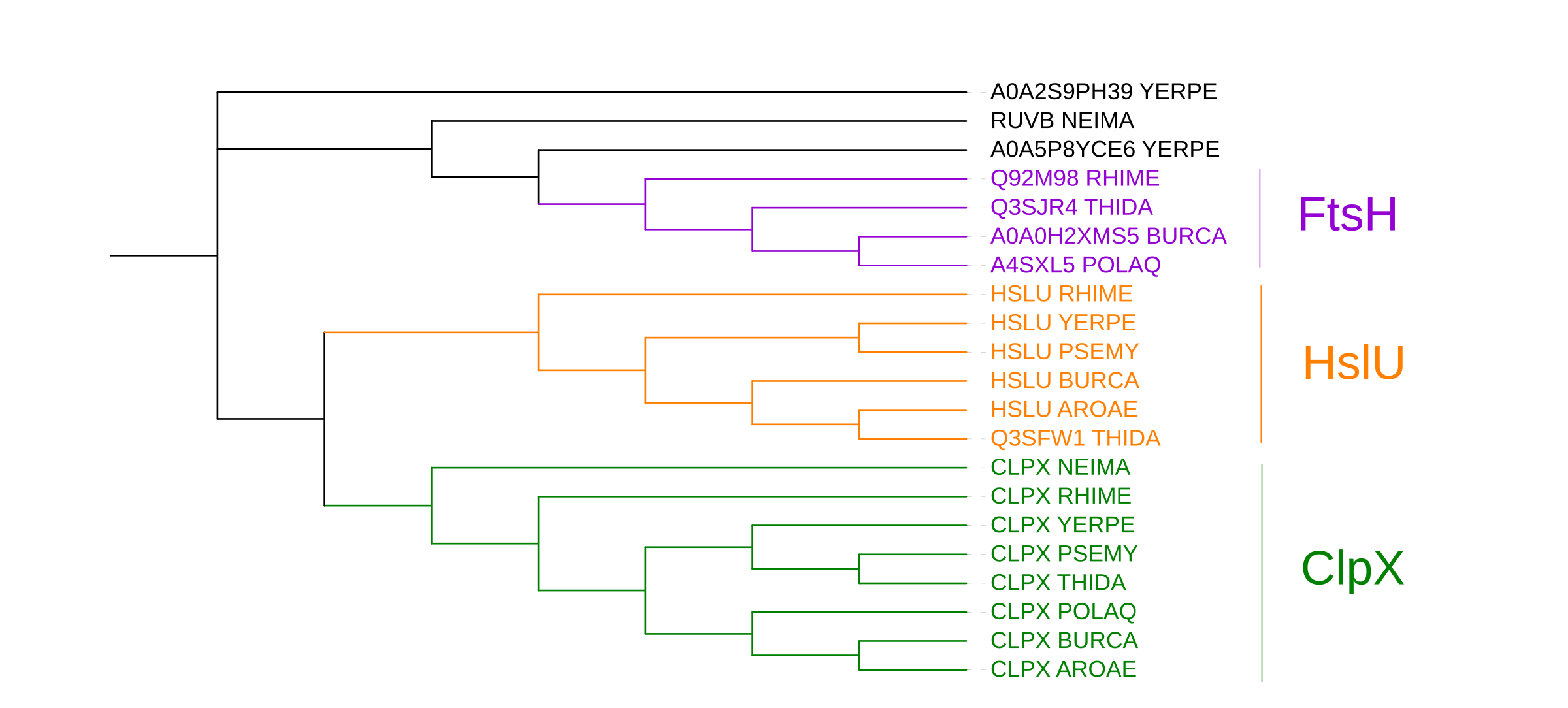
Ортологи: HSLU_AROAE и Q3SFW1_THIDA; CLPX_NEIMA и CLPX_PSEMY; Q92M98_RHIME и Q3SJR4_THIDA
Паралоги: A0A2S9PH39_YERPE и A0A5P8YCE6_YERPE; CLPX_RHIME и HSLU_RHIME; Q3SFW1_THIDA и CLPX_THIDA
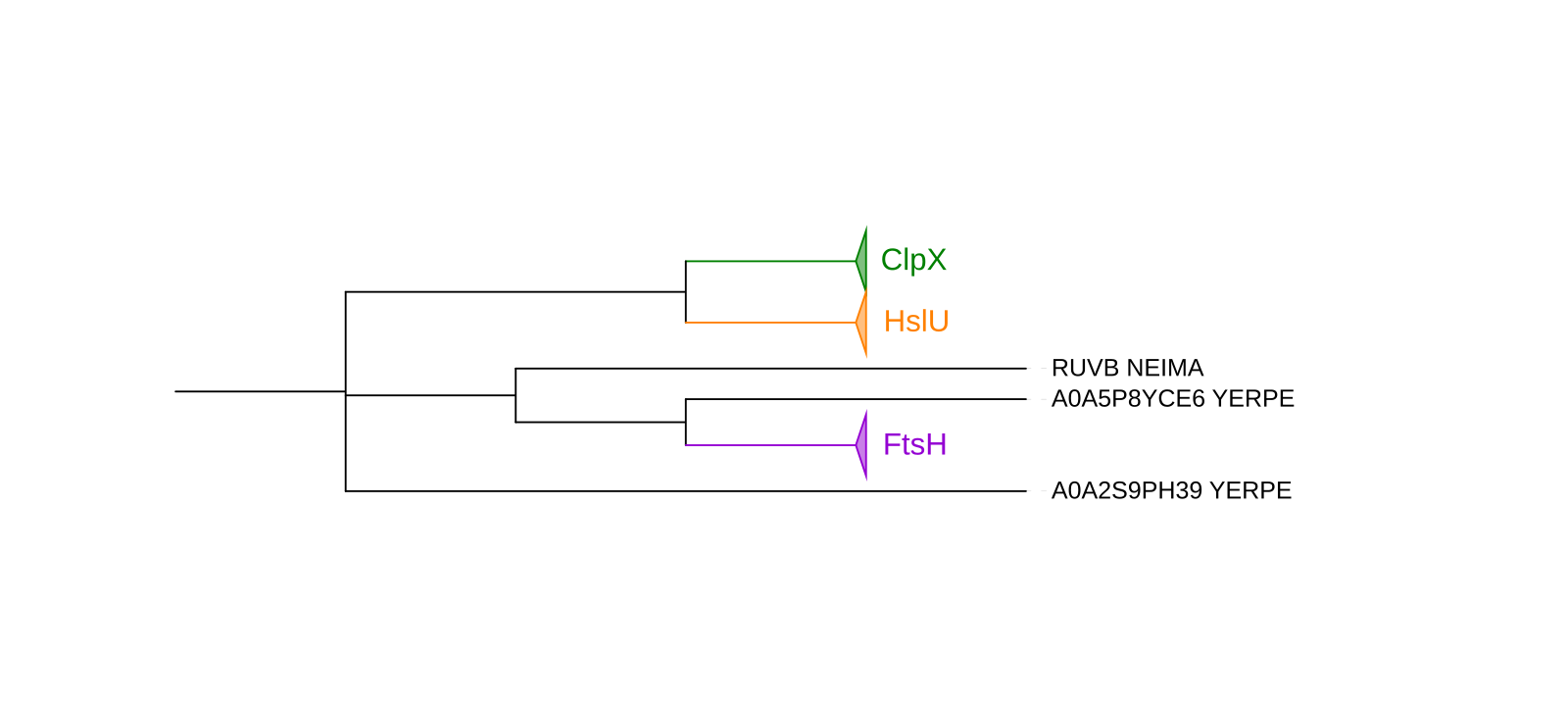
ClpX (ATP-dependent Clp protease ATP-binding subunit ClpX) содержит по белку из всех восьми организмов, филогения не соответствует реальной;
HslU (ATP-dependent protease ATPase subunit HslU) содержит по белку из всех организмов, кроме POLAQ, филогения соответствует реальной;
FtsH (ATP-dependent zinc metalloprotease FtsH) содержит белки только из RHIME, THIDA, BURCA, POLAQ, филогения соответствует реальной.