| Главная страница |
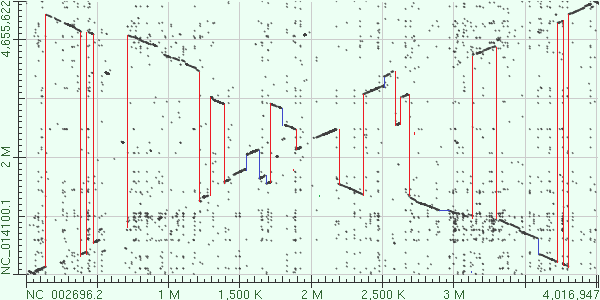
На карте видно, что в геноме происходило очень много инверсий, примерно 1 инверсия на 500 тысяч нуклеотидов, эти места показаны красным. Самая большая длиной около 500 тыс нуклеотидов, она находится примерно с 7000000 по 1200000 нуклеотид (в геноме C. crescentus).
Также имеются места, где произошли вставки или делеции, они отмечены синим: вертикальные линии указывают, что в этом месте последовательность длинее у C. segnis, а горизонтальные - что у C. crescentus. В целом же, геном C. segnis длинее.
У бактерий Caulobacter сложная регуляция клеточного цикла, и очень важно положение гена относительно начала репликации (они должны располагаться приблизительно на том же месте или на том же расстоянии, но с протиоположного конца) [A comparison of the Caulobacter NA1000 and K31 genomes reveals extensive genome rearrangements and differences in metabolic potential]. Этот факт хорошо согласуется с крестообразной формой карты.
Для сравнения близкородственных бактерий я выбрала разные штаммы Helicobacter pylori: F16, F30, F32 и F57, для которых есть полностью секвенированные геномы.
Для них был построен нуклеотидный пангеном (NPG) с помощью пакета NPG-explorer.
Глобальные блоки:
Число блоков: 37
Информация о блоках в виде таблицы.
Выравнивание:
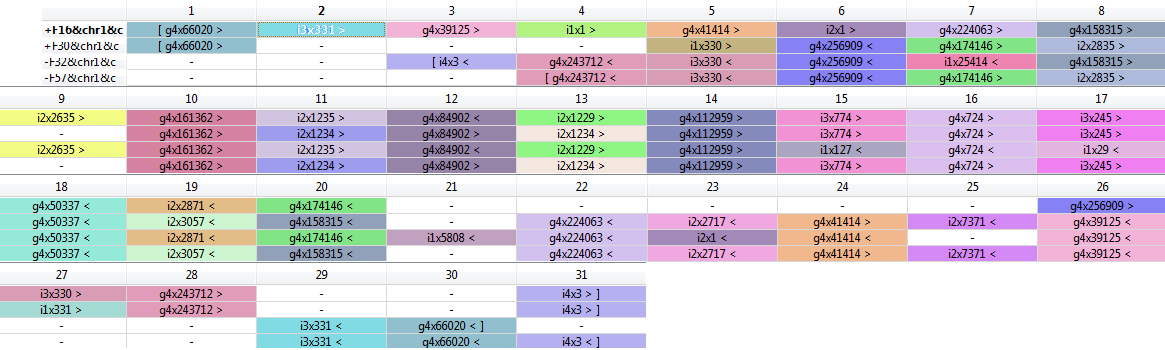
По выравниванию видно, что 5 блоков у всех штаммов расположены на одном и том же месте (10, 12, 14,16,18 столбцы).
8 столбцов выравнивания (7, 8,9,11,13,19,20 и 25) показывают разделение штаммов на две группы: F16 c F32 и F30 c F57.
Но 6 столбцов наоборот свидетельствуют о другом разделении : F16 c F30 и F32 c F57.
Так как во втором случае блоки чаще находятся против гэпов, я склонна считать этот случай менее вероятным, так как делеция в одном месте вероятнее одинаковой транслокации у предков.
У бактерий возможен горизонтальный перенос генов, поэтому установить степень родства довольно сложно.
Стабильные блоки:
Число блоков: 633
Суммарная длина и процент от длины генома: 1551378 (91.24%)
Сходство геномов: 0.943141
Повторы:
Число блоков: 185
Суммарная длина и процент от длины генома: 52557 (3.09%)
Сходство геномов: 0.85911
Пример:
r25x102:
фрагментов - 25
колонок - 102
сходство - 71,56%
генов – 25
Внутри s-блока s4x3164 есть крупные делеции:
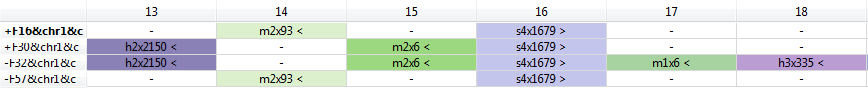
Блока h2x2150 нет у штаммов F16 и F57, а h3x335 есть только у F32.
Уникальные последовательности:
Блок u1x113, есть у штамма F16. Последовательность для различных штаммов не аннотирована, зато есть похожий ген у человека в 5 хромосоме, который кодирует белок, участвующий в биосинтезе органелл (например, лизосом). Либо это какая-то ошибка, либо ккогда-то действительно произошел перенос гена от бактерии к человеку и впоследствии стал выполнять важные функции.
Блок u1x1167 есть у F57, это метилтрансфераза 11 типа, горизонтального переноса из других организмов не происходило.
Расхождения в аннотациях для F16 и F57:
"
CDS 41885..43018
/locus_tag="HPF57_0036
/codon_start=1
/transl_table=11
/product="signal-transducing protein, histidine kinase"
/protein_id="BAJ59110.1"
CDS 41384..42529
/locus_tag="HPF16_0035"
/codon_start=1
/transl_table=11
/product="putative histidine kinase sensor protein"
/protein_id="BAJ54632.1"
Разница в названиях белка, хотя оба определенно связаны с гистидиновой киназой.
CDS 51763..54261
/locus_tag="HPF57_0047"
/codon_start=1
/transl_table=11
/product="Type IIG restriction-modification enzyme"
/protein_id="BAJ59121.1"
CDS 53660..54859
/locus_tag="HPF16_0046"
/codon_start=1
/transl_table=11
/product="adenine specific DNA methyltransferase"
/protein_id="BAJ54643.1"
/db_xref="GI:317176854"
Продукты вообще разные.
В ходе работы с геномами бактерий одного вида, но разных штаммов с помощью NPG-explorer я поняла, что представление всего генома с помощью блоков очень удобно. Также я наглядно увидела, что среди близкородственных бактерий нелегко установить однозначные родственные отношения.