| Главная страница |
| Название | Мнемоника | Таксономия |
| Agrobacterium tumifaciens | AGRRK | Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Agrobacterium; Agrobacterium tumefaciens complex |
| Rhodobacter sphaeroides | RHOS4 | Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Rhodobacter |
| Ralstonia pickettii | RALPJ | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia |
| Pseudomonas aeruginosa | PSEAE | Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales; Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa group |
| Escherichia coli | ECOLI | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia |
| Yersinia pseudotuberculosis | YERPS | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia; Yersinia pseudotuberculosis complex |
| Haemophilus influenzae | HAEIN | Bacteria; Proteobacteria; Gammaproteobacteria; Pasteurellales; Pasteurellaceae; Haemophilus |
| Erwinia tasmaniensis | ERWT9 | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Erwinia |
Скобочная форма: ((AGRRK, RHOS4), (RALPJ, (PSEAE, (HAEIN, (YERPS, (ERWT9, ECOLI))))));
Изображение дерева:
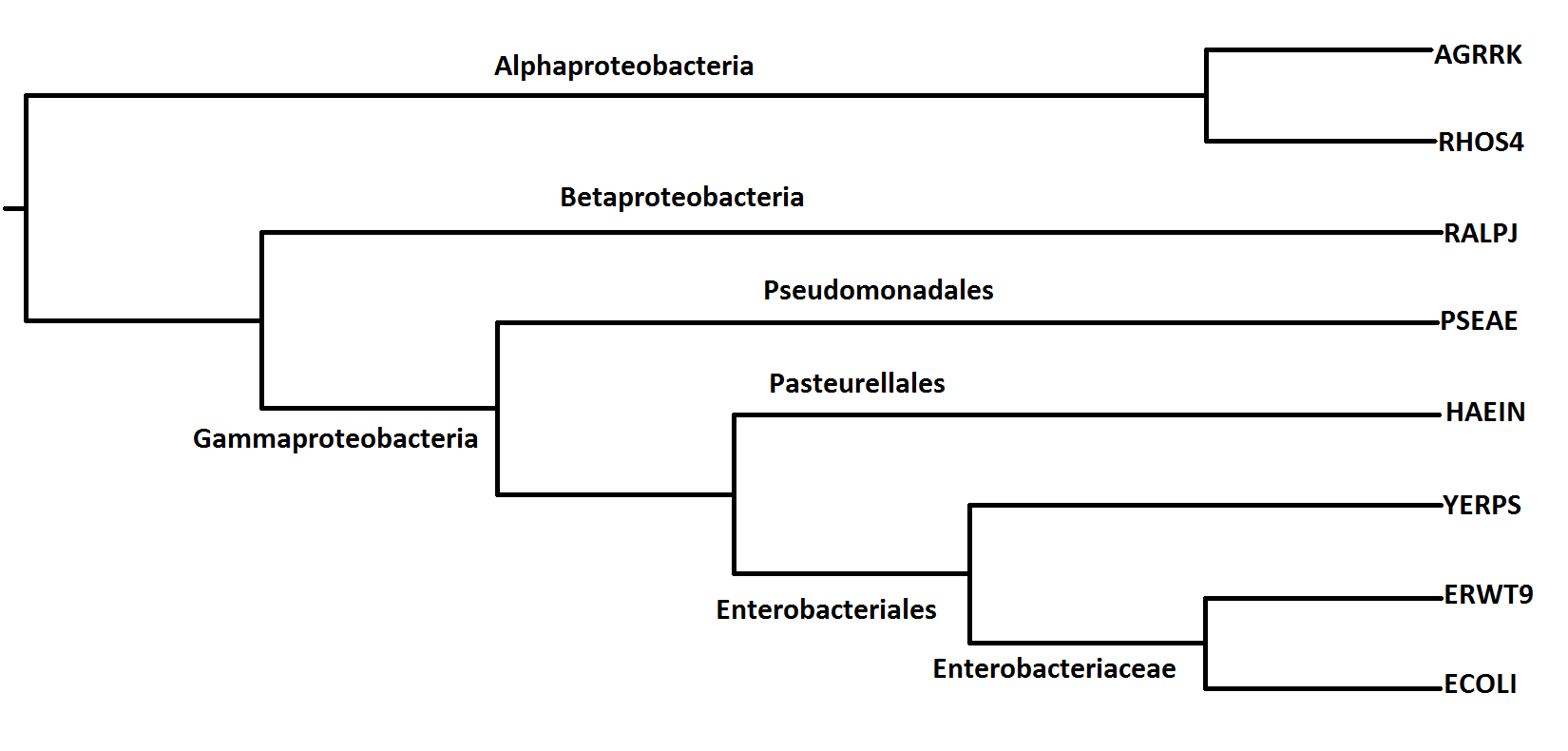
Нетривиальные ветви:
1) {AGRRK, RHOS4}, {RALPJ, PSEAE, HAEIN, YERPS, ERWT9, ECOLI} - отделяет Alphaproteobacteria от Betaproteobacteria и Gammaproteobacteria
2) {AGRRK, RHOS4, RALPJ}, {PSEAE, HAEIN, YERPS, ERWT9, ECOLI} - отделяет Gammaproteobacteria от остальных
3) {AGRRK, RHOS4, RALPJ, PSEAE}, {HAEIN, YERPS, ERWT9, ECOLI} - отделяет Enterobacteriales и Pasteurellales от Pseudomonadales
4) {AGRRK, RHOS4, RALPJ, PSEAE, HAEIN}, {YERPS, ERWT9, ECOLI} - отделяет Enterobacteriales
5) {AGRRK, RHOS4, RALPJ, PSEAE, HAEIN, YERPS}, {ERWT9, ECOLI} - отделяет Enterobacteriaceae
Также было построено дерево по последовательностям рибосомного белка S4 (RS4) этих бактерий и укоренено в среднюю точку:
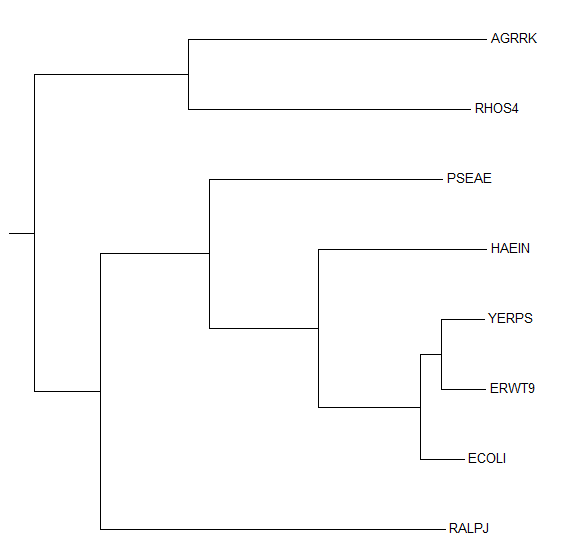
Это дерево отличается от приведенного выше относительным расположением YERPS, ERWT9 и ECOLI. Укоренение произошло в ветвь, отделяющую Alphaproteobacteria от остальных, как и в исходном дереве.
Для этих же бактерий было построено дерево по нуклеотидным последовательностям 16S рРНК.
Последовательности рибосомной РНК можно получить из базы полных геномов NCBI, нужные последовательности находятся в файлах с расширениями frn.
Последовательности были выровнены программой Muscle, дерево реконструировано методом Neighbour Joining.
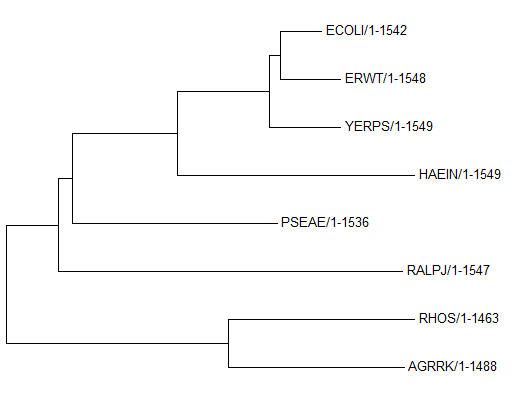
Дерево реконструировано правильно. Оно точнее, чем построенное по белкам.
С помощью BLASTа были найдены гомологи белка CLPX_ECOLI. Последовательности белков были выровнены программой Muscle, дерево построено методом Neighbour Joining.
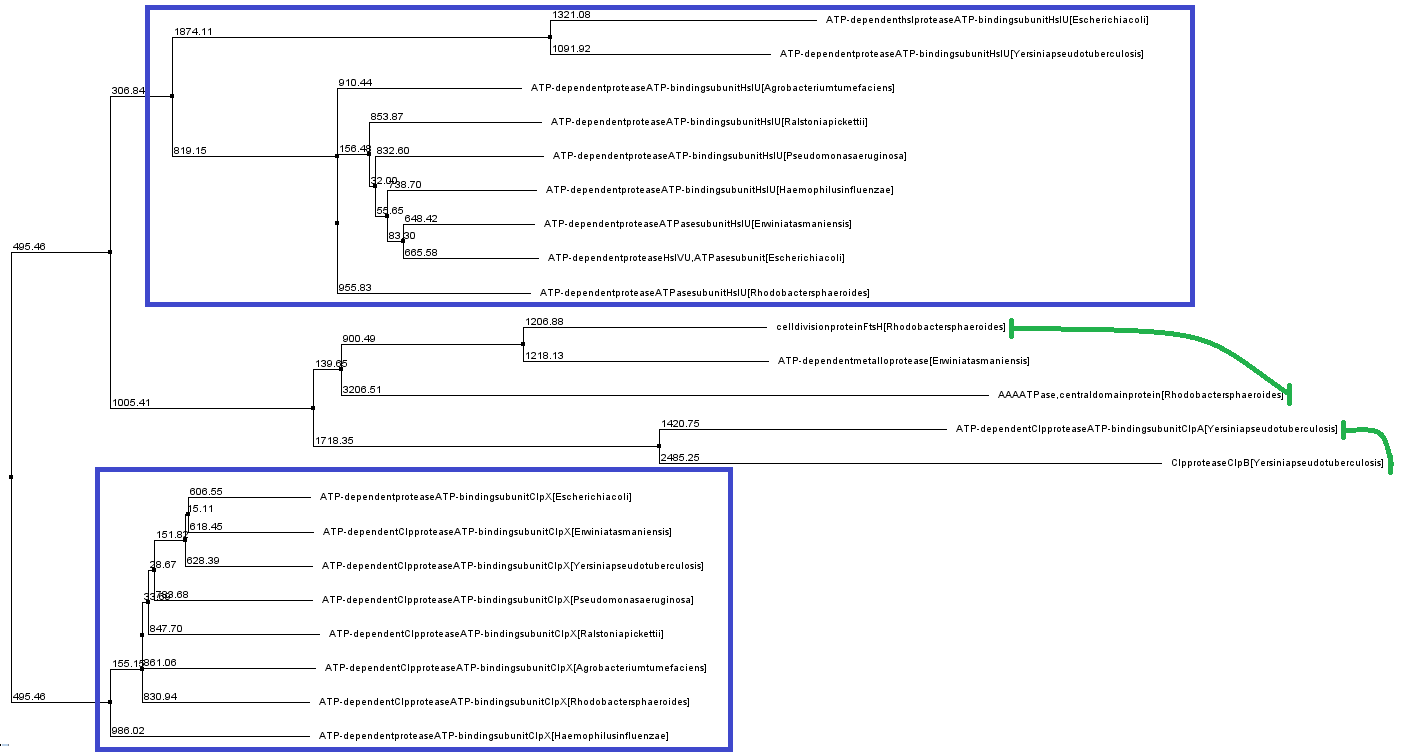
Примеры паралогов, гомологичных белков из одного организма (произошли благодаря дупликации генов), выделены зеленым.
Примеры групп попарно ортологичных белков (произошли из-за разделения путей эволюции белков в результате видообразования) выделены синим.