| Главная страница |
Сравнение систем рестрикции-модификации двух штаммов одного вида
Существование системы рестрикции-модификации предсказывается по меньшему количеству сайтов рестрикции, чем ожидаемое.С помощью метода Карлина был определен контраст (отношение наблюдаемого к ожидаемому числу сайтов) для сайтов рестрикции бактерии Selenomonas ruminantium, полученной из рубца овцы.
Всего избегаемых сайтов с контрастом < 0,78 семнадцать.
Файл веб-сервиса (tmpKDUDwF.tsv), файл с отобранными избегаемыми сайтами (tmpKDUDwF_fin.tsv).
Теперь был определен контраст для набора контигов из метагенома кишечника человека: для такого же порога нашлось всего три сайта, и все из них встречаются в первом случае.
Файл веб-сервиса, файл с отобранными избегаемыми сайтами.
Видно, что у бактерии, которая жила в желудке овцы, больше систем рестрикции-модификации, возможно, это связано с тем, там больше бактерий, между которыми может происходить горизонтальный перенос генов.
Поиск последовательности Шайн-Дальгарно
Поиск последовательности Шайн-Дальгарно проводился для бактерии Flavobacterium branchiophilum.Для работы я отобрала CDS средней длины (400-600 нуклеотидов).
В литературных данных я нашла информацию, что последовательность Шайн-Дальгарно обычно расположена за 7-12 нуклеотидов до старт-кодона. [1, 2, 3] Также я нашла информацию, что в плазмиде Flavobacterium она располагается за 7 нуклеотидов до старт-кодона и выглядит так: AAGG. [4]
В одной статье [3] при поиске последовательности Шайн-Дальгарно исследователи рассматривали область от -20, и я решила сделать так же.
Для поиска по всем генам я взяла область от -30.
Для создания мотива я запустила MEME с параметром длина мотива: от 4 до 6.
Результат представлен на рисунке 1.
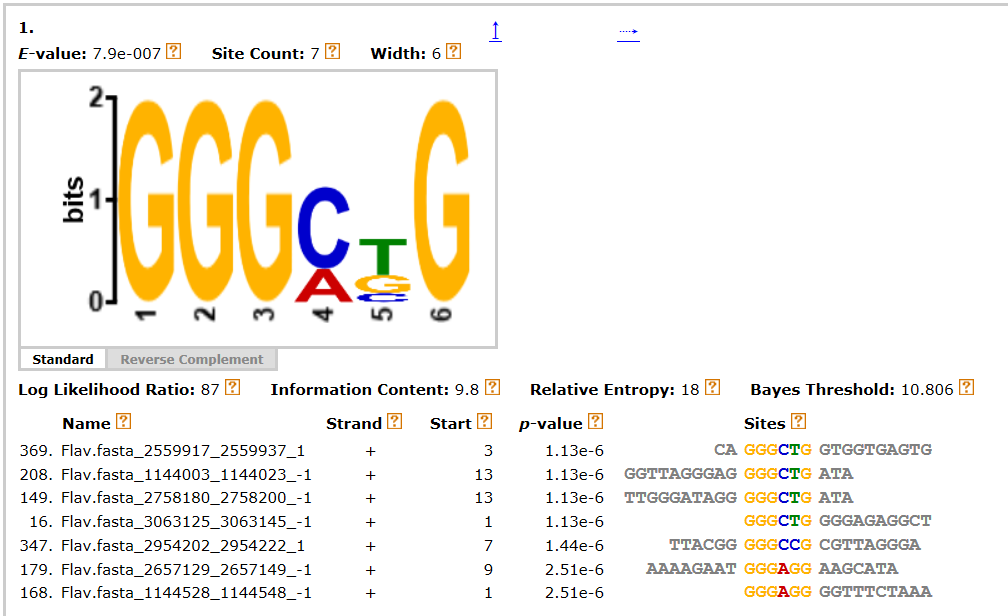
Рис.1 Мотив, полученный с помощью программы MEME.
Позиционная матрица весов для правильного мотива:
-----------------------------------------------------------------
Motif 1 position-specific probability matrix
-----------------------------------------------------------------
letter-probability matrix: alength= 4 w= 6 nsites= 7 E= 7.9e-007
0.000000 0.000000 1.000000 0.000000
0.000000 0.000000 1.000000 0.000000
0.000000 0.000000 1.000000 0.000000
0.285714 0.714286 0.000000 0.000000
0.000000 0.142857 0.285714 0.571429
0.000000 0.000000 1.000000 0.000000
При значении p-value=0,1 такой мотив нашелся в 2433 последовательностях из 3028: файл с результатами.
Первый мотив имеет E-value: 7.9e-007 и встречается всего в 7 последовательностях. Он не очень похож на консенсусную последовательность Шайн-Дальгарно AGGAGG или на предполагаемую последовательность из плазмиды Flavobacterium AAGG.
Если в хромосоме последовательности Шайн-Дальгарно действительно такие же короткие, как и в плазмиде, найти их будет проблемой.
Определение сайтов связывания транскрипционного фактора в участке хромосомы человека
Для анализа был взят файл с ридами Illumina, полученный в результате сhip-seq эксперимента; с помощью программы FastQC оценено качество прочтения – оно довольно высокое: все значения в зеленой области, можно целиком использовать его для анализа (Рис.2).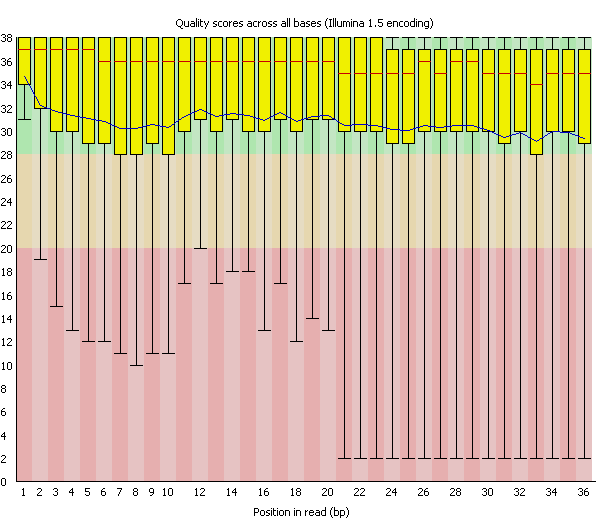
Рис.2 Качество прочтения.
Картирование прочтений на геном человека hg19 выполнялось с помощью команды: bwa mem /srv/databases/ngs/hg19/GRCh37.p13.genome.fa chipseq_chunk45.fastq > chipseq_chunk45.sam
Для дальнейшего анализа я использовала следующие команды:
samtools view -bSo chipseq_chunk45.bam chipseq_chunk45.sam – перевод файла в бинарный формат
samtools sort chipseq_chunk45.bam chipseq_chunk45.sorted.bam – сортировка по координатам
samtools index chipseq_chunk45.sorted.bam - идексирование
samtools idxstats chipseq_chunk45.sorted.bam > chipseq_chunk45.idxstats – получение информации о том, куда откартировались риды и их количестве
samtools view -c chipseq_chunk45.sorted.bam – подсчет ридов, откартировавшихся на геном
Все 40573 рида откартировались на хромосому.
Большинство (388895) ридов откартировались на 14 хромосому, следовательно, для анализа была взята именно эта хромосома. Помимо 14 хромосомы риды картировались на другие хромосомы, включая половые и митохондриальный геном (Рис.3).
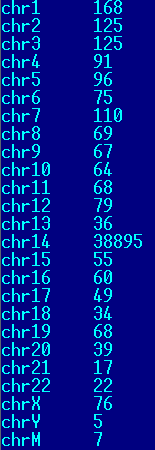
Рис.3 Места картирования ридов.
С помощью команды macs2 callpeak -t chipseq_chunk45.sorted.bam –nomodel было найдено 19 пиков, все они расположены рядом (31028019-32553378), длина колеблется от 200 до 665, средняя – 362 нуклеотида.
Для визуализации информации (Рис.4) в файл NA_peaks.narrowPeak были дописаны строчки:
track type=narrowPeak visibility=3 db=hg19 name="my_peaks" description="Peaks from chunk 45"
browser position chr14:31028019-32553378
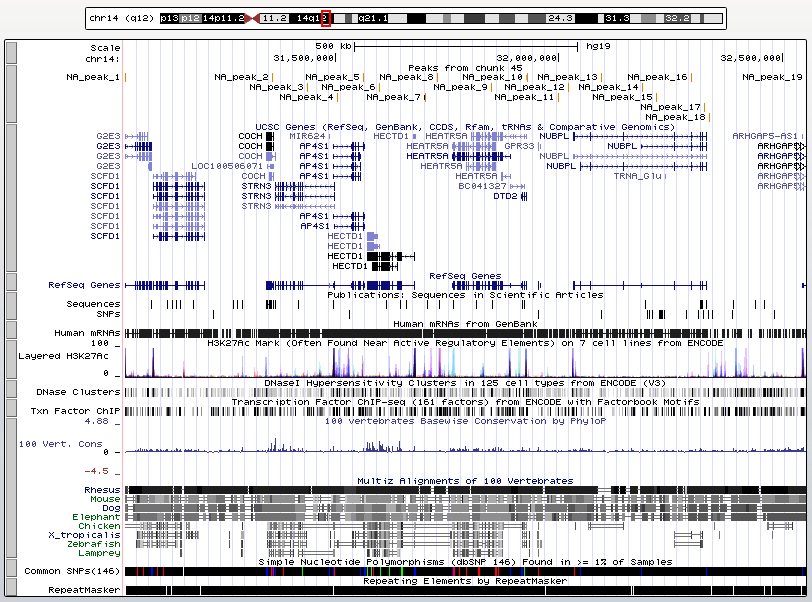
Рис.4 Пики.
Пик 7 имеет следующие характеристики:
Позиция: 31698711-31699227
Ширина: 517
Вершина: 31699026 (на расстоянии 315 нуклеотидов от начала и 201 от конца)
-log10(P-value): 145.145, это наиболее достоверный пик
К сожалению, он расположен далеко от генов.
Пик 1:
Позиция: 31028019-31028397
Ширина: 379
Вершина: 31028208 (на расстоянии 198 нуклеотидов от начала и 189 от конца)
-log10(P-value): 35.3
Своим концом пересекается с началом гена G2E3 G2/M-фазо-спецефичной убиквитин-лигазы, вполне возможно, что это транскрипционный фактор.
TATA-бокс
TATA-бокс связывающий фактор TBP - архейный и эукариотический белок, узнающий восьминуклеотидный сигнал в ДНК с консенсусом TATA(А/Т)AA(A/G). Не все промоторы имеют сигнал TATA-box.Я выбрала эксперимент ChiP-Seq, проводившийся на клеточной линии HeLa-S3, антитела на TBP – ab62126.
Рядом с геном SGO1, вроде бы, имеется сигнал TBP, однако в последовательности нет ничего похожего на TATA-бокс (Рис.5).
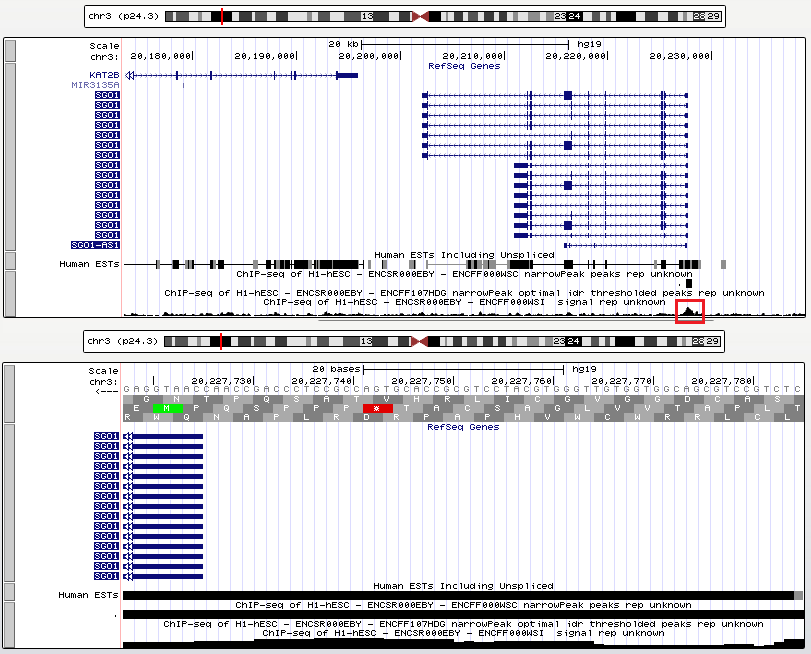
Рис.5 Промоторная область гена SGO1.
Продукт гена защищает центромерный когезин от расщепления.
Начало: 20202085, конец: 20227725, на обратной цепи, длина 2491.
Рядом с геном SSR3 имеется сигнал TBP, и в последовательности можно найти область похожую на TATA-бокс – TATcTAgt (Рис.6).
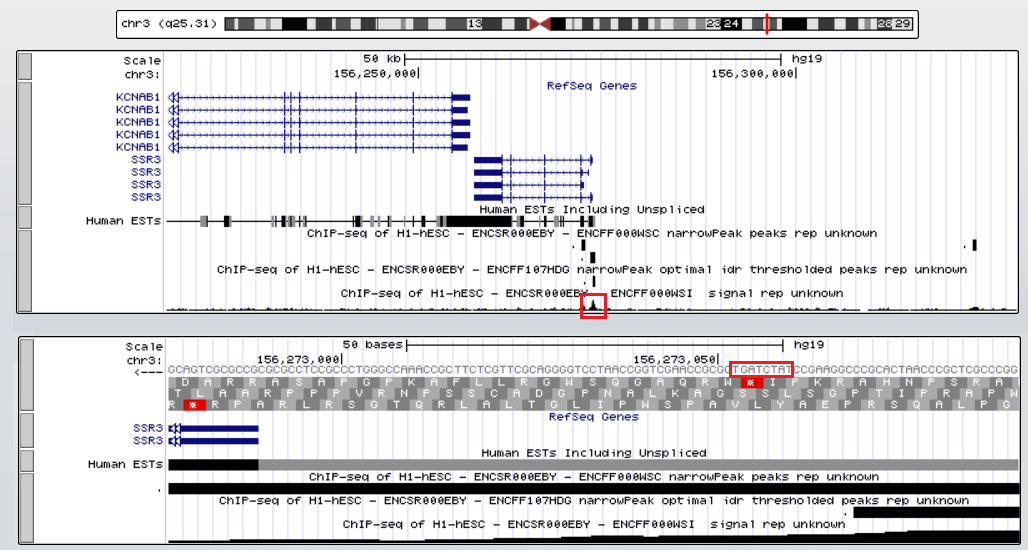
Рис.6 Промоторная область гена SSR3.
Ген кодирует субъединицу гамма транслокон-ассоциированного белка.
Начало: 156257929, конец: 156722973, на обратной цепи, длина 465045.
Рядом с геном RFTN1 имеется сигнал TBP, и в последовательности можно найти область похожую на TATA-бокс: TATAcgAt (Рис.7).
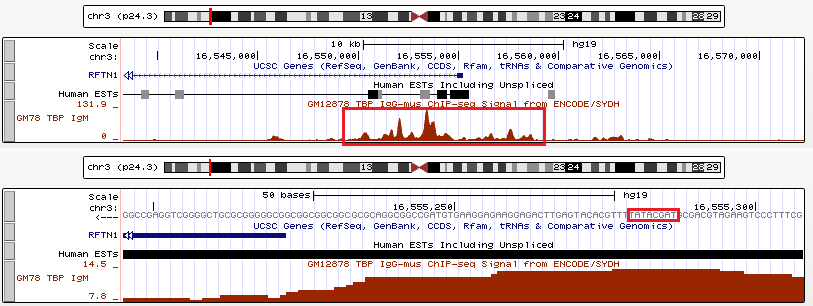
Рис.7 Промоторная область гена RFTN1.
Ген кодирует субъединицу гамма транслокон-ассоциированного белка.
Начало: 16358352, конец: 16555222, на обратной цепи, длина 197871.
Рядом с геном GYG1 имеется сигнал TBP, и в последовательности можно найти область очень похожую на TATA-бокс: TATAAgAG, однако она лежит в начале кодирующей области (Рис.8).
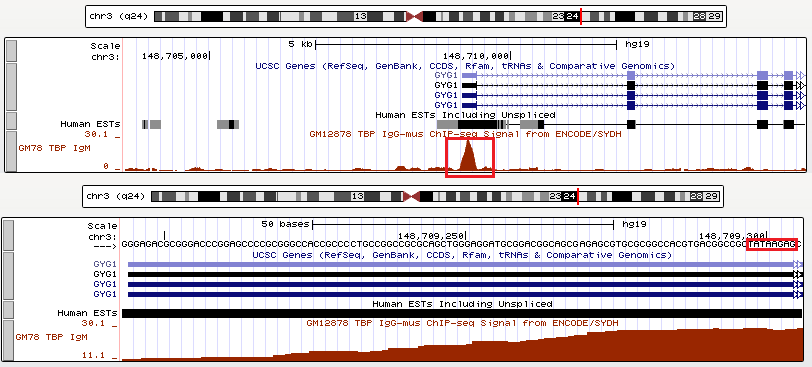
Рис.8 Промоторная область гена GYG1.
Ген GYG1, кодирует гликогенин, катализирующий автогликозилирование.
Начало: 148709195 конец: 148742059, на прямой цепи, длина 32865.
Литература:
[1]-Tompa M. An exact method for finding short motifs in sequences, with application to the ribosome binding site problem.[2]-Starmer J, Stomp A, Vouk M, Bitzer D. Predicting Shine-Dalgarno sequence locations exposes genome annotation errors.
[3]-Motalleb G. Listeria Monocytogenes La111 and Klebsiella Pneumoniae KCTC 2242: Shine-Dalgarno Sequences.
[4]-Wiesmann UN, DiDonato S, Herschkowitz NN. Effect of chloroquine on cultured fibroblasts: release of lysosomal hydrolases and inhibition of their uptake.