import pybel
import subprocess, os
Сравнение структур порфирина, полученных разными способами:
mol=pybel.readstring('smi','C1=CC2=CC3=CC=C(N3)C=C4C=CC(=N4)C=C5C=CC(=N5)C=C1N2')
mol.addh()
mol.make3D()
mol.write(format='pdb',filename='mol.pdb', overwrite=True)
mol
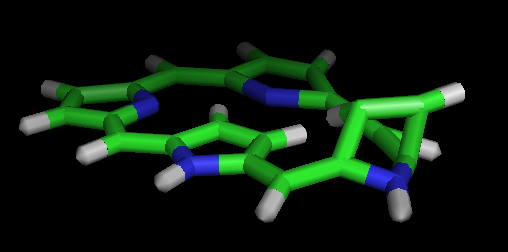
Структура, полученная с помощью pybel, не похожа на нормальный порфирин.
def mopac(mol, name, method, charge=None):
os.system("export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'")
mop = mol.write(format='mopin', filename='%s.mop' % name, opt={'k':'%s CHARGE=%s XYZ' % (method, charge)},
overwrite=True)
os.system("echo | /home/preps/golovin/progs/mopac/MOPAC2016.exe %s.mop" % name)
opt=pybel.readfile('mopout','%s.out' % name)
for i in opt:
i.write(format='pdb',filename='%s.pdb' % name, overwrite=True)
mopac(mol, "mol_AM1", "AM1")
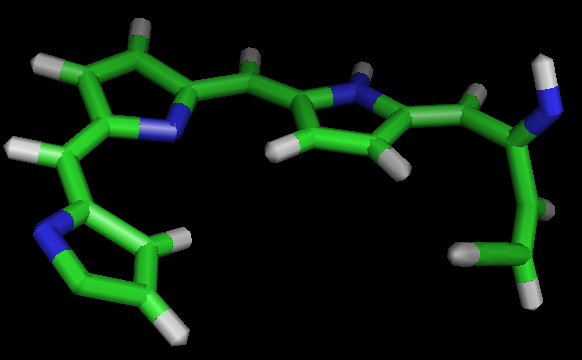
Что-то пошло не так...
mol.make3D(steps=100)
mopac(mol, "mol_AM1", "AM1")
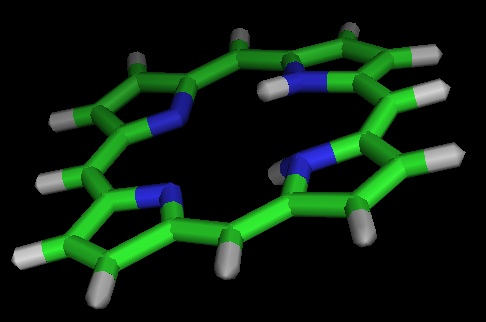
Вот теперь молекула порфирина почти плоская.
mopac(mol, "mol_PM6", "PM6")
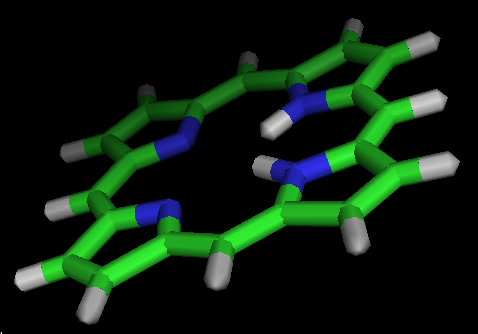
Метод PM6 еще лучше справился с оптимизацией.
os.system("cp mol_PM6.mop mol_spectr.mop")
os.system('echo "" >> mol_spectr.mop')
os.system('echo "cis c.i.=4 meci oldgeo" >> mol_spectr.mop')
os.system('echo "some description" >> mol_spectr.mop')
os.system("export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'")
os.system("echo | /home/preps/golovin/progs/mopac/MOPAC2016.exe mol_spectr.mop")
f = open('mol_spectr.out', 'r')
lines = f.readlines()[1072:1080]
f.close()
energies = []
for line in lines:
energies.append(float(line.split()[1]))
print('energies: ' + str(energies))
print('')
wave_len=[]
for i in range(len(energies)):
wave_len.append(1239.842/energies[i])
print('wave lenghts: ' + str(wave_len))
Имитация возбуждения тиминового димера:
td = pybel.readfile("pdb", "td.pdb").next()
mopac(td, "td_0", "PM6", "0")
td_0 = pybel.readfile("pdb", "td_0.pdb").next()
mopac(td_0, "td_+2", "PM6", "+2")
td_2 = pybel.readfile("pdb", "td_+2.pdb").next()
mopac(td_2, "td_0_2", "PM6", "0")
На рисунке: оптимизация тиминового димера при заряде системы 0, при заряде системы +2, оптимизация предыдущего состояния при заряде 0.
Видимо, система из двух тиминов энергетически выгоднее, чем димер, поэтому возбуждённое состояние не перешло обратно в исходное состояние.
Оптимизация нафталена и азулена:
nap=pybel.readstring('smi','C1=CC=C2C=CC=CC2=C1')
nap.addh()
nap.make3D(steps=100)
nap.write(format='pdb',filename='nap.pdb', overwrite=True)
nap
azu=pybel.readstring('smi','C1=CC=C2C=CC=C2C=C1')
azu.addh()
azu.make3D(steps=100)
azu.write(format='pdb',filename='azu.pdb', overwrite=True)
azu
mopac(nap, "nap_opt", "PM6")
mopac(azu, "azu_opt", "PM6")
def orca(mol, name, method):
mol = pybel.readfile('pdb', '%s.pdb' % mol).next()
mol.write(format='orcainp',filename='nap.inp', overwrite=True)
orca = mol.write(format='orcainp',opt={'k': method}, overwrite=True)
orca_out = orca.replace('! insert inline commands here ', method)
outfile = open('%s.inp' % name, 'w')
outfile.write(orca_out)
outfile.close()
os.system('/srv/databases/orca/orca %s.inp | tee %s.log' % (name, name))
orca('nap_opt', 'nap_hf', '!HF RHF 6-31G')
orca('nap_opt', 'nap_dft', '!DFT RHF 6-31G')
orca('azu_opt', 'azu_hf', '!HF RHF 6-31G')
orca('azu_opt', 'azu_dft', '!DFT RHF 6-31G')
%%bash
grep "Total Energy" *.log
| Вещество | E Naphthalene | E Azulene | ΔE, Hartree | ΔE, kCal/mol |
| Хартри-Фок | -383.22013147 | -383.22005947 | 0.00002915 | 0.01829191 |
| DFT | -382.25557440 | -382.25560355 | 0.00007200 | 0.04518069 |
Энергии нафталена и азулена оказались слишком близки и энергия изомеризации очень сильно отличается от эксперементальных 35.3±2.2 kCal/mol, поэтому нельзя сказать, какой способ лучше.