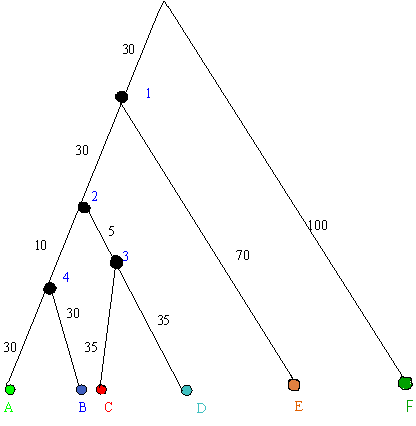
Узлы подписаны цифрами (1, 2, 3, 4), листья — буквами (A, B, C, D, E, F).
Расстояния даны как число мутаций на 100 нуклеотидных остатков.
Длина гена — 945 нуклеотидных остатков.

|
Дерево укоренненое (корень — ген белка TRUB_ECOLI).
Узлы подписаны цифрами (1, 2, 3, 4), листья — буквами (A, B, C, D, E, F). Расстояния даны как число мутаций на 100 нуклеотидных остатков. Длина гена — 945 нуклеотидных остатков. |
A 0.0000 0.4543 0.5807 0.6355 1.0501 1.4484 B 0.4543 0.0000 0.5784 0.6112 1.0248 1.4411 C 0.5807 0.5784 0.0000 0.5065 0.9498 1.4339 D 0.6355 0.6112 0.5065 0.0000 1.0207 1.4935 E 1.0501 1.0248 0.9498 1.0207 0.0000 1.4484 F 1.4484 1.4411 1.4339 1.4935 1.4484 0.0000
+-------------A
+---1
! +-------------B
+------------3
! ! +---------------C
+------------4 +--2
! ! +---------------D
--5 !
! +------------------------------E
!
+-------------------------------------------F
((((A:0.22715,B:0.22715):0.07358,(C:0.25325,D:0.25325):0.04748):0.20495,
E:0.50567):0.22086,F:0.72653);
|
Дерево построено по методу UGPMA с помощью
программы eneighbor. Для построения
филогенетическоо дерева метод UGPMA
использовал матрицу попарных эволюционных расстояний.
Дерево укорененное и ультраметрическое. |
+------C
+--3
! +--------D
!
! +---------------E
--4-----1
! +----------------------------F
!
! +------A
+--2
+------B
((C:0.22599,D:0.28051):0.03916,(E:0.50276,F:0.94564):0.20786,(A:0.23430,
B:0.22000):0.08189);
|
Неукорененное дерево построено по методу ближайших соседей (Neighbor-Joining) с помощью программы eneighbor. Для построения филогенетического дерева метод Neighbor — Joining использовал матрицу попарных эволюционных расстояний. |
+------B ! ! +--------D ! ! --1---2 +-------------------------------F ! ! +-------4 ! +--3 +---------------E ! ! ! +------C ! +------A(B:0.22532,(D:0.28391,((F:1.05160,E:0.52828):0.24568, C:0.22695):0.01704):0.11378,A:0.23755); |
Дерево неукорененное. Построено по методу наибольшего правдоподобия (Maximum Likelihood) с помощью программы ednaml . Метод Maximum Likelihood — символьно-ориентированный. |
+--F
+--5
+--4 +--E
! !
+--3 +-----D
! !
+--2 +--------C
! !
--1 +-----------B
!
+--------------A
(((((F,E),D),C),B),A);
|
Дерево неукорененное. Построено по методу максимальной экономии (Parsimony) с помощью программы ednapars . Метод Parsimony — символьно-ориентированный. |
Табл.1. Сравнение топологии исходного филогенетического дерева модели и 4-х вариантов его реконструкции
| Ветвь | Исходное дерево модели |
UPGMA | NJ | ML | Parsimony |
| ABCDEF | |||||
| 110000 | + | + | + | + | + |
| 001100 | + | + | + | - | - |
| 000011 | + | + | + | + | + |
| 001011 | - | - | - | + | - |
| 000111 | - | - | - | - | + |
Методы UGPMA и
Neighbor-Joining на 100% реконструировали исходную топологию
модельного филогенетического дерева гена белка TRUB_ECOLI.
Топологии деревьев, построенных методами наибольшего правдоподобия
(Maximum Likelihood) и максимальной экономии (Parsimony), не содержат описания
ветви, на одном конце которой находятся листья A, B, E, F, а на другом — C,
D, зато содержат иные описания ветвей (см. Таблицу 1).
Реконструкция хода эволюции для такого уровня расстояниях между
последовательностями достаточно надежно воспроизводится методами
UGPMA и Neighbor-Joining
.
Наиболее эффективным мне показался метод UGPMA
, который правильно
реконструировал и укоренил дерево.
На главную страницу третьего семестра
© Армаш Татьяна,2005