Схема ферментативной реакции:
Ингибиторы фермента EC 3.5.4.4
Ингибиторов очень много. Среди них, например, 2-деоксикоформицин, аденин,
АТФ, АМФ, ЭДТА, фолиевая кислота, гуанозин, гепарин, инозин, лизоцим и многие
другие вещества.
Ниже представлены 2 ингибитора: ЭДТА и фолиевая кислота
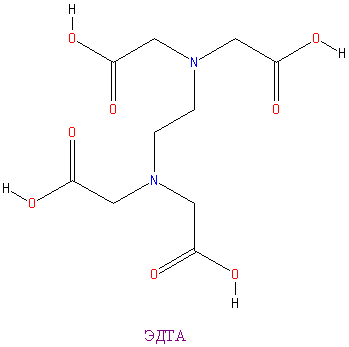
|
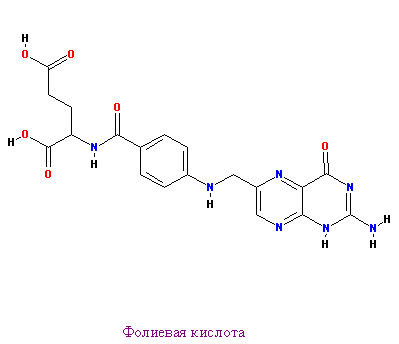
|
Активаторы фермента EC 3.5.4.4
Количество приведенных активаторов значительно уступает количеству ингибиторов.
Интересно, что в организме Candida albicans АМФ является как активатором, так и
ингибитором фермента EC 3.5.4.4, а АТФ, инозин и аденин в Candida albicans
играют роль активаторов, а в других организмах — ингибиторов. К активаторам
также относятся N-ацетил-D-глюкозамин, дибутурил-цАМФ.
Ниже представлены АМФ и дибутирил-цАМФ
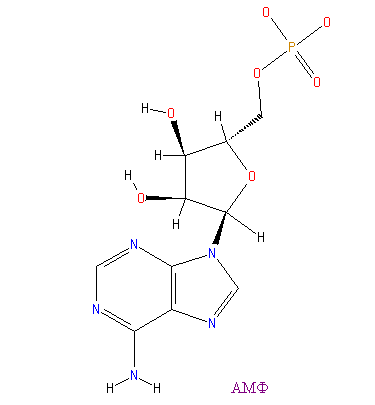
|

|
Таблица №1 Оптимум рН фермента EC 3.5.4.4
Таблица №2. Субъединичные структуры, характерные для фермента EC 3.5.4.4

|
Для данного типа фермента характерна мономерная структура. Но так же встречаются белки с димерной и полимерной структурами. |
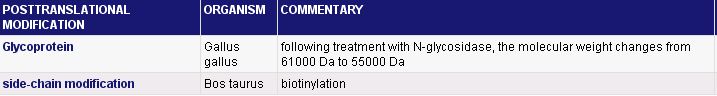
Таблица №3. Посттрансляционные модификации фермента EC 3.5.4.4
В 3 организмах (Escherichia coli, Methanococcus jannaschii, Homo sapiens) в базе данных белковых последовательностей Swiss-Prot был произведен поиск ферментов с кодом EC 3.5.4.4 по следующему запросу:
"((([swissprot-EntryName:*_Ecoli*] | [swissprot-EntryName:*_Human*]) |
[swissprot-EntryName:*_Metja*]) & [swissprot-ECNumber:3.5.4.4*]) "
В результате было найдено лишь 2 белка из Homo sapiens (
P00813) и
Escherichia coli (P22333
)
Найденные белки содержат домен Adenosine/AMP deaminase, описанный в Pfam.
Идентификатор домена Adenosine/AMP deaminase в InterPro —
IPR001365
Идентификатор домена Adenosine/AMP deaminase в Pfam —
PF00962
Таблица №4. Расположение домена Adenosine/AMP deaminase в белках P00813 (ada_human) и P22333 (add_ecoli)
| Организм | Начало домена | Конец домена |
| Homo sapiens ( P00813) | 7 | 345 |
| Escherichia coli (P22333) | 5 | 329 |
#=======================================
#
# Aligned_sequences: 2
# 1: ADA_HUMAN
# 2: ADD_ECOLI
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 352
# Identity: 113/352 (32.1%)
# Similarity: 175/352 (49.7%)
# Gaps: 40/352 (11.4%)
# Score: 416.0
#
#
#=======================================
ADA_HUMAN 1 DKPKVELHVHLDGSIKPETILYYGRRRGIALPANTAEGLL---NVIGMDK 47
..|..::|.||||:|:|:|||..||:..|:|||.:.|.|: .||..:.
ADD_ECOLI 1 TLPLTDIHRHLDGNIRPQTILELGRQYNISLPAQSLETLIPHVQVIANEP 50
ADA_HUMAN 48 PLTLPDFLAKFDYYMPAIAGCREAIKRIAYEFVEMKAKEGVVYVEVRYSP 97
.|. .||.|.|:.:..:|.. :|.:|:|:|.:|..|:.|:.|||:|:||
ADD_ECOLI 51 DLV--SFLTKLDWGVKVLASL-DACRRVAFENIEDAARHGLHYVELRFSP 97
ADA_HUMAN 98 HLLANSKVEPIPWNQAEGDLTPDEVVALVGQGLQEGERDFGVKARSI--- 144
..:|.:...|:. .||..|..|::||.|.|||:|:.|
ADD_ECOLI 98 GYMAMAHQLPVA-----------GVVEAVIDGVREGCRTFGVQAKLIGIM 136
ADA_HUMAN 145 ------LCCMRHQPNWSPKVVELCKKYQQQTVVAIDLAGDET-IPGSSLL 187
..|.:. :|....::.| :.|:||||||. .|||..|
ADD_ECOLI 137 SRTFGEAACQQE--------LEAFLAHRDQ-ITALDLAGDELGFPGSLFL 177
ADA_HUMAN 188 PGHVQAYQEAVKSGIHRTVHAGEVGSAEVVKEAVDILKTERLGHGYHTLE 237
.| :..|..:|.|.||||||....|.:.:|:..|..||:|||...:|
ADD_ECOLI 178 -SH---FNRARDAGWHITVHAGEAAGPESIWQAIRELGAERIGHGVKAIE 223
ADA_HUMAN 238 DQALYNRLRQENMHFEICPWSSYLTGAWKPDTEHAVIRLKNDQANYSLNT 287
|:||.:.|.::.:..|.|..|:..|........|.:..........|:||
ADD_ECOLI 224 DRALMDFLAEQQIGIESCLTSNIQTSTVAELAAHPLKTFLEHGIRASINT 273
ADA_HUMAN 288 DDPLIFKSTLDTDYQMTKRDMGFTEEEFKRLNINAAKSSFLPEDEKRELL 337
|||.:....:..:|.:.....|.:.|:.::..||..:.:||..:|||.|.
ADD_ECOLI 274 DDPGVQGVDIIHEYTVAAPAAGLSREQIRQAQINGLEMAFLSAEEKRALR 323
ADA_HUMAN 338 DL 339
:.
ADD_ECOLI 324 EK 325
#---------------------------------------
#---------------------------------------
Процент идентичности последовательностей доменов очень маленький (32.1%), но при
этом данные последовательности представляют один и тот же домен (элемент
третичной структуры белка) Adenosine/AMP deaminase. Этот факт еще раз подтверждает,
что пространственные структуры более консервативны, чем последовательности.
На главную страницу четвертого семестра
© Армаш Татьяна,2006