Практикум 10: макромолекулярный докинг¶
Оформление: 10.05.2018
Для начала я задала путь к zdock:
1772 export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib 1773 export PATH=${PATH}:/home/preps/golovin/progs/bin
Далее я скачала файлы amilase.pdb и camelid.pdb и добавила к ним водороды с помощью программы pdb2gmx:
1788 pdb2gmx -f camelid.pdb -o camelid_h.pdb -p -ignh 1773 pdb2gmx -f amylase.pdb -o amylase_h.pdb -p -ignh
Провела препроцессинг утилитой marksur. Программа вставляет в pdb-файл дополнительные колонки: вероятно, заряд и радиус атома.
1998 mark_sur amylase_h.pdb amylase_h_m.pdb 1999 mark_sur camelid_h.pdb camelid_h_m.pdb
Вручную удалила строки MODEL и TER из полученных pdb-файлов.
Запустила докинг командой (работает несколько минут):
zdock -N 1000 -R amylase_h_m.pdb -L camelid_h_m.pdbВ файле zdock.out.cp.zr.out записана характеристика всех полученных структур. Небольшой командой можно получить топ-10 из них:
%%bash
! sort -n -k2 zdock.out.cp.zr.out | head
Посмотрим на лучшую структуру complex.595.pdb:
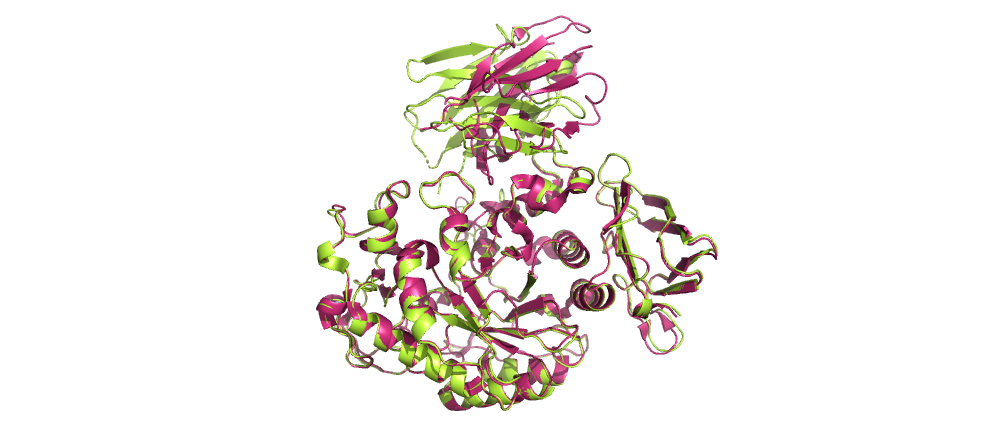 Cтруктура complex.455.pdb:
Cтруктура complex.455.pdb:
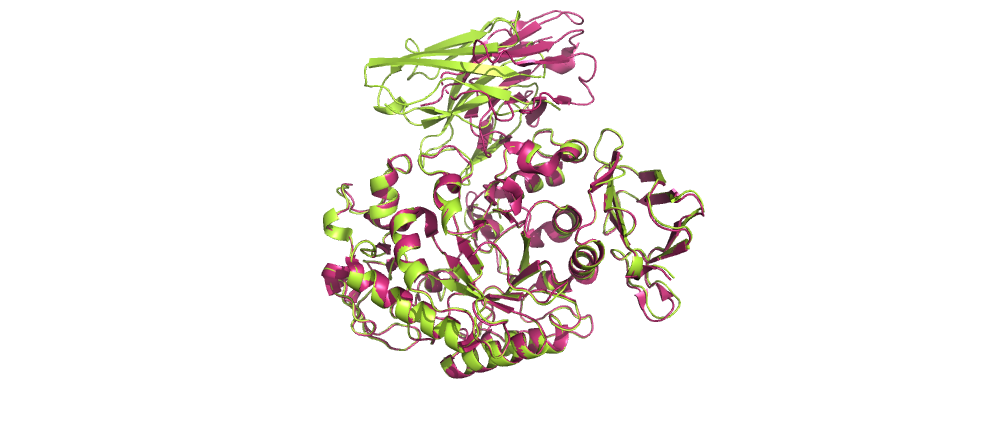 Cтруктура complex.474.pdb:
Cтруктура complex.474.pdb:
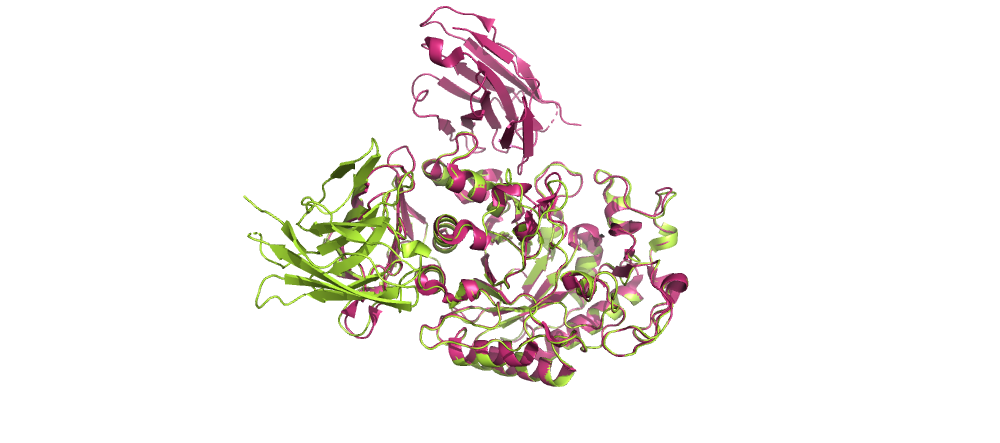 Cтруктура complex.883.pdb:
Cтруктура complex.883.pdb:
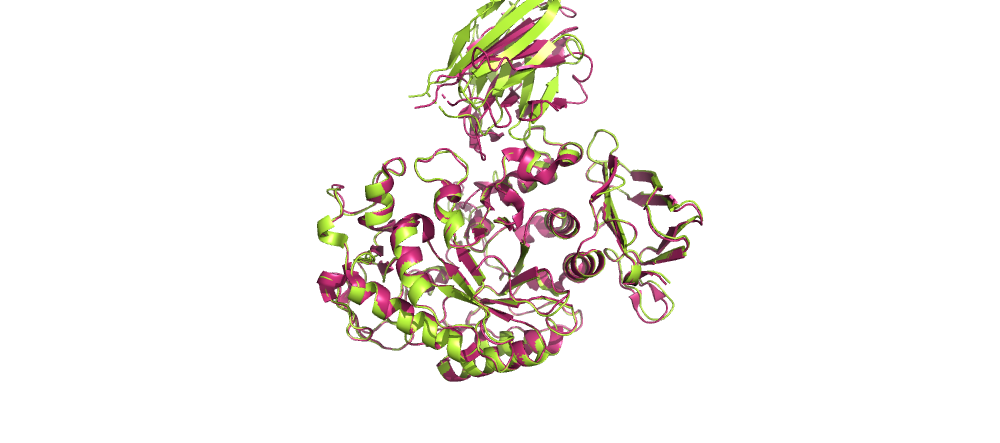 Cтруктура complex.737.pdb:
Cтруктура complex.737.pdb:
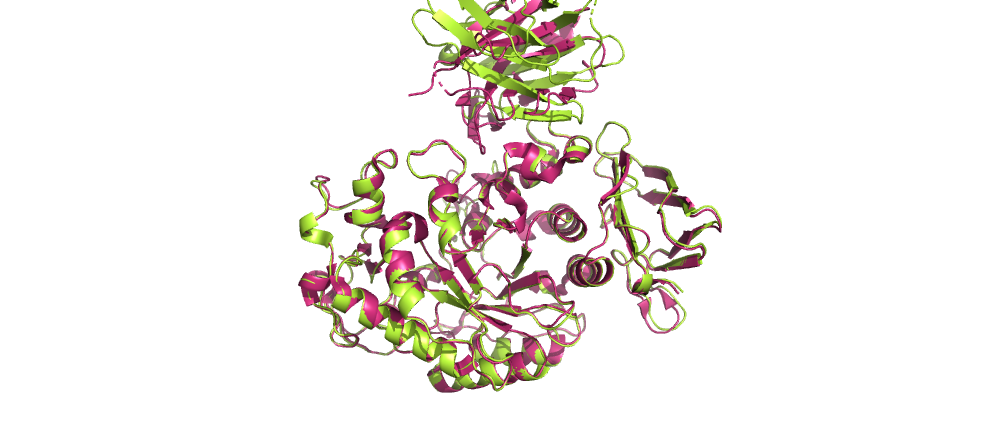
Розовым здесь отмечена референсная структура 1kxt.pdb, зеленым - результат докинга.
Видно, что лучшая структура, полученная в результатае докинга, довольно похожа на референсную.