Практикум 12: Анализ молекулярной динамики биологических молекул в GROMACS¶
12.05.2018
%%bash
trjconv -f b_md.xtc -s b_md.tpr -o b_pbc_1.pdb -skip 20 -pbc mol
Из него и было получено видео, выложенное мной в практикуме 11:
Последняя модель похожа на переходную стадию между липидной мицеллой и бислоем, так как гидрофильные головки не располагаются параллельно, а образуют вытянутую окружность. Возможно, это происходит из-за того, что липиды встали в ячейке кривовато, и вместо образования слоя с "мнимыми" соседними ячейками образовали получившуюся форму.
С помощью стороннего скрипта я определила, как располагается бислой относительно координатных осей. Большую часть времени ось Y является нормалью к поверхности слоя, хотя в некоторые моменты он несколько поворачивается в плоскости XY.
Я перемножила колонки XX и ZZ из файла box_1.xvg, разделила полученные значения на 32 - примерное число липидов в слое.
Ниже приведен полученный график:
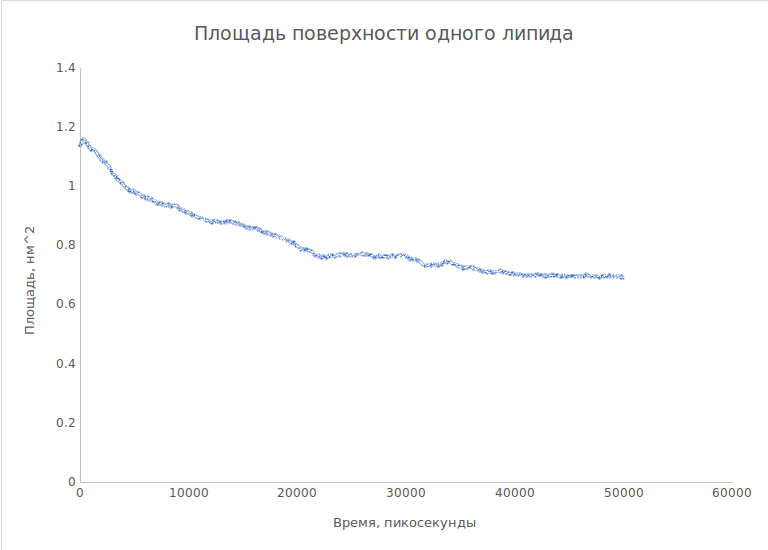
С небольшими флуктуациями, которые как раз могут быть вызваны поворотами всего бислоя, площадь заметно убывает.
Гидрофобная и гидрофильная поверхность, доступная для растворителя¶
Следующей коммандой получим значения площадей гидрофобной и гидрофильной поверхностей.
g_sas -f b_md.xtc -s b_md.tpr -o sas_b.xvg
Построим графики зависимости площадей этих поверхностей от времени в одних и тех же границах значений по оси y:
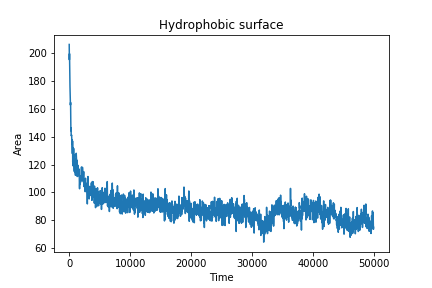
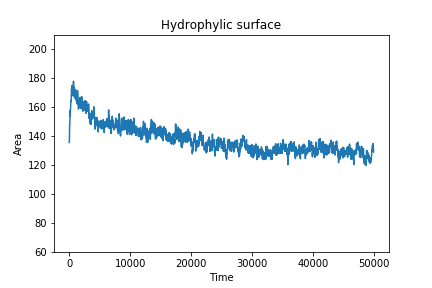
Резкое падение площади гидрофобной поверхности доступной для растворителя - это и есть этап обращения гидрофобных хвостов внутрь "мицеллы". Площадь гидрофильной поверхности сначала растет, это происходит, возможно, просто из-за сближения групп при формировании мицеллы. Потом она начинает медленно снижаться в процессе "утряски" бислоя.
Оценка меры порядка¶
С помощью команд:
g_order -s b_md -f b_md.xtc -o ord_end.xvg -n sn1.ndx -b 45000 -d Y
g_order -s b_md -f b_md.xtc -o ord_start.xvg -n sn1.ndx -e 5000 -d Y
получаем файлы с данными по мере порядка для начала и конца траектории. Построим графики по данным файлов.
f1 = open('ord_start.xvg', 'r')
x_axis = []
y_axis = []
for line in f1:
if line[0] == ' ':
l = line.strip().split()
x_axis.append(l[0])
y_axis.append(l[1])
f1.close()
from matplotlib import pyplot as plt
plt.plot(x_axis, y_axis)
plt.title('Order tensor diagonal elements')
plt.xlabel('Atom')
plt.ylabel('S')
plt.show()
f1 = open('ord_end.xvg', 'r')
x_axis = []
y_axis = []
for line in f1:
if line[0] == ' ':
l = line.strip().split()
x_axis.append(l[0])
y_axis.append(l[1])
f1.close()
from matplotlib import pyplot as plt
plt.plot(x_axis, y_axis)
plt.title('Order tensor diagonal elements')
plt.xlabel('Atom')
plt.ylabel('S')
plt.show()