BLAST
Задание 1. Нахождение гомологов белка BAB98887.1 в БД Swissprot
С помощью сервиса BLAST были найдены возможные гомологи белка. Для моего белка нашлось всего лишь 33 последовательности, гомология которых была не так уж очевидна, поэтому решено было взять белок из резерва. Все параметры я оставила по умолчанию, кроме количества результатов поиска (было выбрано максимально возможное значение - 20000). Результат выдал 71 белок, Evalue которых меньше 10. Полученную таблицу можно скачать здесь.
Описание параметров
| Enter Query Sequence | Вводится или сразу последовательность белка, гомологов которого надо найти, или его AC. Можно сразу загрузить файл или две и больше последовательности. Также есть возможность указать координаты участка белка (начиная с 1 до длины белка) в том случае, если мы ищем гомологов не ко всей последовательности. |
|---|---|
| Choose Search Set | Тут можно выбрать банк последовательностей, в котором мы хотим искать гомологи. Чем больше банк, тем больше гипотетических гомологов будет выдавать BLAST. Например, доступны такие банки, как Refseq, Swissprot, PDB. Также можно исключить любые таксоны из поиска или наоборот ограничить поиск в пределах царства, типа и т.д. |
| Max target sequences | Есть возможность ограничить количество результатов, чтобы выдавалось определенное число находок с наименьшим Evalue. Максимальное число находок - 20000 |
| Short queries | Так как для коротких последовательностей обычный алгоритм не подходит (потому что, например, если это глицин богатая последовательность, то будет множество сходных участков повторяющихся глицинов у негомологичных белков). Если поставить галочку, то алгоритм будет использовать автоматические параметры для коротких входящих последовательностей. |
| Expect threshold | Ограничение результатов по Evalue. Понятно, что чем больше этот параметр, тем больше результатов будет выдавать программа. Но чем выше Evalue, тем меньше вероятность того, что последовательность гомологична входящей, так как это показатель того, наколько вероятно получение выравнивания с таким же или больше весом последовательности из банка случайных последовательностей. Хотя нет точного порога, но последовательность, для которой Evalue меньше 0,001, часто оказывается действительно гомологичной. |
| Word size | Длина слова. Чем больше длина слова, тем быстрее работает BLAST, тем меньше последовательностей будет выдано, а значит есть вероятность пропустить гомолога. По умолчанию стоит длина слова 3, но возможно выбрать 2 или 6. BLAST разбивает последовательность на участки заданного числа, далее сопоставляет эти фрагменты с участками последовательностей из банка, составляя таблицу, в которой указывает последовательность, содержащую данное слово, и координаты в ней. Из таблицы программа берет подходящие последовательности и выравнивает с ними исходную. |
| Scoring Parameters | В этом блоке можно выбрать матрицу. Например, матрицы BLOSUM62 (стоит по умолчанию) и PHAT отличаются исходными данными, поступающими на обработку. 62 у BLOSUM62 означает порог кластеризации (не будут находиться две последовательности с процентом схожести больше 62 в блоке выравнивания). Тут же можно выбрать штрафы за выравнивание. |
| Filters and Masking | Нужно, в основном, для коротких последовательностей. Белки малой сложности (например, содержащие в большом количестве одну аминокислоту) имеют множество сходных участков с негомологичными беклами. Поэтому если поставить галочку напротив Low complexity regions, то алгоритм будет понижать ценность участков с повторяющейся аминокислотой и повышать ценность соответствия у двух последовательностей редких аминокислот. |
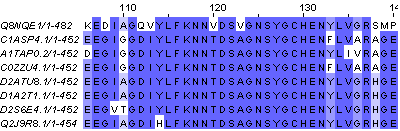
Из таблицы в основном выбирались белки с хорошим процентом покрытия (больше 70%) и из разных организмов. Так было выбрано 9 белков (+1 исходный в файле) и для них построено выравнивание с помощью программы Jalview командой muscle with defaults. Были удалены две последние последовательности, которые выбивались из общей картины. Гомологичность оставшихся не подвергается сомнению (окончательное выравнивание можно посмотреть здесь). Для подтверждения гомологичности можно посмотреть на позиции 115-132 на рисунке 1. Этот участок начинается с лизина и заканчивается аспарагином. Он не содержит гэпов, в нем практически все колонки консервативны, а значит он полностью подходит под критерии. Стоит отметить, что таких блоков в выравнивании не мало, что свидетельствует о высокой консервативности этих белков.
Задание 2. Построение карты локального сходства белков F4RBD6_MELLP и A0A0M2LYI0_9MICO.
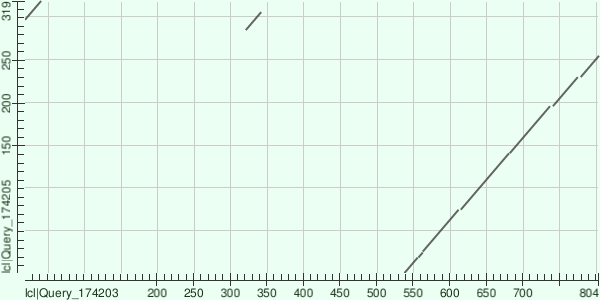
Для построения карты был выбран белок F4RBD6_MELLP (неизвестный белок из организма базидиального гриба) и A0A0M2LYI0_9MICO (7,8-dihydroneopterin aldolase из Leucobacter sp.). Карта построена с помощью BLAST и представлена на рисунке 2.
Задание 3. Изменение параметров BLAST.
Чтобы изучить, как от изменения параметров BLAST зависит количество и характеристика результатов, была выбрана полностью выдуманная последовательность:
MAYTHEFRCEBEWITHYULUKENEDESNTSIMPLYWALKINTMRDRTHEWINTERISCMINGALANNISTERALWAYSPAYSHISDEBTSSTARWARSRINGSGAMETHRNSИзменение банка данных
Банк: Swiss-Prot
Количество находок: 11
Банк: Protein Reference Sequences
Количество находок: 2
Банк: PDB
Количество находок: 7
Результаты мне показались странными, потому что я считала, что база данных Swiss-Prot самая маленькая, но именно в ней BLAST нашел больше всего гипотетических гомологов моему несуществующему белку, причем белки разные, процент покрытия около 40, а наименьшее значение Evalue=0,003.
Добавление таксонов
Ограничение на таксоны: нет
Количество находок: 15
Ограничение на таксоны: Metazoa
Количество находок: 12
Ограничение на таксоны: Bacteria
Количество находок: 5
Evalue было изменено на 15, чтобы получить больше находок, а банк во всех трех случаях был выбран Swiss-Prot. Опять же неожиданные для меня результаты, так как я думала, что у бактерий будет больше гипотетически гомологичных белков, чем у многоклеточных. Также стоит отметить, что поиск без ограничений дал меньше результатов, чем сумма находок в двух других поисках.
Expect threshold
Значение: 5
Количество находок: 3
Значение: 10
Количество находок: 11
Значение: 15
Количество находок: 15
Тут цифры говорят сами за себя, так как понятно, что чем больше возможное значение Evalue, тем больше рузльтатов будет выдавать программа. также наименьшее значение равно 0,003, что еще раз указывает на то, что данная последовательность никакой белок не кодирует.
Word size
Значение: 2
Количество находок: 11
Значение: 3
Количество находок: 11
Значение: 6
Количество находок: 0
К сожалению, из-за небольшой длины последовательности и ее выдуманности, результаты оказались не такими интересными, как ожидалось, потому что при W=2 и 3 количество найденных последовательностей одинаково, а при 6 их вообще нет. Но даже так понятна закономерность, что чем больше значение W, тем меньше будет гипотетичсеких гомологов. И хотя скорость выдачи результата будет выше, есть опасность пропустить гомологичный белок. Поэтому как компромисс по умолчанию стоит значение W=3.
Gap Costs
Значение: Existence: 13, extension: 1
Количество находок: 6
Значение: Existence: 6, extension: 2
Количество находок: 6
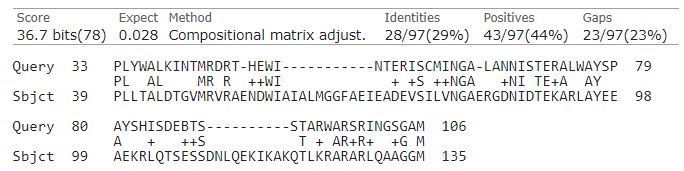
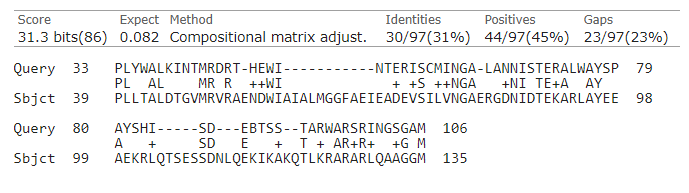
Чтобы лучше понять разницу в качестве значения были выбраны два крайних случая: когда за первый гэп штраф 13, а за каждый последующий 1; за первый - 6, а за каждый следующий -2. И хотя количество полученных последовательностей одинаково, Evalue, Score и сами белки в каждом случае разные. У первого белка в первом случае Evalue ниже, чем во втором. В его выравнивании видно, что в одном и том же участке последовательности, когда за первый гэп штраф 13, индель один(рисунок 3), а во втором случае их три (рисунок 4).