Поиск по сходству (BLAST)
Задание 1. Определение таксономии и функции нуклеотидной поседовательности
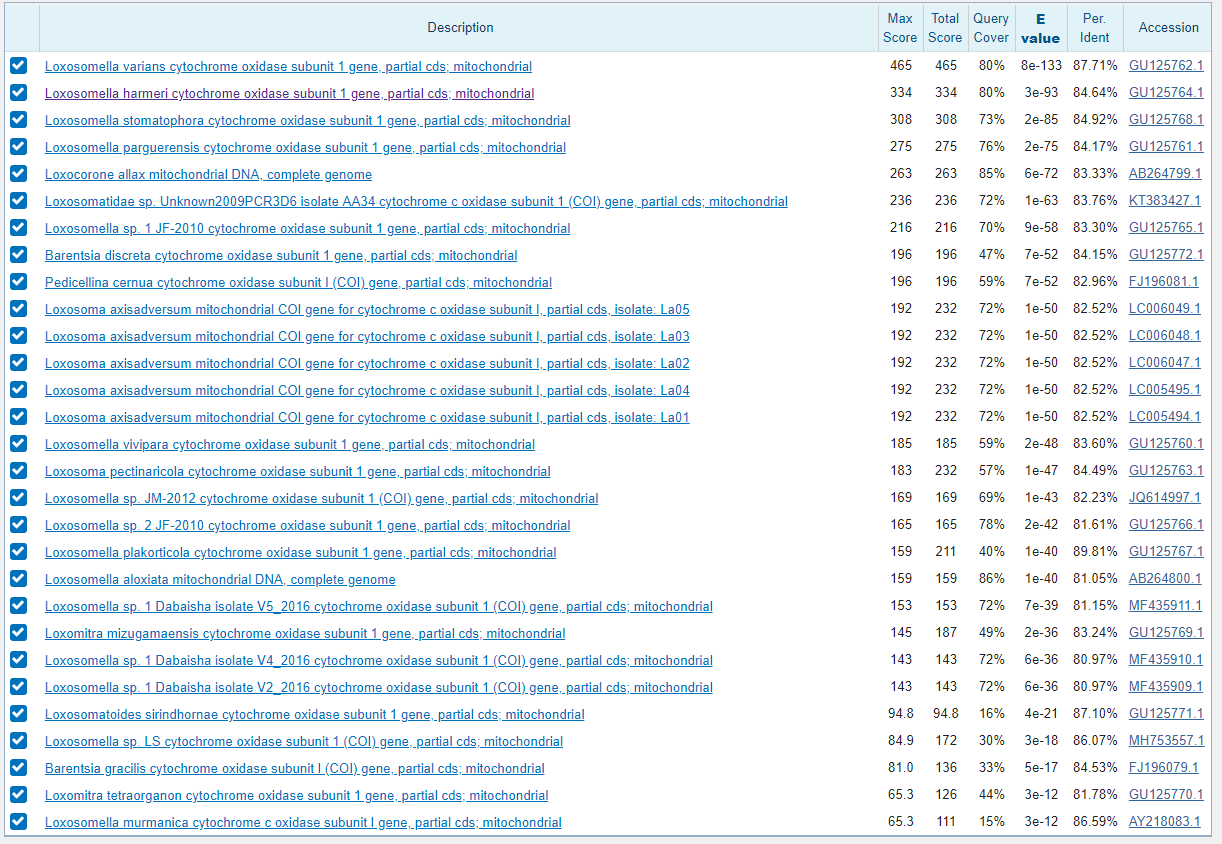
С помощью blastn на сайте NCBI был произведен поиск возможных гомологичных последовательностей. Все параметры я оставила без изменений. Все найденные последовательности кодировали митохондриальный ген белка субъединицы 1 цитохром с-оксидазы. Этот белок присутствует во внутренней мембране митохондрий всех эукариот и катализирует конечный этап переноса электронов на кислород в процессе окислительного фосфорилирования. Для определения таксономии гена были выбраны первые 7 находок, которые были наилучшими по показателям Evalue, веса, процента идентичности и покрытия. Находки отличались уже по типу: 4 из них были из Nemertea и 3 из Entprocta. На рисунке 1 видны результаты blastn.
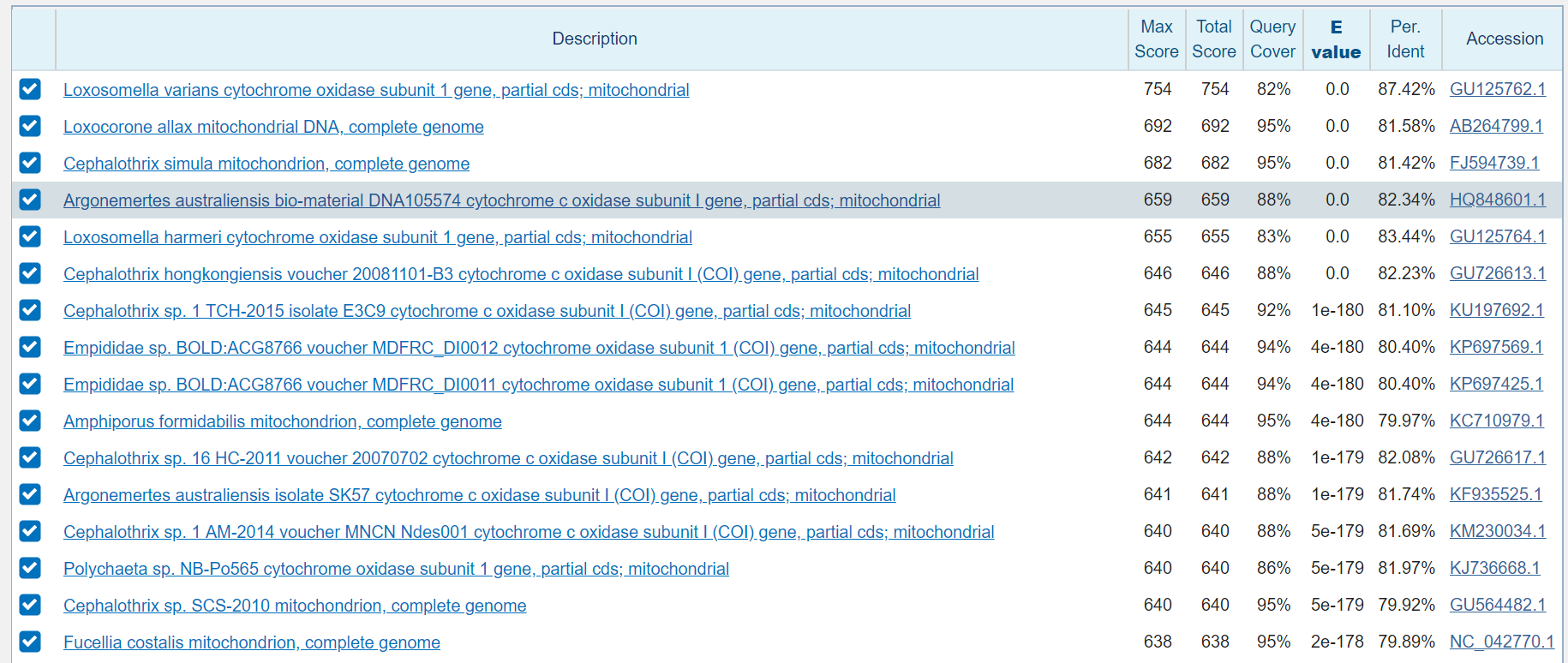
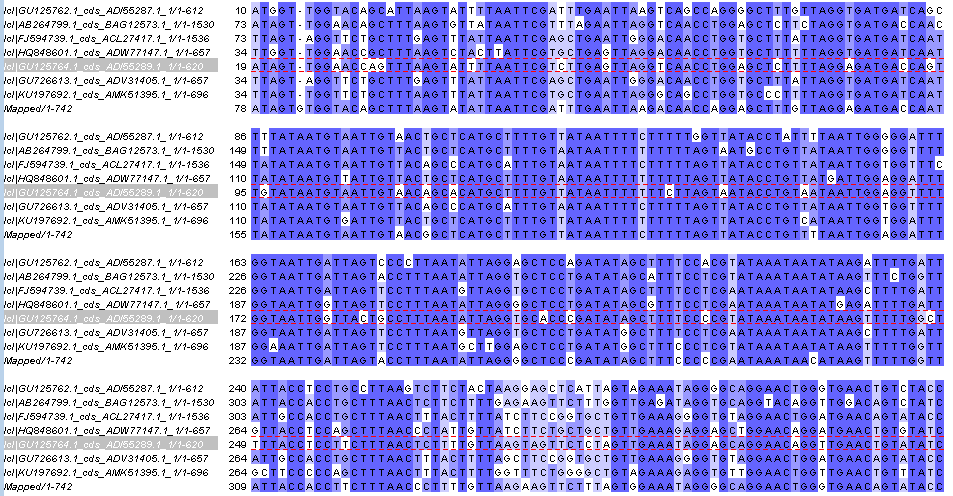
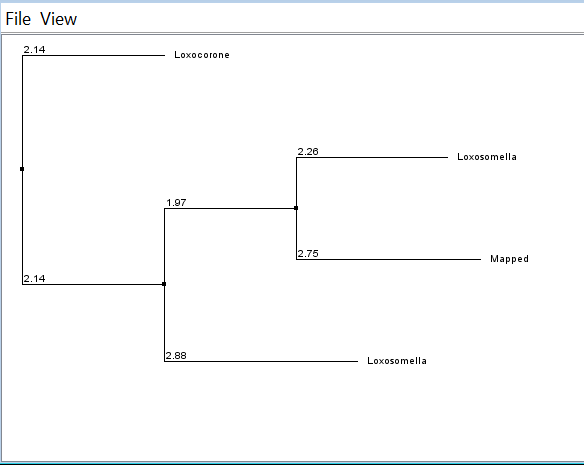
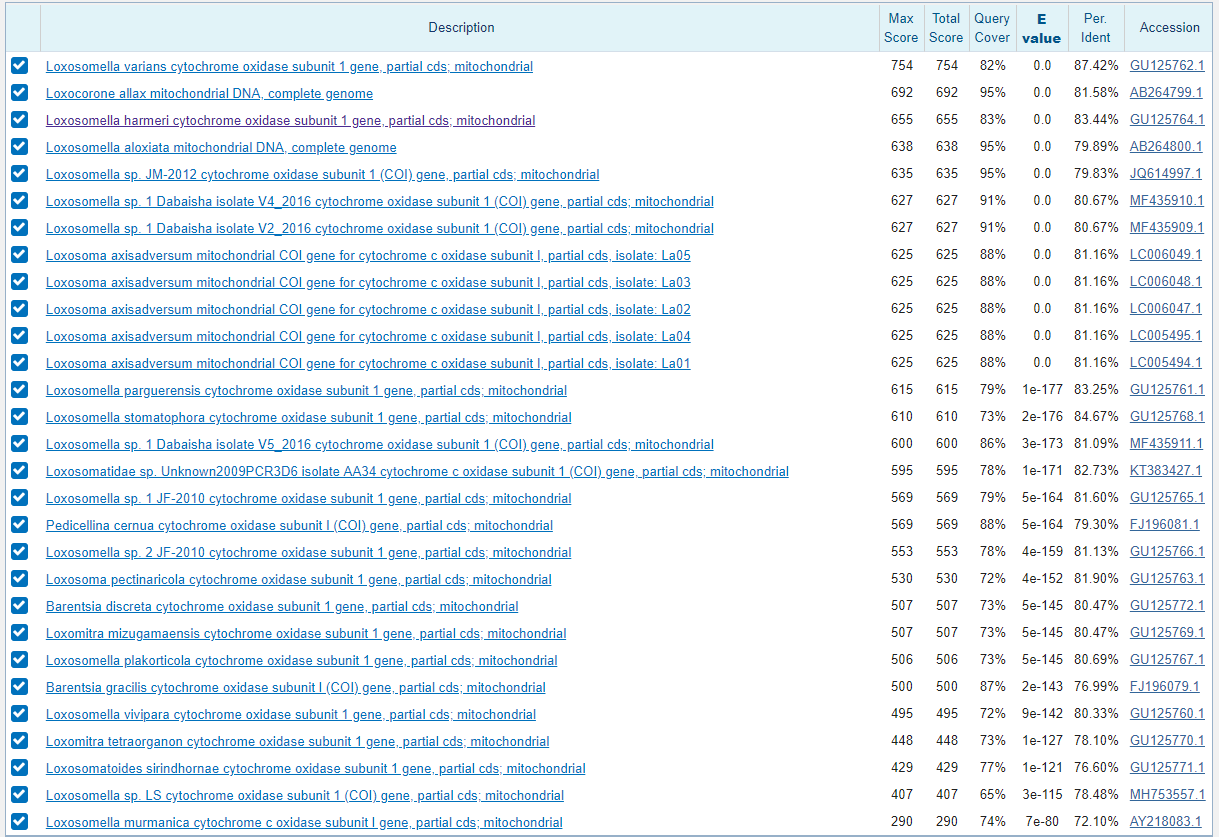
Далее последовательности каждой находки соответствующие по локальному выравниванию моему гену были выравнены вместе с ним множественным выравниванием в программе Jalview (рисунок 2). На этом рисунке первая, вторая и пятая последовательности относятся к одному типу. Поэтому можно сделать вывод, что моя последовательность из организма, принадлежащего типу Entoprocta. Точно также был определен род (рисунок 3) - Loxosomella. На рисунке 4 можно видеть дерево, которое только подтверждает правильность выбранного рода.

Задание 2. Сравнение списка находок нуклеотидных последовательностей тремя разными вариантами blast
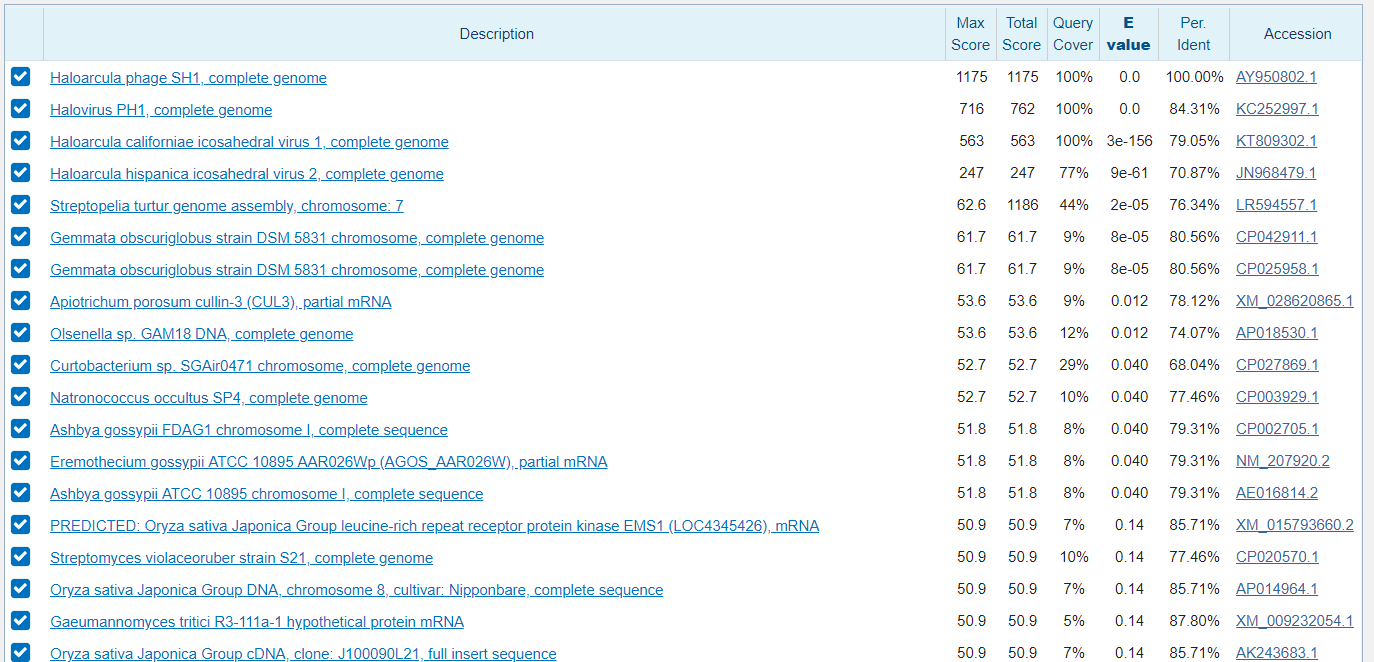
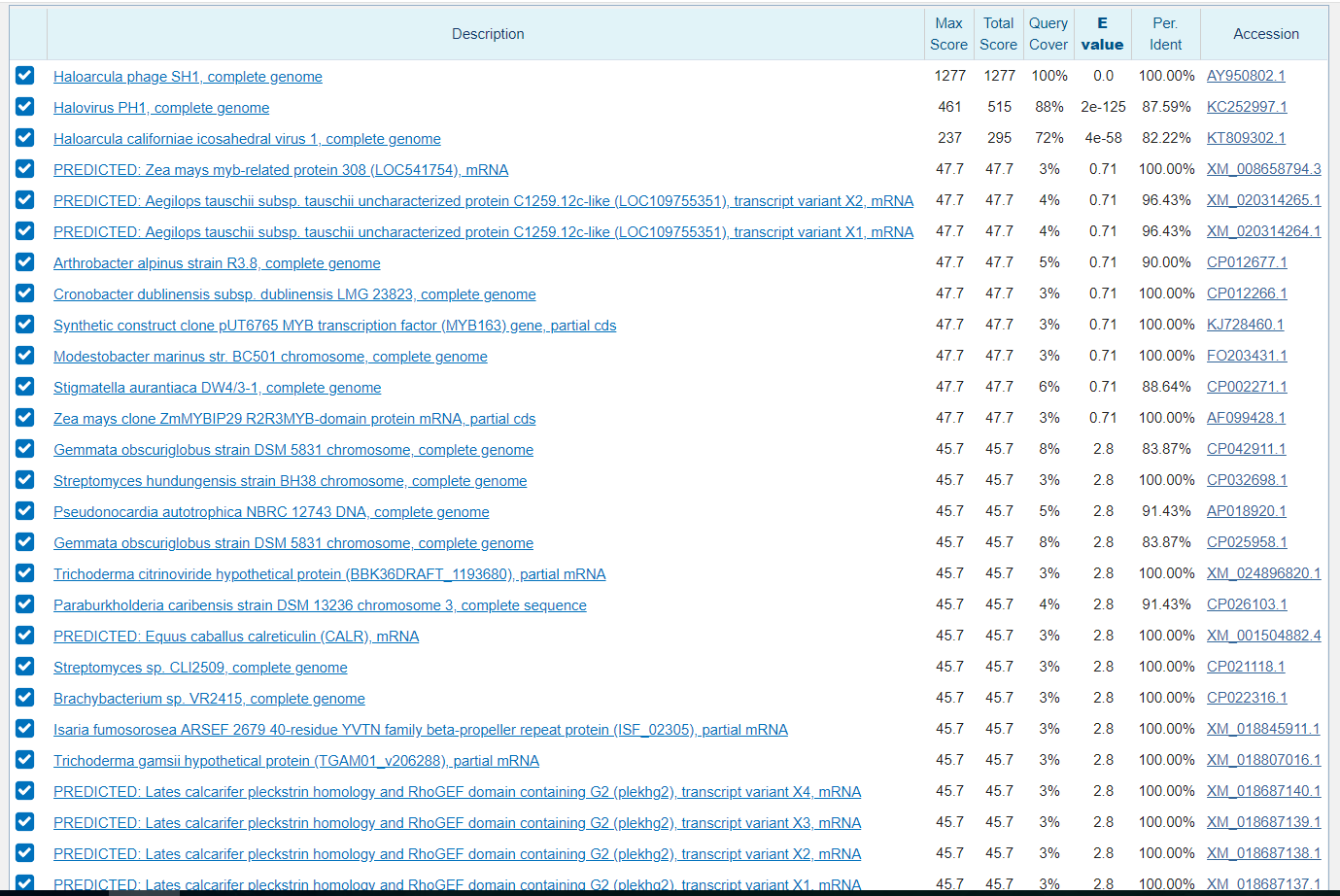
Для начала сравнивали по гену из задания 1. Без ограничений на организмы получилось слишком много результатов, поэтому было решено ограничить по типу Entoprocta. Evalue для всех трех поисков было выбрано 0,001. Вначале поиск произведен алгоритмом megablast c его стандартными значениями, потом blastn с параметрами по умолчанию и третий поиск - blastn с измененными параметрами поиска. Неудивительно, что алгоритмом megablast нашлось меньше всего последовательностей, так как значение слова у него 28, что обеспечивает быстроту поиска, но при этом существует возможность пропуска гомологичной последовательности. Находки двух других алгоритмов не отличаются и включают все находки первого поиска, но их порядок отличается, так как в результате изменения параметров изменяется вес локальных выравниваний, Evalue и другие значения. Находок со значением Evalue=0 стандартного алгоритма blastn - 12, некоторые из них имеют в первом алгоритме значение Evalue больше. Показатели находок последнего алгоритма самые плохие из-за больших значений за несоответствие и маленьких за совпадение нуклеотидов. Например, процент покрытия у последней находки на рисунке 7 15%, тогда как у той же самой находки на рисунке 6 процент покрытия 74 и Evalue намного ниже.
| Алгоритм | Word size | Match/Mismatch scores | Gap Costs | Количество находок гена цитохрома | Количество находок гена вируса |
|---|---|---|---|---|---|
| megablast | 28 | 1, -2 | Linear | 6 | 2 |
| blastn | 11 | 2, -3 | 5, 2 | 29 | 225 |
| blastn с измененными параметрами | 7 | 1, -4 | 1, 2 | 29 | 51 |
Тоже самое было проделано для гена длиной примерно 600 нуклеотидов белка ORF 20 вируса Haloarcula phage SH1. В данном случае megablast нашел две подходящие последовательности, хотя никаких ограничений не было, что еще раз говорит о том, что этот алгоритм нужно использовать для нахождения только очень сходных последовательностей. Также в данном случае различалось количество находок двумя другими способами. Находки с плохим Evalue относились к бактериям и даже рыбам.

Задание 3. Проверка наличия гомологов трех белков в неаннотированном геноме
Выбранные белки: HSP71_YEAST, EIF3G_SCHPO и TBB_NEUCR. Последовательности данных белков были найдены на сайте Uniprot в поиске по названию белков. Скачав файлы в формате fasta, c помощью локального BLAST+ на kodomo и генома из файла X5.fasta как базы для каждого белка были найдены гомологи. Таблицы ниже получены с помощью команды tblastn -query EIF3G_SCHPO.fasta -db X5.fasta -outfmt 7 > EIF3G_SCHPO.txt.
HSP71_YEAST - белок теплового шока, шаперон, который связывает развернутые полипептидные цепи, предотвращая возможную агрегацию, и освобождает их в цикле связывания и последующего гидролиза АТФ. Функции Hsp70 связаны с наличием в его структуре двух доменов. С-терминальный пептид - связывающий домен, который не участвует в регуляции специфичности связывания с пептидами. N-терминальный АТФазный домен, который может находиться в двух разных состояниях: АДФ- и АТФ-связанном, стабилизация которых зависит от взаимодействия с другими регуляторными белками и шаперонами.
По таблице сразу у двух скэффолдов (199, 96, 423) хорошие показатели, потому что длина самого белка 642, так что процент покрытия больше 90 %. Ответ положительный.
query id subject id % id length mismat gap q. start q. end s.start s. end Evalue bit score sp|P10591|HSP71_YEAST scaffold-199 78.98 609 125 2 2 607 1109256 1107430 0.0 920 sp|P10591|HSP71_YEAST scaffold-199 55.56 27 12 0 82 108 1110027 1109947 0.002 40.8 sp|P10591|HSP71_YEAST scaffold-96 66.06 607 201 4 3 606 89928 91742 0.0 744 sp|P10591|HSP71_YEAST scaffold-423 65.73 607 203 4 3 606 1313216 1311402 0.0 737 sp|P10591|HSP71_YEAST scaffold-423 37.34 391 228 5 4 378 781726 782895 8e-64 232 sp|P10591|HSP71_YEAST unplaced-999 81.90 315 56 1 2 315 945 1 8e-171 540 sp|P10591|HSP71_YEAST unplaced-980 77.54 334 73 1 276 607 1 1002 9e-142 461 sp|P10591|HSP71_YEAST scaffold-157 46.77 402 196 6 216 607 165338 166519 1e-81 285 sp|P10591|HSP71_YEAST scaffold-157 54.42 215 93 3 5 217 164418 165053 5e-64 233 sp|P10591|HSP71_YEAST scaffold-157 29.03 310 193 5 61 343 219904 218975 5e-32 133 sp|P10591|HSP71_YEAST scaffold-693 46.77 402 196 6 216 607 1114528 1115709 2e-80 281 sp|P10591|HSP71_YEAST scaffold-693 54.42 215 93 3 5 217 1113608 1114243 1e-63 232 sp|P10591|HSP71_YEAST scaffold-693 27.79 331 212 5 61 364 1168679 1167687 2e-29 125 sp|P10591|HSP71_YEAST unplaced-804 71.50 193 53 1 417 607 17964 17386 2e-74 264 sp|P10591|HSP71_YEAST scaffold-499 71.50 193 53 1 417 607 3580 4158 6e-74 262 sp|P10591|HSP71_YEAST unplaced-959 37.34 391 228 5 4 378 9193 10362 1e-63 231 sp|P10591|HSP71_YEAST scaffold-469 76.92 104 24 0 437 540 2 313 5e-43 150 sp|P10591|HSP71_YEAST scaffold-418 76.92 104 24 0 437 540 312 1 5e-43 150 sp|P10591|HSP71_YEAST unplaced-113 68.97 87 26 1 9 94 1 261 1e-32 122 sp|P10591|HSP71_YEAST scaffold-138 57.75 71 28 1 539 607 249 37 3e-17 78.6 sp|P10591|HSP71_YEAST scaffold-61 57.75 71 28 1 539 607 5 217 3e-17 78.6 sp|P10591|HSP71_YEAST unplaced-721 68.97 29 9 0 579 607 272 186 2e-05 43.9
EIF3G_SCHPO - фактор инициации трансляции у эукариот, поддерживает свободное состояние малой суъединицы, препятствуя преждевременной ассоциации большой и малой субъединицы, а также стимулирует связывание мRNA и Met-tRNA с 40S субъединицей. В данном случае, все параметры сходства у первых двух находок, включая процент покрытия (длина исходного белка - 282), кроме процента идентичности, свидетельствуют о гомологии, поэтому скорее всего, гомологичны не все белки, а домены.
query id subject id % id length mismat gap q. start q. end s.start s. end Evalue bit score sp|P78795|EIF3G_SCHPO scaffold-20 37.98 287 146 10 20 279 8619 7774 2e-21 95.5 sp|P78795|EIF3G_SCHPO scaffold-444 37.28 287 148 8 20 279 817421 816576 2e-20 92.0 sp|P78795|EIF3G_SCHPO scaffold-444 37.21 43 27 0 236 278 189369 189497 0.78 30.8 sp|P78795|EIF3G_SCHPO scaffold-444 28.79 66 45 1 201 264 188955 189152 5.8 28.1 sp|P78795|EIF3G_SCHPO scaffold-170 31.48 54 37 0 225 278 220797 220636 0.002 38.9 sp|P78795|EIF3G_SCHPO scaffold-17 39.62 53 32 0 226 278 2050544 2050702 0.008 37.4 sp|P78795|EIF3G_SCHPO scaffold-17 39.62 53 32 0 226 278 183975 184133 0.023 35.8 sp|P78795|EIF3G_SCHPO scaffold-17 37.50 40 25 0 225 264 1688251 1688132 0.063 34.3 sp|P78795|EIF3G_SCHPO scaffold-17 37.21 43 27 0 236 278 914230 914358 0.78 30.8 sp|P78795|EIF3G_SCHPO scaffold-17 30.30 66 44 1 201 264 913816 914013 4.4 28.5 sp|P78795|EIF3G_SCHPO scaffold-199 35.29 51 33 0 225 275 253346 253194 0.010 37.0 sp|P78795|EIF3G_SCHPO scaffold-212 40.91 66 39 0 200 265 296156 296353 0.023 35.8 sp|P78795|EIF3G_SCHPO scaffold-105 37.50 40 25 0 225 264 330352 330471 0.052 34.7 sp|P78795|EIF3G_SCHPO scaffold-96 31.15 61 39 1 225 282 197429 197611 0.12 33.5 sp|P78795|EIF3G_SCHPO scaffold-423 31.15 61 39 1 225 282 1206586 1206404 0.17 33.1 sp|P78795|EIF3G_SCHPO scaffold-287 41.67 60 32 1 195 251 56264 56443 0.30 32.3 sp|P78795|EIF3G_SCHPO scaffold-22 34.09 88 58 0 195 282 120213 119950 0.68 31.2 sp|P78795|EIF3G_SCHPO scaffold-22 30.23 43 27 1 228 267 120375 120247 2.4 29.3 sp|P78795|EIF3G_SCHPO scaffold-57 30.23 43 27 1 228 267 11654 11526 2.1 29.6
Тубулин (TBB_NEUCR) является основной структурной единицей микротрубочек. Он представляет собой гетеродимер, состоящий из двух близких белков, а- и b-тубулина. Каждая молекула а- и b-тубулина связывает молекулу ГТФ. Строение гетеродимера тубулина показывает, что ГТФ, связанный с а-тубулином, расположен поблизости от места его контакта с b-тубулином и никогда не гидролизуется. При сборке микротрубочек ГТФ, связанный с β-субъединицей, гидролизуется до ГДФ. Этот гидролиз приводит к изменению конформации гетеродимера тубулина, что играет важную роль в динамических превращениях микротрубочек.
Белок в организме присутствует, так как все показатели у скэффолда-26 свидетельствуют о гомологичности. Длина самого белка - 447, поэтому процент покрытия составляет 86.
query id subject id % id length mismat gap q. start q. end s.start s. end Evalue bit score sp|P05220|TBB_NEUCR unplaced-665 81.56 450 61 1 1 428 7236 5887 0.0 742 sp|P05220|TBB_NEUCR scaffold-26 86.79 386 51 0 43 428 109811 108654 0.0 693 sp|P05220|TBB_NEUCR scaffold-26 66.67 51 17 0 1 51 110007 109855 4e-13 71.6 sp|P05220|TBB_NEUCR unplaced-5 39.91 441 252 3 1 431 3257 4570 1e-105 348 sp|P05220|TBB_NEUCR scaffold-57 40.14 441 251 3 1 431 88586 87273 1e-105 348 sp|P05220|TBB_NEUCR scaffold-423 28.79 389 236 8 80 428 190311 191474 6e-49 161 sp|P05220|TBB_NEUCR scaffold-423 27.50 80 40 1 9 70 189959 190198 6e-49 45.4 sp|P05220|TBB_NEUCR scaffold-423 76.92 13 3 0 2 14 189873 189911 6e-49 26.6 sp|P05220|TBB_NEUCR scaffold-423 27.34 384 237 8 80 422 1101339 1100191 2e-46 150 sp|P05220|TBB_NEUCR scaffold-423 33.33 63 34 1 16 70 1101622 1101434 2e-46 47.4 sp|P05220|TBB_NEUCR scaffold-423 76.92 13 3 0 2 14 1101728 1101690 2e-46 26.9
Задание 4. Поиск гена белка в одном из скэффолдов
С помощью команды infoseq X5.fasta -only -name -length -out был получен список скэффолдов с их длинами. Был выбран скэффолд-126, длина которого составляла 92370. Отдельно последовательность этого скэффолда получена в результате команды seqret X5.fasta:scaffold-126 -out scaffold-126.fasta. Здесь можно скачать последовательность скэффолда. Последовательность была загружена в blast и использовался алгоритм blastx, поиск осуществлялся по базе Refseq_protein и был ограничен по таксону Fungi. Результат можно увидеть на рисунке 11. Из получившихся 97 находок первые пять были генами гипотетических белков, но значения идентичности (52%), Evalue и покрытия шестой находки позволяют говорить о гомологичности. А значит в скэффолде есть ген, кодирующий белок синтазы хоризмата. Выравнивание этой находки можно увидеть на рисунке 12.