Паралоги, визуализация
Командой makeblasb -dbtype prot -in proteomes.fasta были проиндексированы все последовательности выбранных белков. Далее проводился поиск программой blastp для нахождения достоверных гомологов белка CLPX_ECOLI.
blastp -query clpx_ecoli.fasta -db proteomes -evalue 0.001 -out homology.txt -outfmt 7
Выходной файл содержит все найденные белки с показателями выравнивания в виде таблицы. Его можно скачать здесь. Из этой таблицы были взяты идентификаторы белков, чтобы по ним скачать их последовательности с сайта Uniprot. Их последовательность лежит в этом файле, а после выравнивание (программой muscle) полученный файл можно скачать здесь.
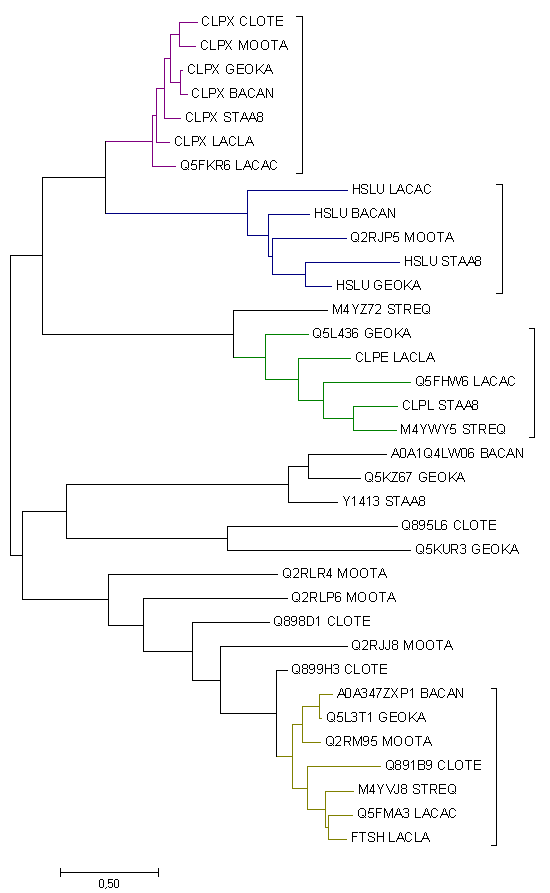
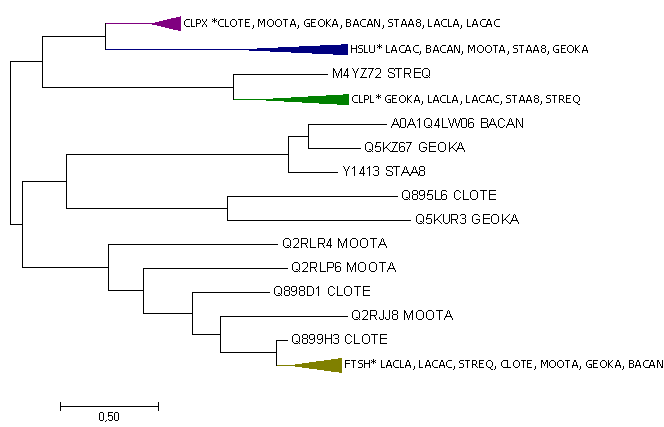
Дерево строилось программой Mega7 методом максимального правдоподобия. Полученный результат можно увидеть на изображении ниже или скачать скобочную формулу, где уже разные ортологичные группы (всего 4 группы, где больше трех ортологичных белков) покрашены в разные цвета. Таким образом, ортологами являются следующие пары: CLPX_BACAN и CLPX_GEOKA, HSLU_LACAC и HSLU_STAA8, Q5L3T1_GEOKA и Q5FMA3_LACAC. Тогда как паралогами являются: CLPX_BACAN и HSLU_BACAN, FTSH_LACLA и CLPX_LACLA, Q2RLR4_MOOTA и Q2RJP5_MOOTA.