Валидация
В данном практикуме изучались показатели качества PCA модели и способы их улучшения.
Задание 1.
Для анализа показателей качества модели, полученной рентгеноструктурным анализом, была выбрана структура 5Е89, представляющая домен, узнающий углеводы, галектина-3 человека. Галектины - это семейство белков, способных связывать бета-галактозиды определенным доменом (CRD), благодаря этому они сшивают гликопротеины, содержащие D-галактопиранозид, и тем самым влияют на локализацию и транспортировку гликопротеинов. Было показано, что галектин-3 играет важную роль во многих биологических процессах, таких как эмбриогенез, пролиферация, апоптоз, фибриогенез и воспаление. Так как было показано, что антагонисты галектина-3 имеют терапевтический эффект при воспалении, то велись разработки молекулы антагониста с высоким сродством к белку. Для одной из таких молекул (3-fluophenyl-1,2,3-triazolyl thiodigalactoside) была получена структура в комплексе с галектином-3 рентгеноструктурным анализом для определения мест связывания и выяснения причин высокой аффинности. Оригинальная статья.
Проанализируем основные показатели полученной модели, описанные на странице PDB. Разрешение данной структуры составляет 1,5 А с полнотой данных 92,1 %, что говорит о приемлемом восстановлении.
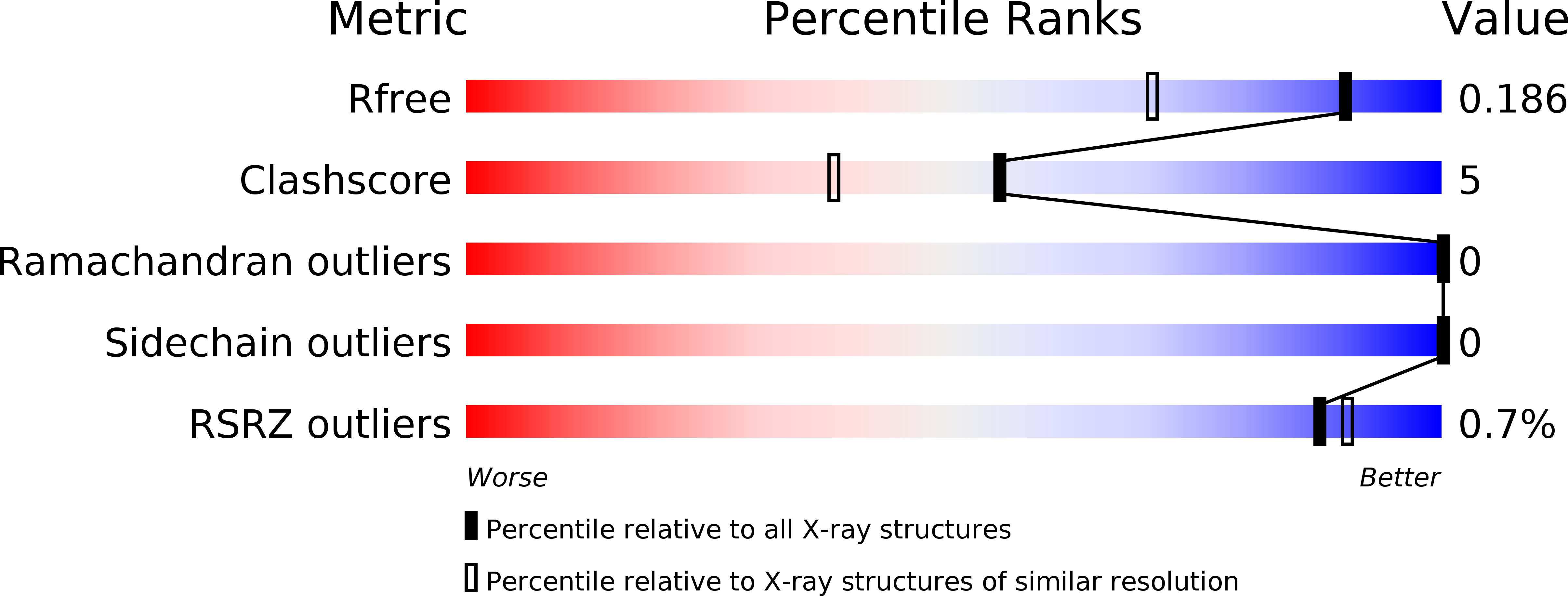
R=0,168; R_free=0,190. R-фактор, который характеризует соответствие между полученной моделью и структурными данными, лучше, чем у большинства структур с таким же разрешением. Переоптимизации не произошло, так как разница между R фактором и R_free составляет 5,1 %.
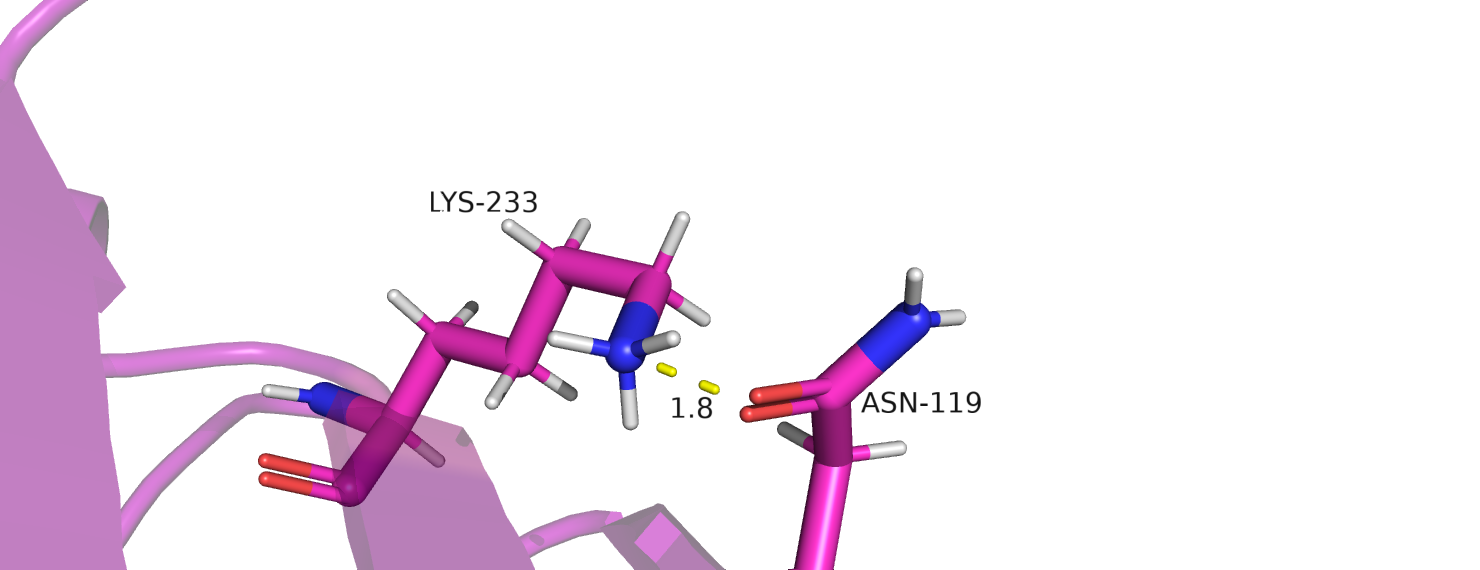
Clashscore, характеризующий количество атомов, у которых перекрываются Ван-дер-Ваальсовы радиусы, является единственным показателем, который у данной структуры хуже, чем в среднем по всем образцам с таким же разрешением. После добавления водородов таких перекрываний оказалось 13, причем пять из них это перекрывания между атомами Asn-119 и Lys-233, а четыре между аминокислотными остатками белка и молекулами воды.
Маргиналов по картам Рамачандрана (соответствие углов ψ и φ остова белка разрешенным значениям) и по торсионным углам боковых радикалов не оказалось, хотя три аминокислотных остатка находятся не в предпочитаемой, а в разрешенной зоне карт Рамачандрана.
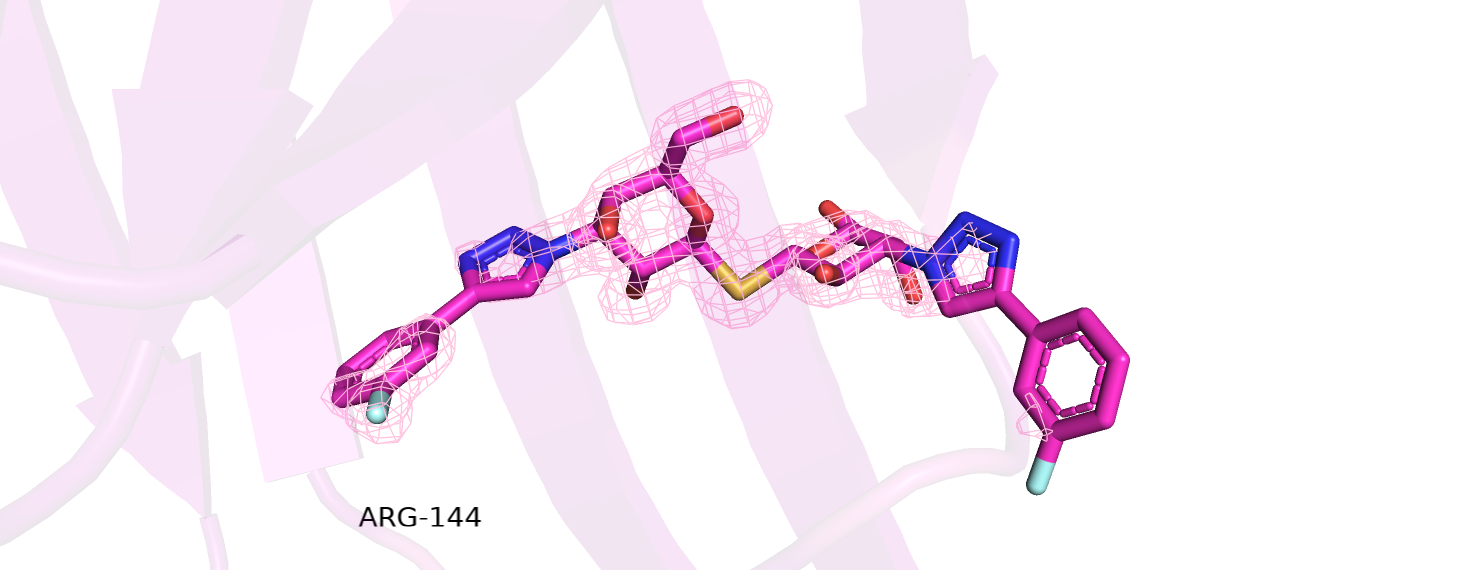
При наложении электронной плотности на структуры белка (рисунок 2) кажется, что в целом все атомы вписываются в электронную плотность.
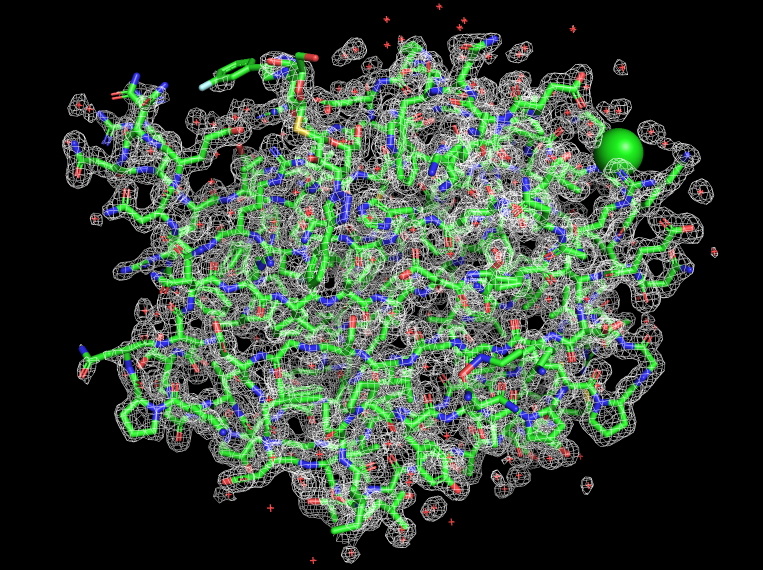
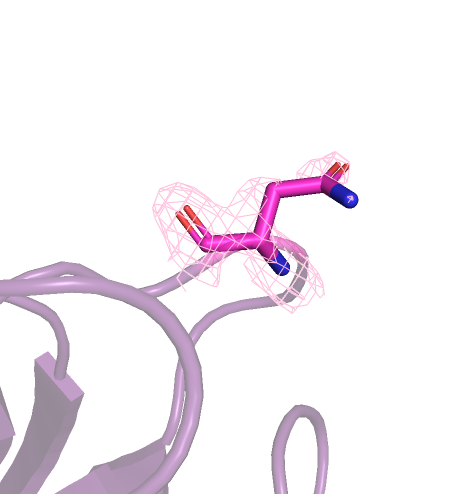
На самом деле так и есть. RSRZ, сравнивающий экспериментальную плотность и электронную плотность модели, больше 2 только у одного аминокислотного остатка (Asn-141, RSRZ=2,6), что говорит о том, что координаты атомов хорошо вписываются в электронную плотность. У Asn-141 местоположение атома азота (ND2) никак не подтверждается электронной плотностью, хотя остальные атомы неплохо вписываются в нее.
Задание 2.
Для дополнительного анализа структуры был использован сервис MolProbity. После анализа геометрии была получена таблица с основными характеристиками, которые совпадают с данными из файла о валидации с PDB, так как также не обнаружено маргиналов по боковым радикалам, по карте Рамачандрана или остатков с запрещенными углами и длинами связей (рисунок 4).
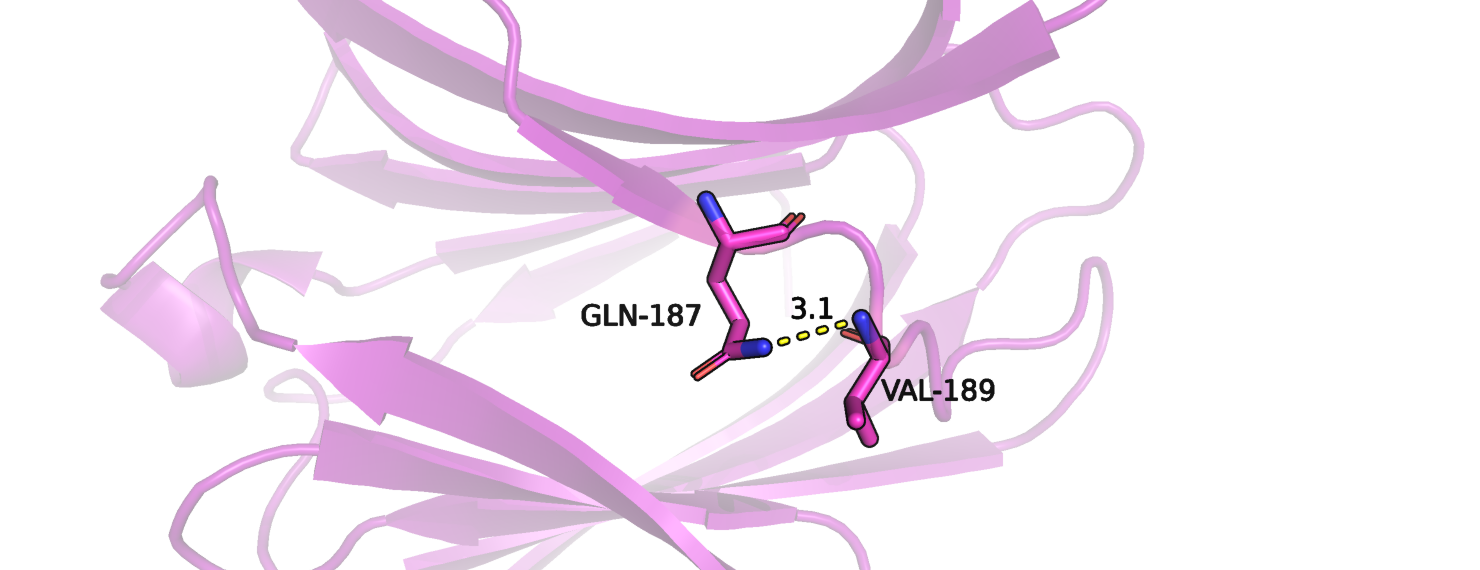
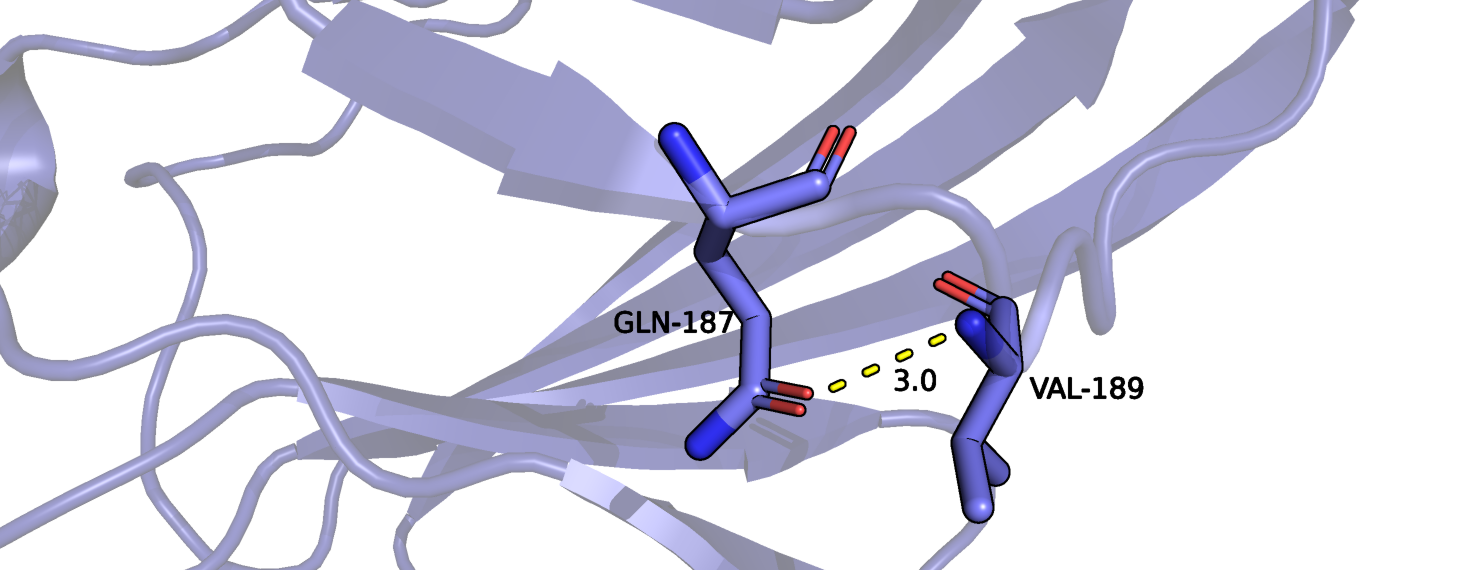
В проанализированной модели не было атомов водорода, поэтому после их добавления была вновь проверена геометрия полученной структуры. Один из остатков (Gln-187) было предложено развернуть. И действительно по рисунку 5 видно, что в изначальном состоянии NH2 группа глутамина сближена с NH группой валина, а при повороте становится возможным образование водородной связи между кислороом глутамина и NH группой валина.
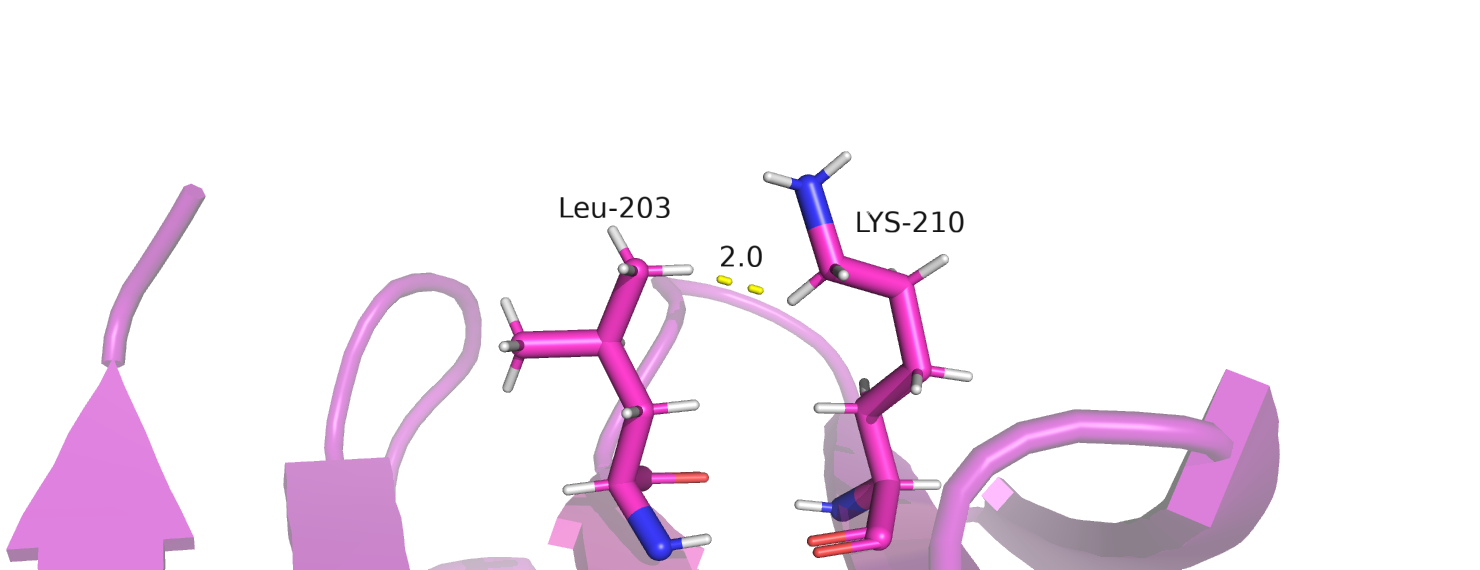
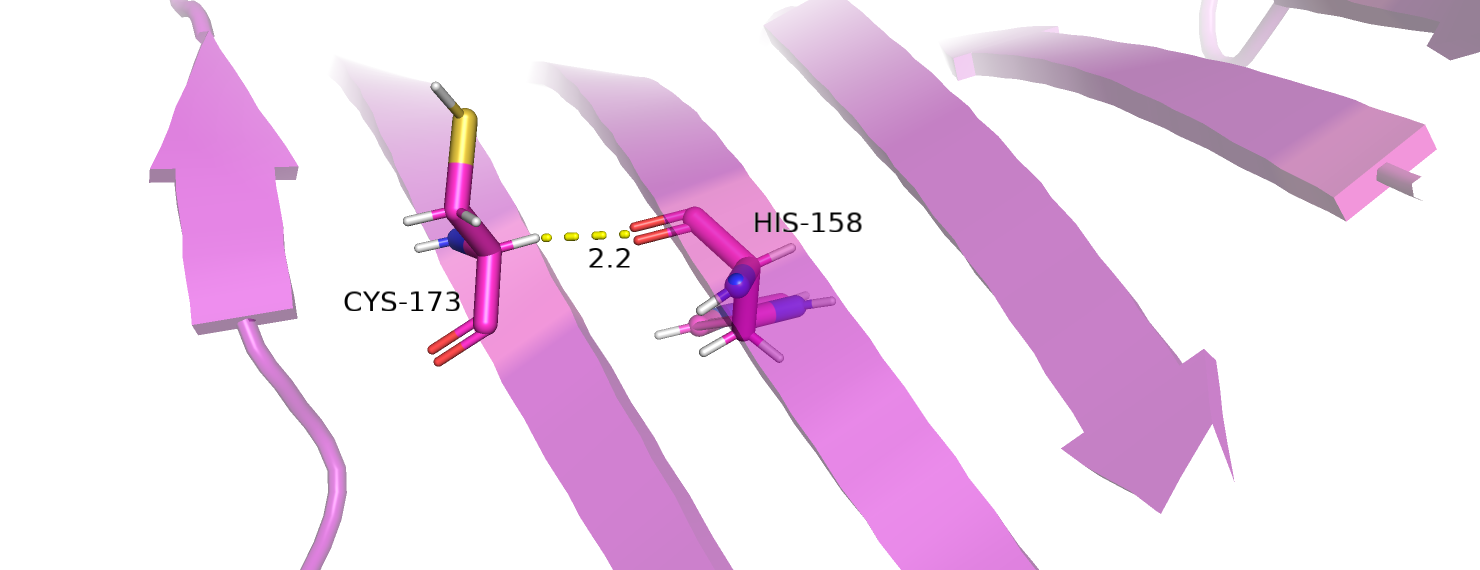
После анализа всех взаимодействий между атомами появился параметр Clashscore. Всего было выявлено три перекрывания, показанных на рисунке 6, которые также присутствовали в PDB отчете о валидации.
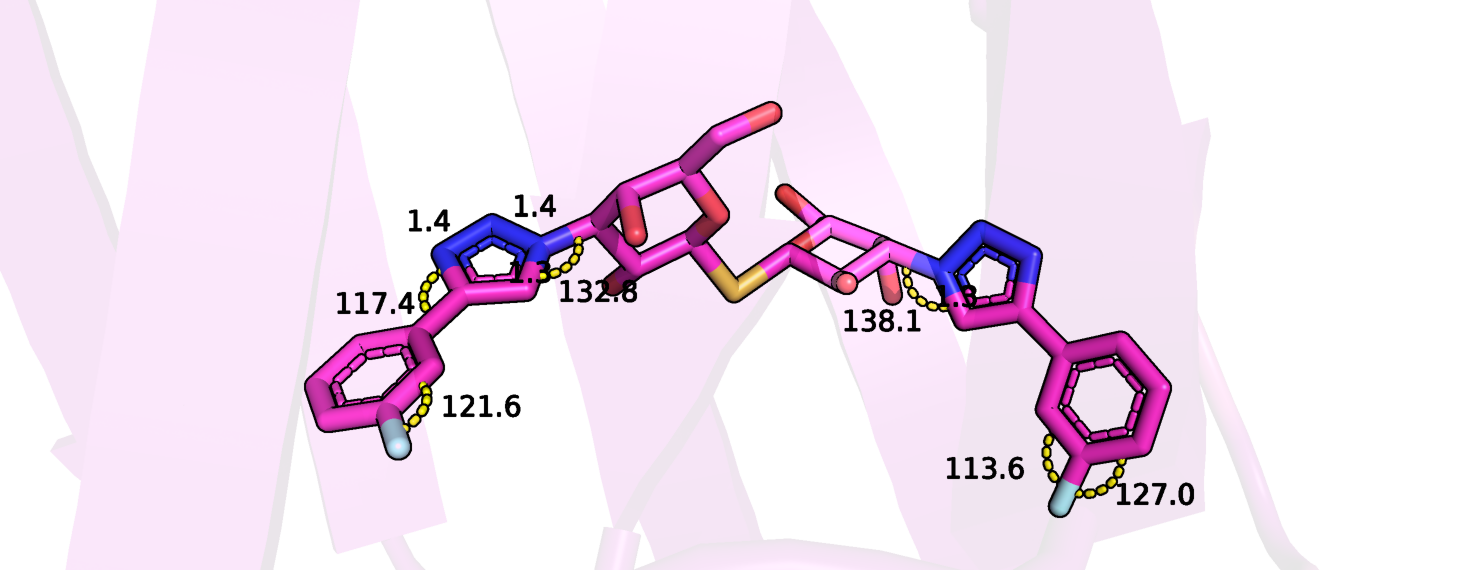
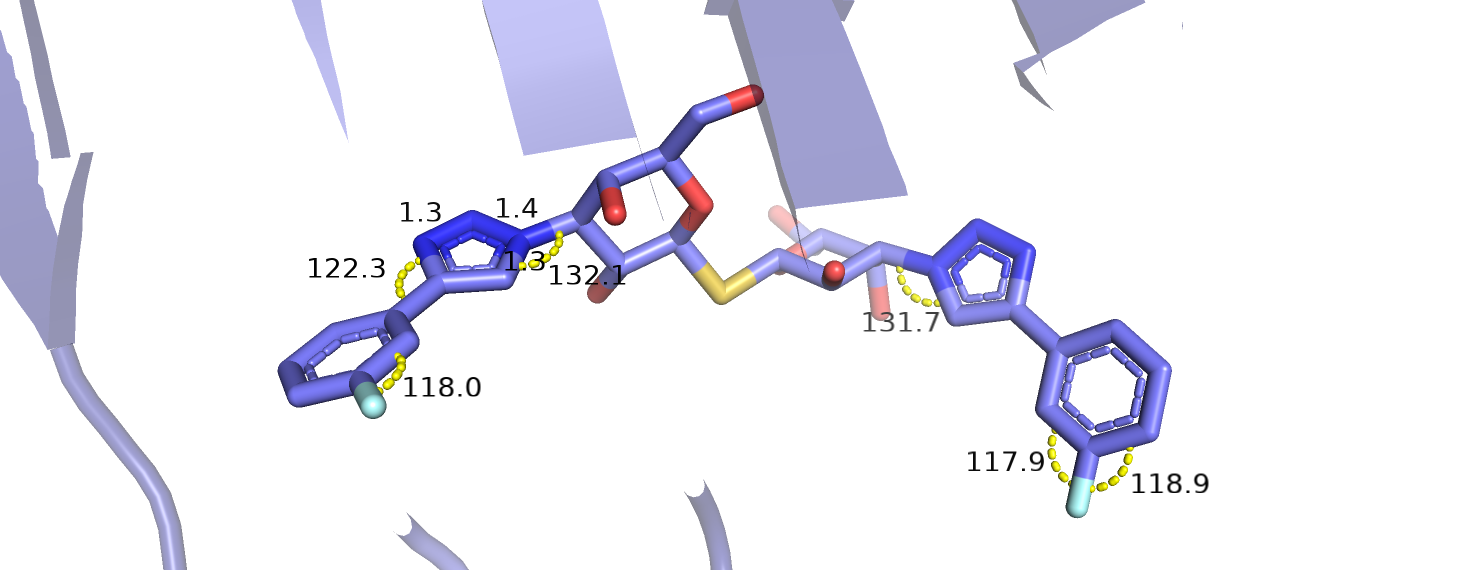
Несоответствие в полученных значениях углов и длин связей обнаружилось в молекуле лиганда (3- fluophenyl-1,2,3-triazolyl thiodigalactoside inhibitor). На рисунке 7 показаны те некоторые углы и стороны, величины которых отклоняются от идеальных (для длин связи отклонение +-0,5 А, для углов - +-10°).
Задание 3.
Как уже было сказано выше, данная модель хорошо соответствует полученной электронной плотности и имеет мало отклонений в значениях углов и длин связей от идеальных. Так как данная трехмерная структура была получена для изучения взаимодействия между лигандом и белком, то именно места их связывания должны обладать хорошим качеством, поэтому настораживают множественные отклонения в величинах углов и связей в молекуле лиганда, что скорее всего произошло из-за того, что электронная плотность покрывает не весь лиганд (рисунок 8). Не считая этого, данная структура прекрасно подходит для изучения свойств белка.
Задание 4.
PDB-REDO - сервис для улучшения геометрии структур, полученных рентгеноструктурным анализом. На рисунке 9 продемонстрированы показатели качества для исходной модели и измененной. Видно, что уменьшились значения R-фактора и R_free, что говорит о лучшем соответствии электронной плотности модели структурным факторам. Остальные показатели либо не изменились, либо стали хуже, хоть и ненамного.
Рисунок 10, изображающий карту Рамачандрана для исходной и измененной модели, также показывает, что отредактированная модель не сильно отличается от основной, во всяком случае по углам ψ и φ остова белка. Все аминокислотные остатки по-прежнему находятся в разрешенной зоне.
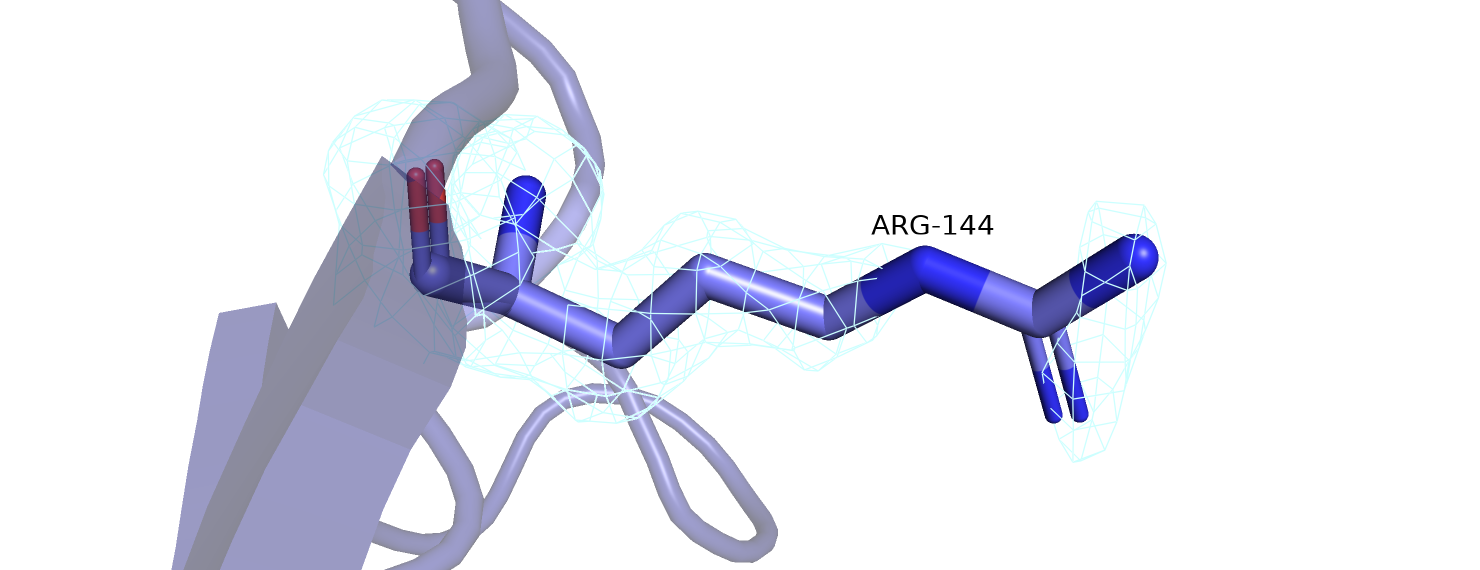
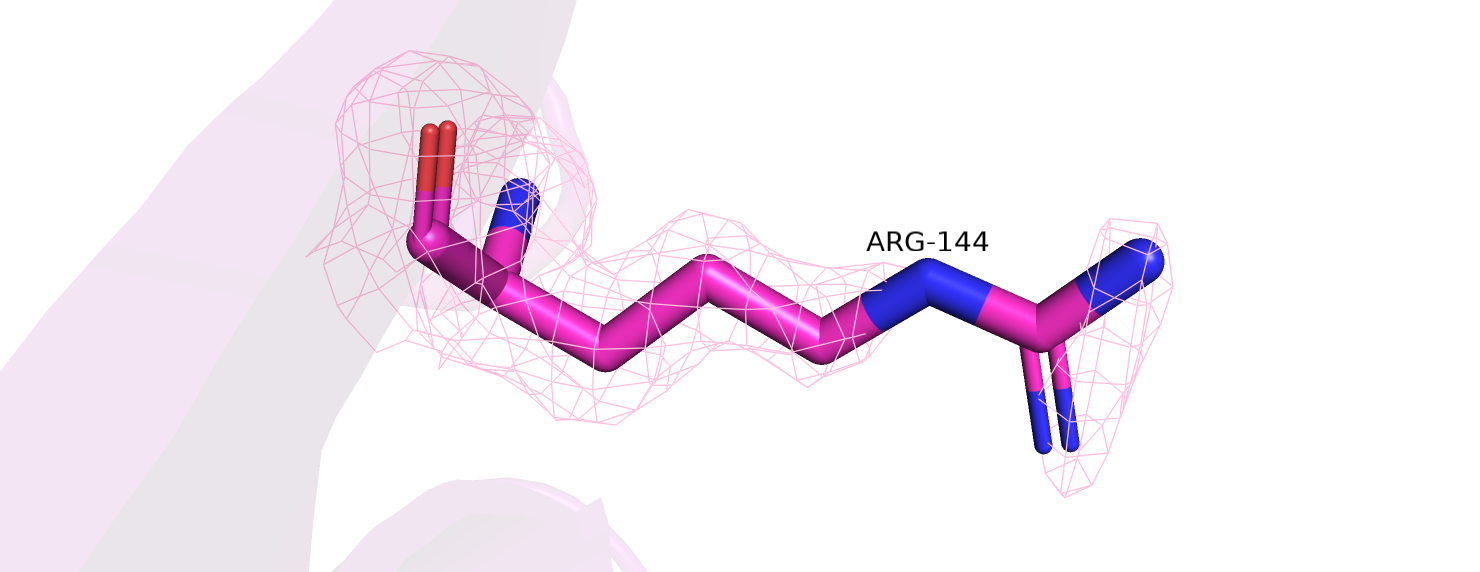
Изменилась вписываемость атомов в полученную электронную плотность. Для 25% аминокислотных остатков она значительно улучшилась (RSCC > 0,02), для остальных не стала значимо хуже. Но визуально эта разница не видна, так как даже по самому улучшенному аминокислотному остатку (Arg-144) не видно никаких отличий (рисунок 11).
Но есть и значимые изменения: так, Gln-187 был повернут и стало возможным образование водородной связи (рисунок 12).

Проанализируем измененную модель с помощью сервиса MolProbity.
Основные показатели не изменились, например, перекрывание для всех рассмотренных выше атомов сохранилось. Поменялось количество углов (2), значения которых не характерно для аминокислотных остатков. Рассмотрим один из них. Для аминокислотного остатка Lys-226 значение угла изменилось на 3°, но этого уже достаточно, чтобы такой угол рассматривался как недопустимый.
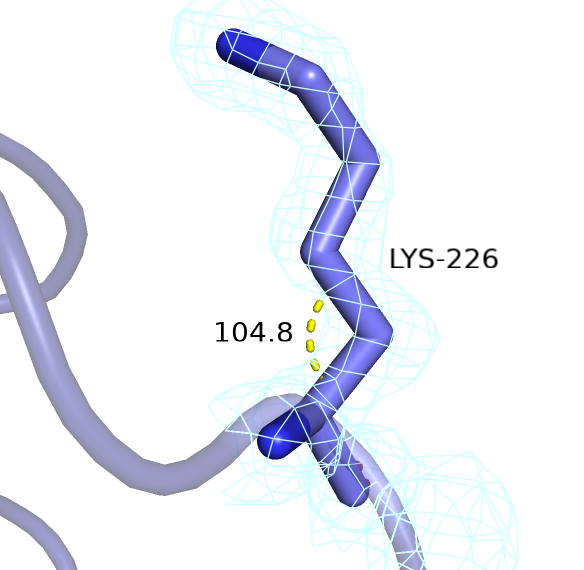
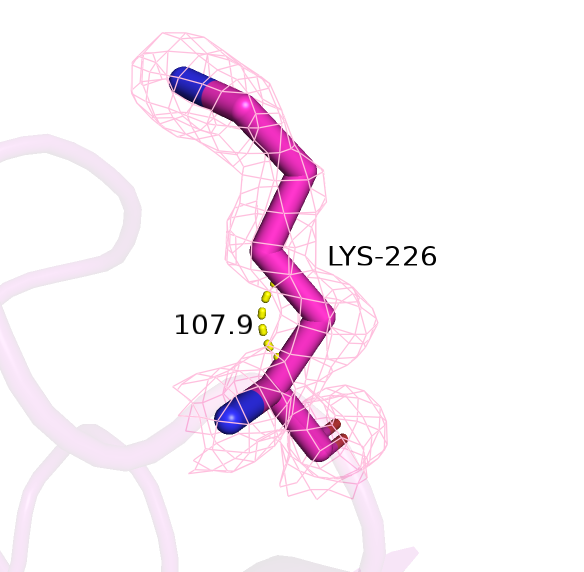
Рассмотрим изменения, произошедшие в лиганде (рисунок 15): хотя длины связей не изменились, но значения пяти углов стали близки к идеальным, что говорит о том, что структура лиганда действительно стала лучше.
Таким образом, изменения, внесенные PDB-REDO, кажутся незначительными, так как аутлайеров в исходной структуре и так не наблюдалось, но они все равно позволили в целом улучшить всю структуру и показатели ее качества.