Практикум 12. Алгоритмы и программы множественного выравнивания. Базы гомологичных доменов.
1. Сравнение выравниваний, выполненных разными программами
Таблица 1. Cравнение Muscle и Mafft
| Параметр | Muscle | Mafft |
|---|---|---|
| Длина выравнивания | 148 | 148 |
| Процент выровненных колонок | 97.30% | 97.30% |
| Блок 1 | (1-99) | (1-99) |
| Блок 2 | (103-122) | (103-122) |
Таблица 2. Cравнение Muscle и TCoffee
| Параметр | Muscle | TCoffee |
|---|---|---|
| Длина выравнивания | 148 | 147 |
| Процент выровненных колонок | 97.30% | 97.96% |
| Блок 1 | (1-99) | (1-99) |
| Блок 2 | (103-124) | (103-124) |
Таблица 3. Cравнение Mafft и TCoffee
| Параметр | Mafft | TCoffee |
|---|---|---|
| Длина выравнивания | 148 | 147 |
| Процент выровненных колонок | 97.30% | 97.96% |
| Блок 1 | (1-122) | (1-122) |
| Блок 2 | (131-148) | (130-147) |
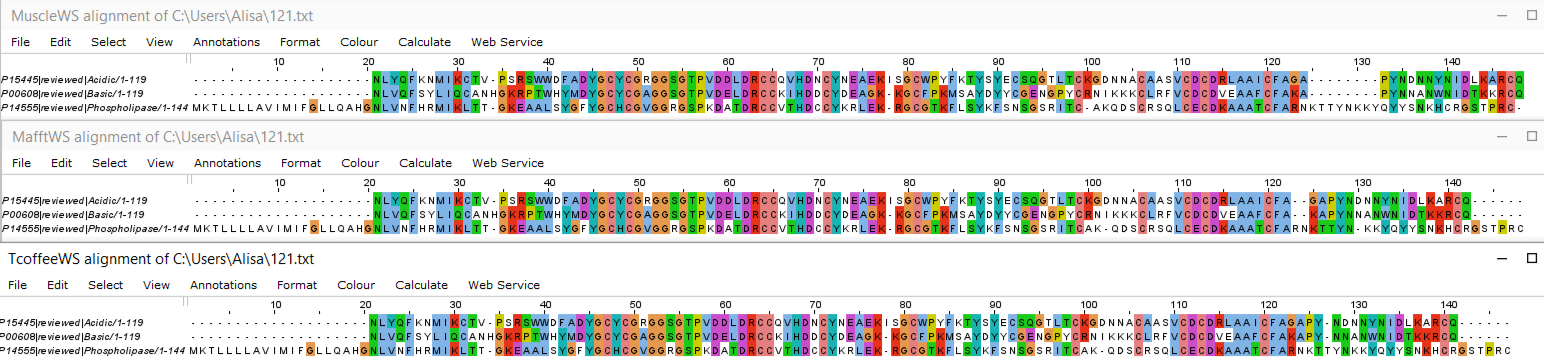
Можно заметить, что результаты выравнивания программ Mafft и TCoffee между собой похожи больше, чем другие возможные комбинации сравнения этих 3 программ, возможно, это потому что их алгоритмы работают сходным образом. Количество блоков во всех программах совпадет. С 1 по 99 позицию все 3 выравнивания совпадают полностью.
2.Выравнивание по совмещению структур
Я выбрала семейство фосфолипаз А2 (PF00068). Далее я выбрала 3 белка: фосфолипаза А2 из индийской кобры, фосфолипаза А2 из тигровой змей и человеческая ассоциированная с мембраной фосфолипаза А2.
Выравнивание, использующее совмещение пространственных структур, было выполнено на PDB с помощью встроенного инструмента Pairwise Structure Alignment. (Рис.1 и Рис.2). Также было проведено множественное выравнивание с помощью программы Muscle в Jalview. Ссылка на файл с проектом Jalview Jalview

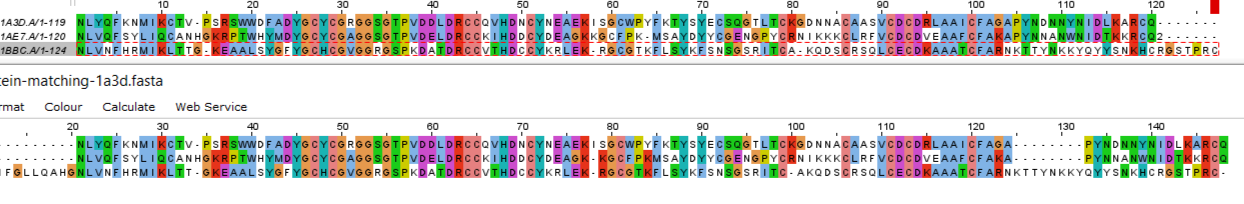
Эти выравнивания совпадют почти во всех позициях. Различаются выравнивания С-конца последовательностей. Видно, что в третей последовательности произошла вставка, возможно этот кусок не имеет какую-то важную структурную функцию (например, это выпетливание, а не фрагмент альфа-спирали) и поэтому в PDB - выравнивании этот фрагмент не выглядит как вставка.
3.Краткое описание программы Muscle
Этап 1.
Цель первого этапа - произвести множественное выравнивание, делая упор на скорость, а не на точность.
1.1 Расстояние в k-мерах вычисляется для каждой пары входных последовательностей, что дает матрицу расстояний D1.
1.2 Матрица D1 кластеризуется UPGMA, создавая бинарное дерево TREE1.
1.3 Прогрессивное выравнивание строится путем следования порядку ветвления TREE1.
На каждом листе профиль строится на основе входной последовательности.
Узлы в дереве посещаются в порядке: дочерние перед родительскими.
На каждом внутреннем узле создается попарное выравнивание двух дочерних профилей, дающее новый профиль,
который присваивается этому узлу. Это приводит к многократному выравниванию всех входных последовательностей MSA1.
Этап 2, улучшенная прогрессия .
Основным источником ошибок на черновой прогрессивной стадии является приблизительная мера расстояния в k-мерах,
что приводит к неоптимальному дереву. Поэтому MUSCLE повторно оценивает дерево, используя
расстояние Кимуры, которое является более точным, но требует выравнивания.
2.1 Расстояние Кимуры для каждой пары входных последовательностей вычисляется из MSA1, что дает матрицу расстояний D2.
2.2 Матрица D2 кластеризуется UPGMA, создавая бинарное дерево TREE2.
2.3 Последовательное выравнивание производится после TREE2 (аналогично 1.3),
что приводит к множественному выравниванию MSA2. Это оптимизировано путем вычисления выравниваний
только для поддеревьев, порядок ветвления которых изменился относительно TREE1.
Этап 3, Доработка.
3.1 Ребро выбирается из TREE2 (ребра посещаются в порядке уменьшения расстояния от корня).
3.2 TREE2 разделяется на два поддерева путем удаления ребра. Вычисляется профиль множественного выравнивания в каждом поддереве.
3.3 Новое множественное выравнивание производится путем повторного выравнивания двух профилей.
3.4 Если оценка SP (сумма баллов по парному выравниванию) улучшена, новое выравнивание сохраняется, в противном случае оно отбрасывается.
Шаги 3.1–3.4 повторяются до тех пор, пока не будет достигнута сходимость. Сходимость достигается, когда нет значительных изменений в выравнивании.