Практикум 10
Смысл этого практикума - подобрать пару нуклеотидных последовательностей, для которых карты локального сходства, построенные Megablast и BLASTN, заметно различаются.
Я выбрала 2 родственные бактерии Mycobacterium ulcerans (NZ_AP017624.1 - индентификатор хромосомы из refseq) и Mycobacterium tuberculosis (NC_000962.3). В качестве запроса в NCBI изпользововала видовое название бактерий и выбирала референсный геном, потом взяла идентификаторы хромосом для blast.
Результаты программ blastn (word size - 15; Рис.1) и megablast (word size - 28; Рис.2)
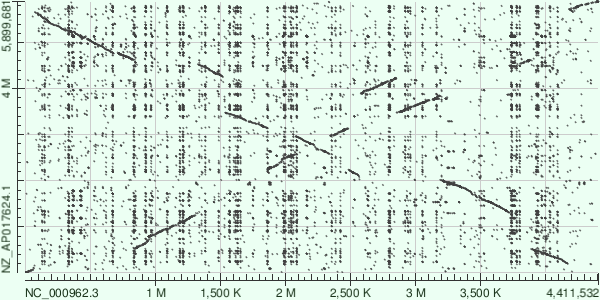
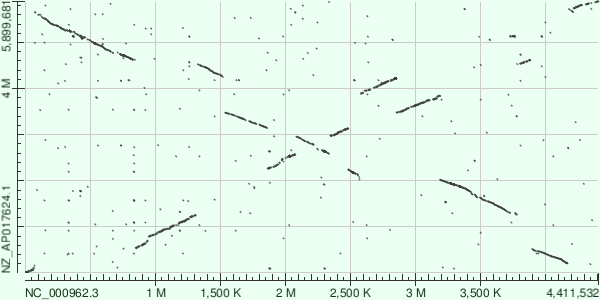
На dotplot BlastN больше мусора, так как word size меньше. Если сравнивать геномы бактерий, можно заметить многочисленные транслокации, причем некоторые кусочки еще и инвертировались.