Практикум 8
1. Ген, кодирующий δ-субъединицу АТФ-синтазы
В данном практикуме использовалась геномная сборка организма Acinonyx jubatus (гепард).
Последовательность белка, аннотированного как δ-субъединица АТФ-синтазы.
Текстовый поиск по файлу с белковыми последовательностями белков гепарда выявил запись белковой последовательности дельта-субъединицы АТФ-синтазы (XP_026905295.2).
| >XP_026905295.2 ATP synthase subunit delta, mitochondrial [Acinonyx jubatus] MLPVAVLRRPGLRCLVRQARAYAEAAAAPAPASGPGQMSFTFASPTQVFFNGANVRQVDVPTQTGAFGILAAHVPTLQVL RPGLVVVHAEDGTTSKYFVSSGSVTVNADSSVQLLAEEAVTLDMLDVGAAKVNLEKAQSELSGAADEASRAEIQIRIEAN |
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок.
NC_069383.1
Последовательность ДНК, непосредственно содержащая ген δ-субъединицы АТФ-синтазы.
2. Разные варианты BLAST для фрагмента ДНК
Так как исследуемый организм принадлежит к вторичноротым, в качестве отдаленного таксона сравнения было выбрано семейство пауков (Araneae). С помощью BLAST было проведено два поиска гомологичных последовательностей.
Для оценки консервативности белковой последовательности, полученной ранее, был проведен поиск гомологов с помощью алгоритма tblastn (сравнение белковой последовательности с нуклеотидной базой данных) в далеком семействе пауков (taxid:6893). В эту базу данных входит 4 геномные сборки.Длина слова - 5. Анализ выявил четыре совпадения с идентичностью 55-65%. Это говорит о высокой консервативность данной субъединицы АТФ-синтазы, так как мы ее нашли в далекой группе. Это можно объяснить необходимостью отсеивать мутации, так как фермент выполняет важную для жизнедеятельности функцию.
Мы поняли, что белковая последовательность достаточно консервативна. Интересно как эта консервативность проявляется на уровне гена. Проведем поиск гомологов с помощью blastn (поиск гомологов нуклеотидной последовательности в нуклеотидной базе данных). По моему запросу не нашлось ни одной статистически значимой находки, даже с разными параметрами. Это значит что гены сильно отличаются, возможно из-за отсутствия стабилизирующего отбора в белок-некодирующих частях гена.
3. Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу
В данном задании проводился поиск гомологов 16S и 23S рРНК E. coli в геноме эукариота — гепарда (Acinonyx jubatus). Эти рибосомные РНК, формирующие структурные элементы рибосомы, играют ключевые роли в синтезе белка: 16S рРНК участвует в связывании последовательности Шайна-Дальгарно в мРНК (малая субъединица), а 23S рРНК — в пептидилтрансферазной реакции (большая субъединица).
Геном эукариота был проиндексирован с помощью makeblastdb для последующего поиска нуклеотидных последовательностей. Программа создала локальную базу данных BLAST из файла FASTA-последовательностей (параметр -in), указав тип данных как нуклеотидный (-dbtype nucl).
makeblastdb -in GCF_027475565.1_VMU_Ajub_asm_v1.0_genomic.fna -dbtype nucl
После загрузки последовательностей рРНК E. coli (16S и 23S), был запущен алгоритм blastn для поиска гомологичных последовательностей (метод эффективен даже при поиске отдаленных гомологов). Далее были выполнены следующие команды:
blastn -task blastn -query 16.fna -db GCF_027475565.1_VMU_Ajub_asm_v1.0_genomic.fna -out res16.txt -outfmt 7
blastn -task blastn -query 23.fna -db GCF_027475565.1_VMU_Ajub_asm_v1.0_genomic.fna -out res23.txt -outfmt 7
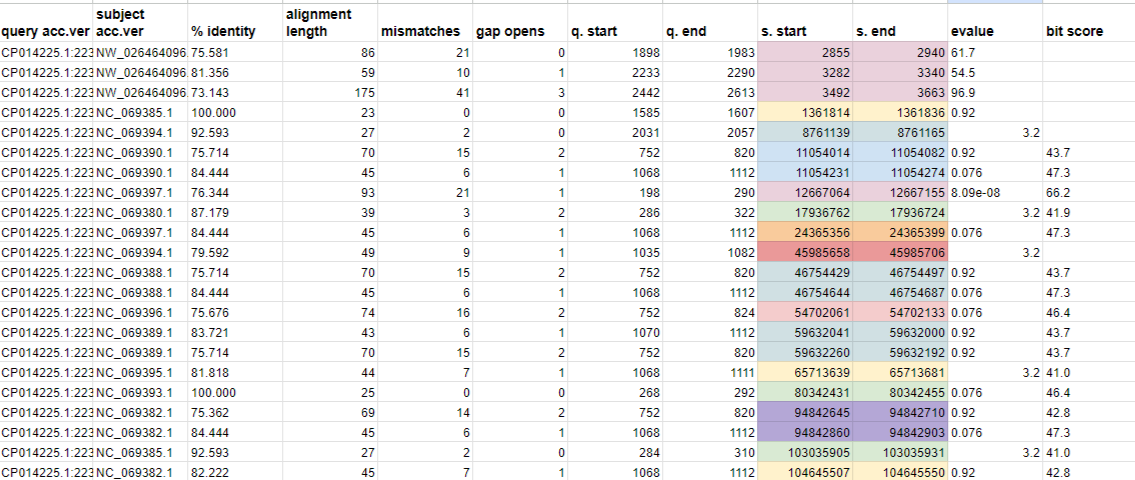
Для 16S было найдено 11 совпадений. На картинке я покрасила по группам находки с близкими коордианатами. Но хорошее значение e-value только у одной из них, поэтому я считаю, что нашелся только 1 гомолог 16S - ген 18S находится на хромосоме Е1 (NC_069397.1), что подтверждается данными о локализации последовтельностей рРНК.
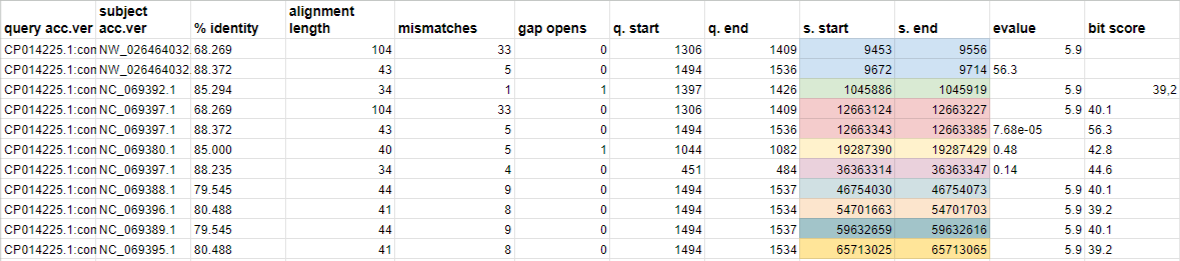
Для 23S было найдено 22 совпадения. Их я тоже раскрасила по координатам, что хорошо соотносится с разными хромасомами. Только одна находка имеет нормальное значение e-value, следовательно один гомолог, ген 28S тоже находится на хромосоме Е1 (NC_069397.1). В NCBI не указано расположение 28S рРНК. Ну это и логично, что 18S и 28S находятся на одной хромосоме, так как они транскрибируются единым предшественником рРНК.