Практикум 10. Мотивы в белках
1. Консервативные мотивы в выравнивании
Для выполнения задания было выбрано семейство белков Trp_DMAT (PF11991), которое представляет собой триптофан-диметилаллилтрансферазу, которая катализирует первую стадию биосинтеза алкалоидов спорыньи. Эти ферменты встречаются как у бактерий, так и у эукариот, имея типичную длину от 390 до 465 аминокислотных остатков. Анализируемое выравнивание SEED содержало 93 последовательностей, что соответствует заданным критериям (более 20, но менее 200 последовательностей). Согласно данным UniProt, этот домен присутствует примерно в 3000 белках, при этом в SwissProt аннотировано только 91 соответствующих белков, что также удовлетворяет условиям задания.
На рисунке 1 изображен фрагмент выравнивания, столбики покрашены по консервативности аминокислот, Above identity threshold 96%, видно 3 консервативные аминокислоты, паттерн в Jalview: R[LIVF]K[IVL]Y.
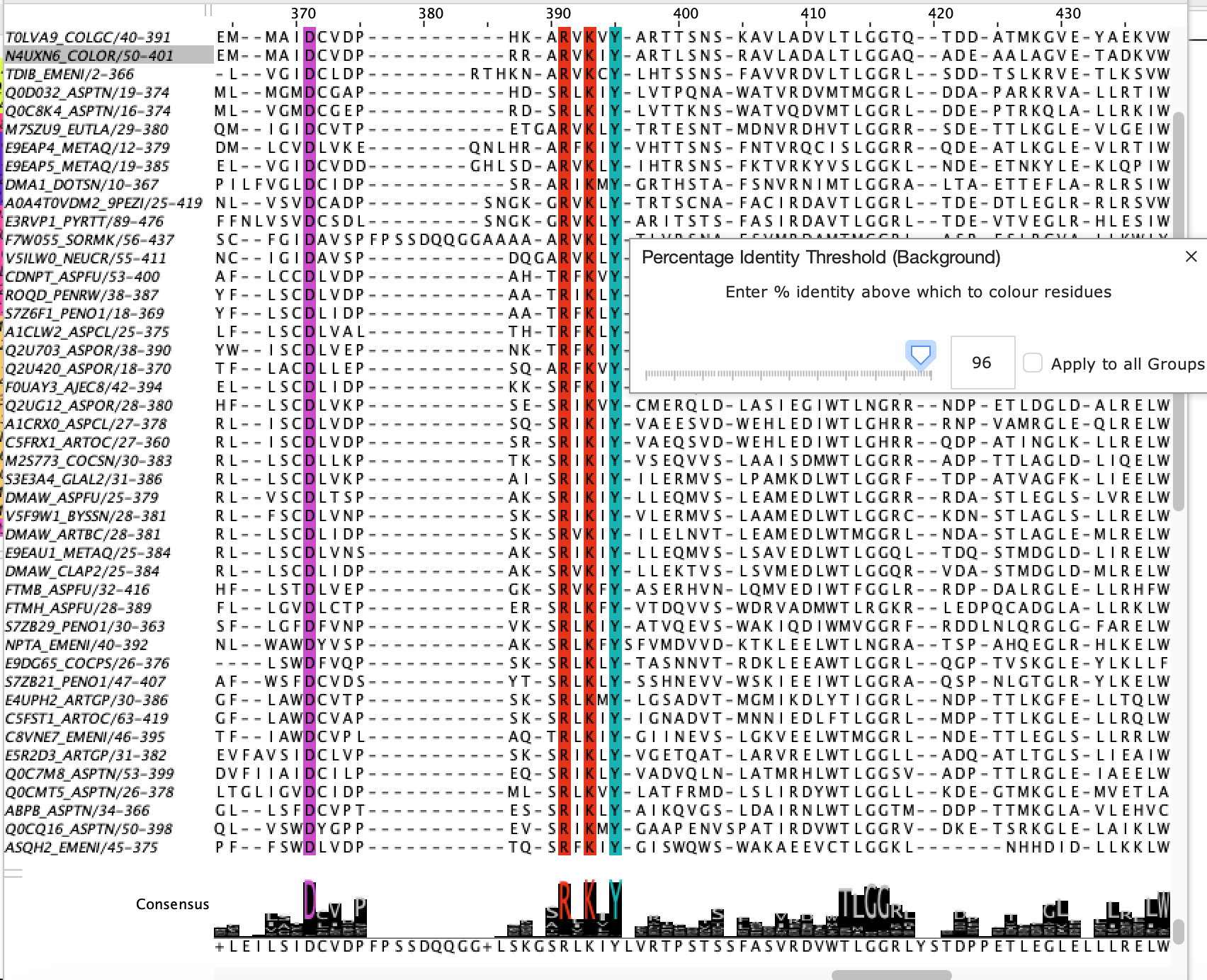
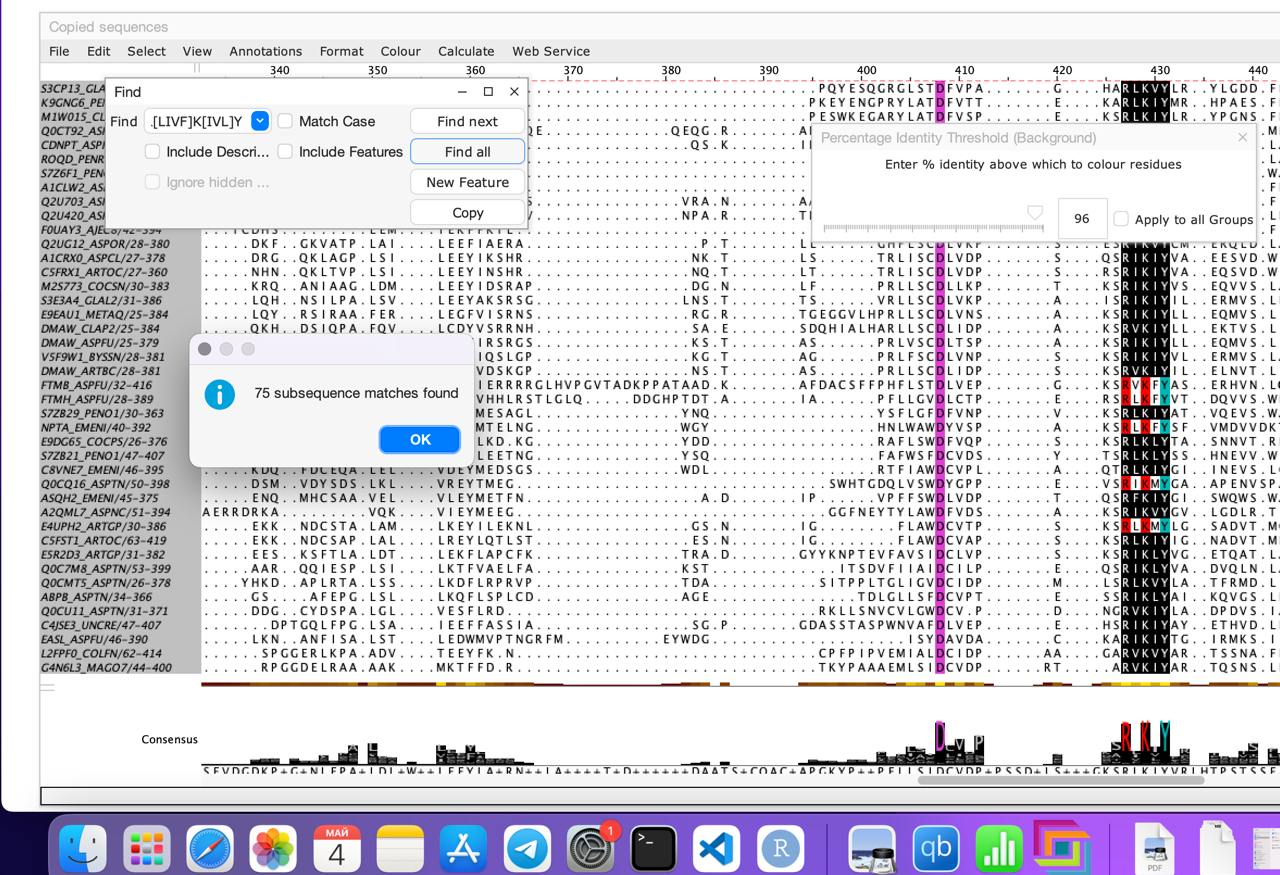
Выполним поиск по паттерну R[LIVF]K[IVL]Y во всем выравнивании. Было найдено 75 находок, все друг под другом (всего 93 последовательности).
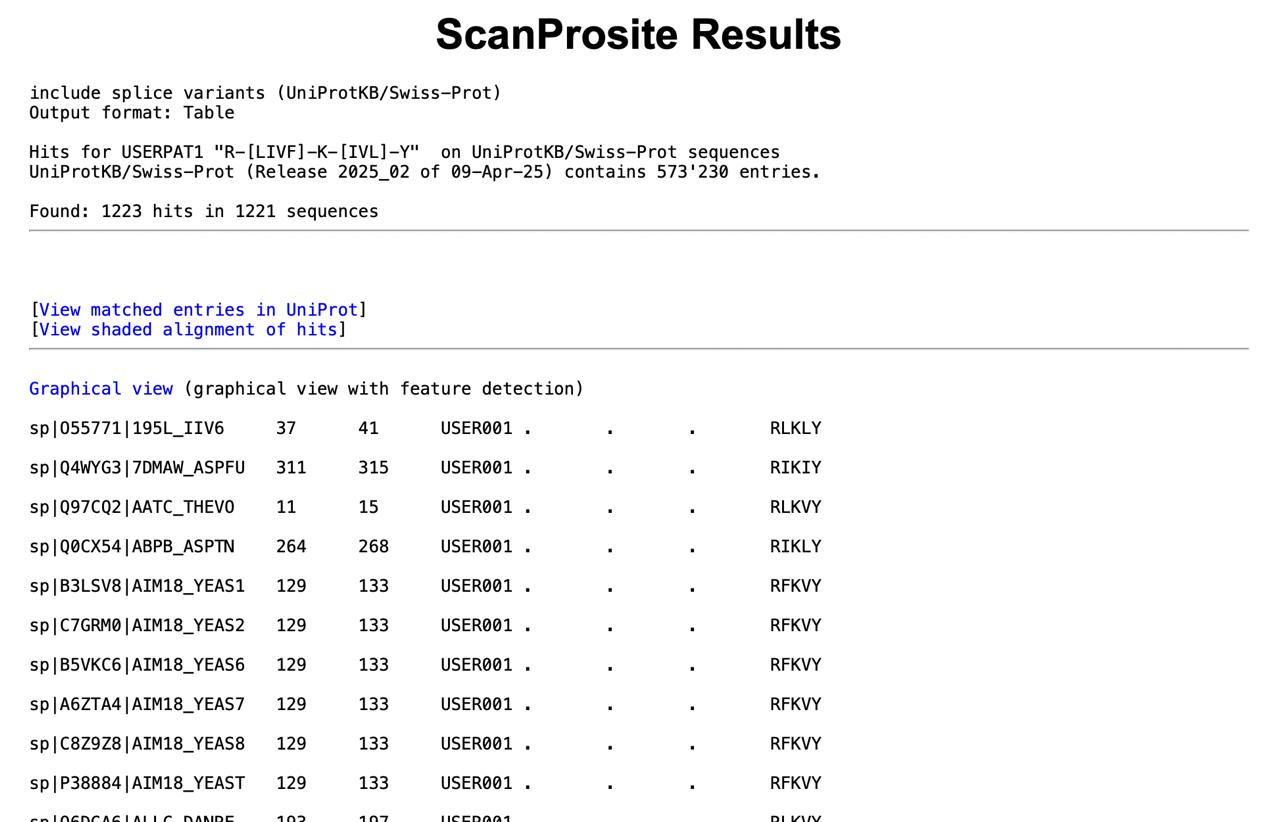
Переведем паттерн в формат Prosite: R-[LIVF]-K-[IVL]-Y. По этому паттерну был проведен поиск на сайте ScanProsite. В результате было найдено 1223 находки в 1221 последовательности. См Рисунок 3.
2. Мотив, специфичный для одной клады филогенетического дерева
Построим в Jalview филогенетическое дерево, методом. Выберем ветвь, отрезающую одну кладу (на рисунке 4, обозначена синим цветом, состоит из 7 видов: E3QZW8_COLGM/35-395, ASQH1_EMENI/54-409, XPTA_EMENI/33-405, B6QJI2_TALMQ/25-397, R0ILN8_EXST2/19-399, M2SSQ0_COCH5/19-398, M7TBS5_EUTLA/28-377)
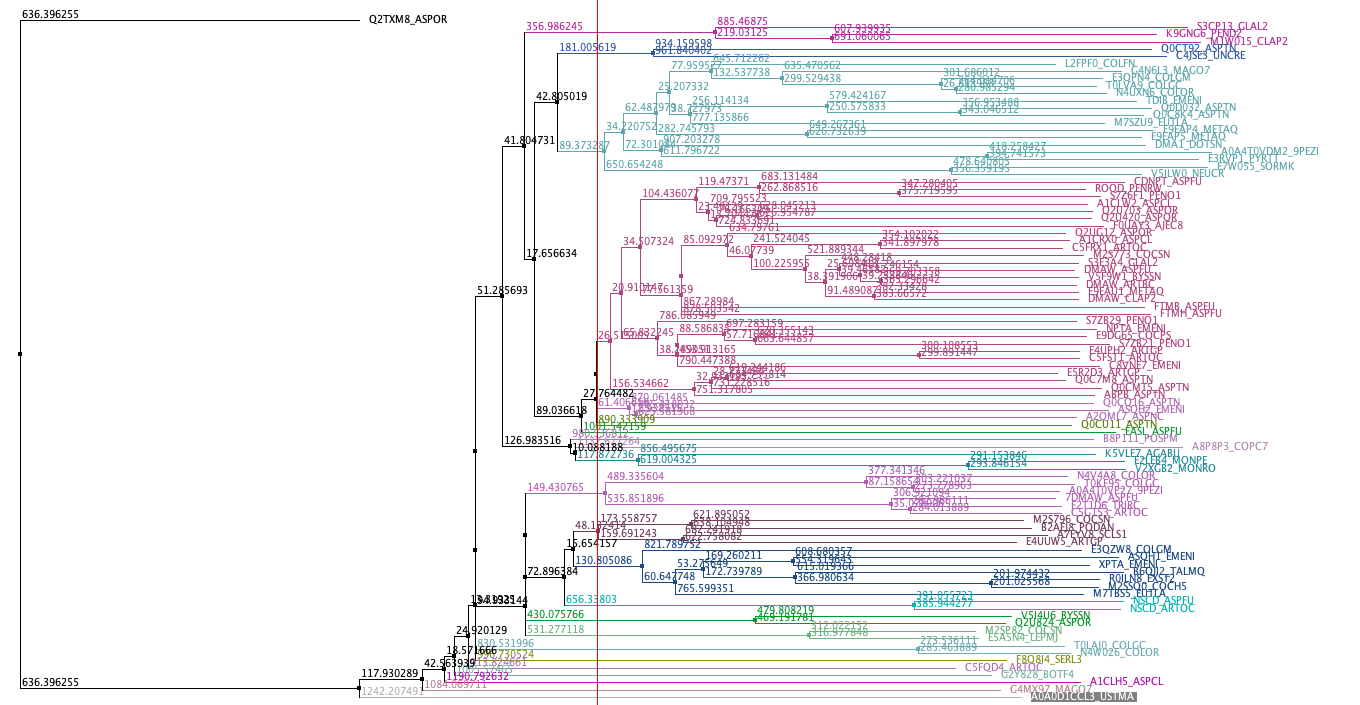
Отделим выравнивание этой клады в отдельное окно. Найдем консервативный мотив в этой кладе, его паттерн: Y..PR..G
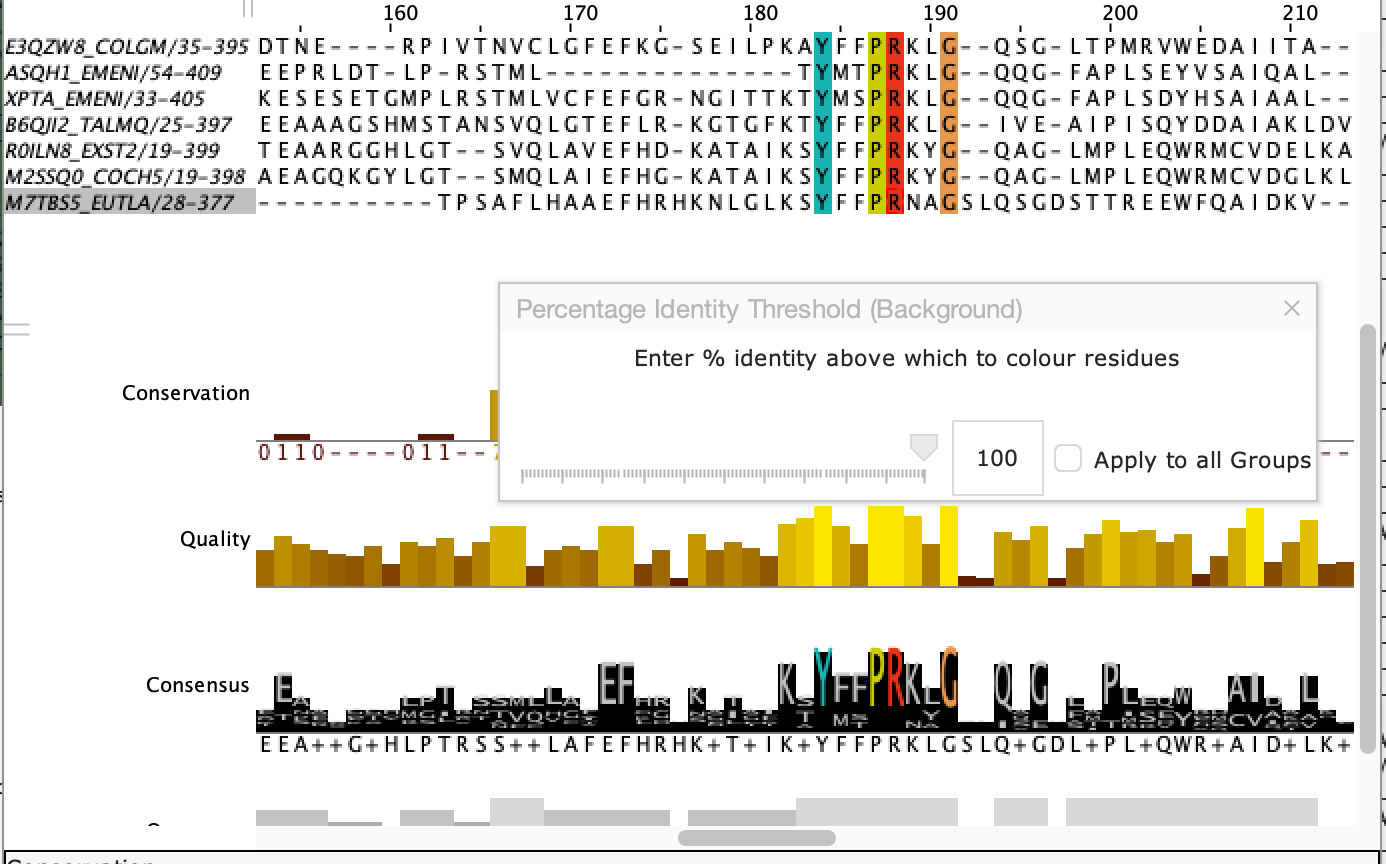
Выполним поиск этого мотива во всем выравнивании. Этот мотив встречается во всех последовательностях клады и не встречается больше нигде в выравнивании, значит он специфичен для данной клады.

3. PSI-BLAST
Был проведён анализ белка с идентификатором Q7VDL2, известного как MinC (белок цианобактерии Prochlorococcus marinus (strain SARG / CCMP1375 / SS120)) - ключевой компонент системы регуляции клеточного деления у бактерий. Данный белок выполняет важную регуляторную функцию: ингибирует образование полярных Z-колец (септальных перегородок) путём дестабилизации нитей FtsZ и предотвращения их полимеризации.
PSI-BLAST анализ проводился с порогом значимости E-value=0.005 по базе данных Swiss-Prot. Результаты трёх последовательных итераций представлены ниже на рисунке 7: На первой итерации было обнаружено 146 значимых гомологов, при этом граничное значение E-value составляло 0.005 (для белка Q9AG20.1). Уже на второй итерации количество значимых совпадений увеличилось до 188, а значение E-value для худшей значимой находки (B6JKX0.1) улучшилось до 7,00E-08. Третья итерация показала стабилизацию результатов - количество находок осталось на уровне 188, при этом минимальное E-value достигло 2,00E-12 (белок Q9ZM51.1), а лучшая незначимая находка (A7H8E6.1) имела E-value=0.014.
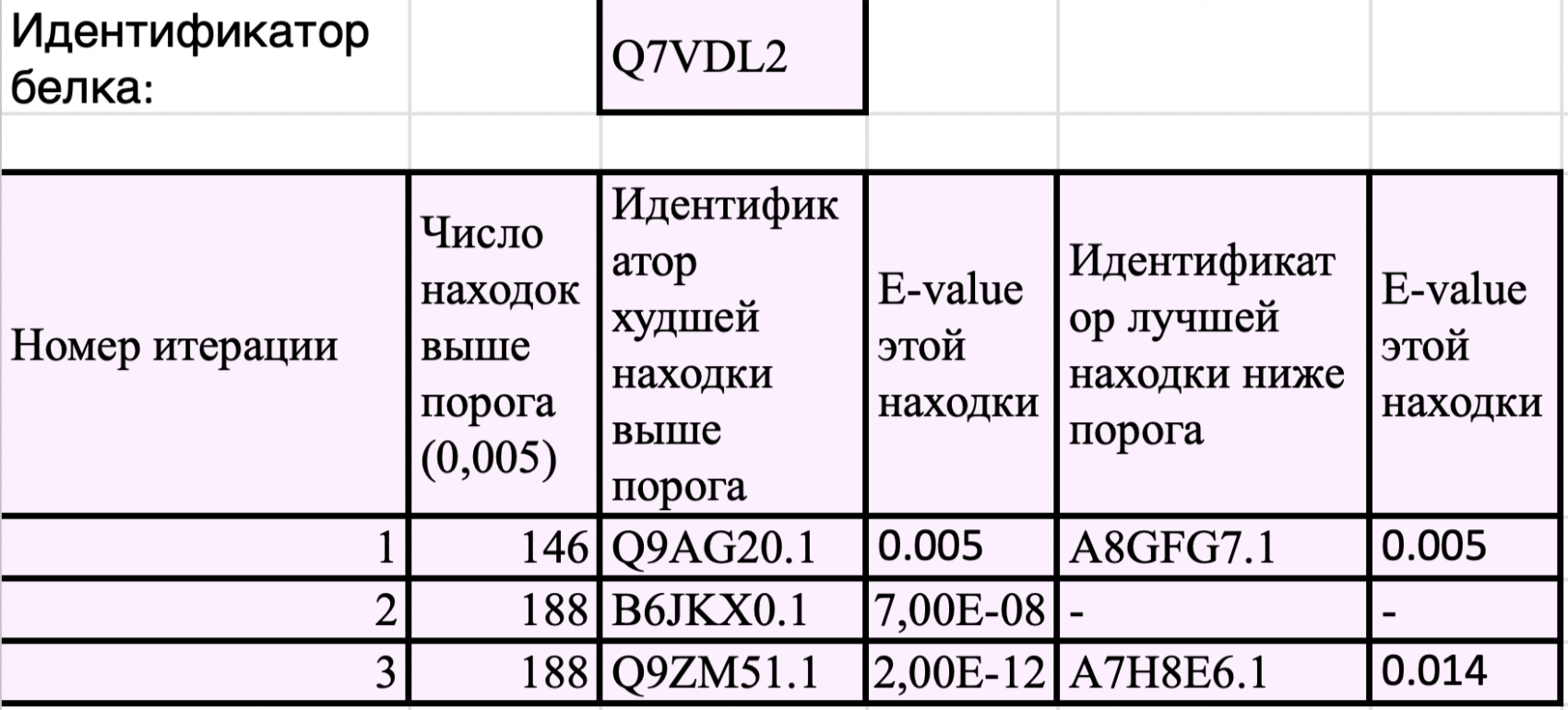
Полученные результаты демонстрируют отличное качество собранного семейства гомологов: Алгоритм PSI-BLAST сошёлся за три итерации, Наблюдается чёткая "ступенька" в значениях E-value (разница в 11 порядков).
4. Поиск в выборке поcледовательностей с доменом из SwissProt de novo мотивов с помощью MEME
Для домена PF11991 был проведен поиск паттернов. Сначала я скачала 236 последовательностей, которые пренадлежат бактериям рода Streptomyces. Потом отобрала 60 (remove redundancy, порог 92% identity). Запустила поиск мотивов de novo по этим 60 последовательностям:
meme 60seq.fa -o results -mod anr -minw 4 -maxw 10 -nmotifs 5 Выдачу программы meme можно посмотреть по ссылке: выдача meme

Далее по 236 последовательностям был проведен поиск мотивов программой fimo: выдача fimo
В Jalview я визуализировала этот мотив в 236 последовательностях: рисунок 9

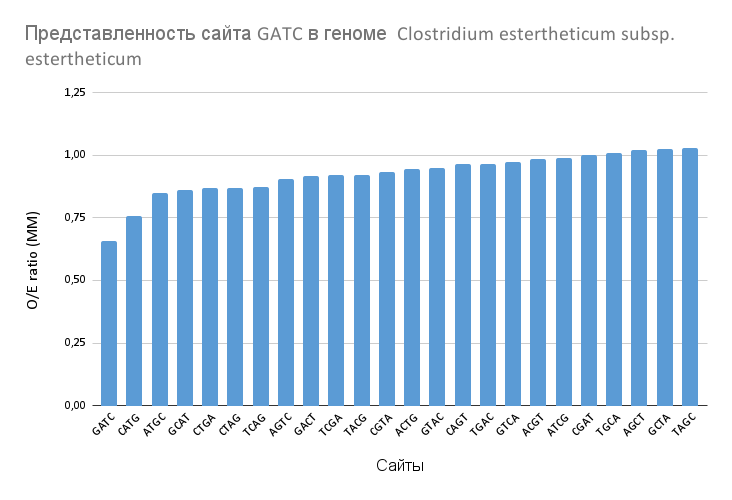
5. Оценка представленности сайта GATC в геноме бактерии Clostridium estertheticum subsp. estertheticum
Был проведен анализ представленности сайта GATC в геноме Clostridium estertheticum subsp. estertheticum с использованием программы cbcalc. Исходными данными послужили геномная последовательность из файла GCF_001877035.1_ASM187703v1_genomic.fna и список исследуемых сайтов рестрикции, сохраненный в sites.txt. Команда запуска анализа включала параметр -M для расчета относительной представленности сайтов и перенаправляла результаты в файл result.tsv.
cbcalc -s sites.txt -M -o result.tsv ../pr9/GCF_001877035.1_ASM187703v1_genomic.fnaВ полученных данных особый интерес представляет значение 0,657 для сайта GATC, которое указывает на его недопредставленность в геноме по сравнению с ожидаемой частотой встречаемости. Такое отклонение может объясняться несколькими биологическими причинами: во-первых, сайт GATC может быть мишенью системы рестрикции-модификации бактерии, что приводит к его активному подавлению; во-вторых, эволюционное давление могло снизить частоту этой последовательности из-за ее функциональной значимости, например, участия в регуляции генов или упаковке ДНК.