Практикум 7. Трансмембранные белки
1. Сравнение предсказаний трансмембранных участков в бета-листовом белке
Я выбрала белок активатор плазминогена PLA (коагулаза/фибринолизин) (англ. Plasminogen activator PLA (coagulase/fibrinolysin)) из бактерии Yersinia pestis (возбудитель чумы). Его PDB-идентификатор — 2X55, а UniProt-идентификатор — COLY_YERPE. Этот белок локализуется в наружной мембране бактерии и играет ключевую роль в патогенезе: он активирует плазминоген хозяина, превращая его в плазмин, что приводит к разрушению фибриновых сгустков (фибринолизу) и способствует распространению бактерий в тканях. При 37°C преобладает фибринолитическая активность, а при 28°C — коагулазная. Белок также может обладать автокаталитической активностью. Его действие облегчает инвазию Yersinia pestis, ускоряя прогрессирование инфекции.
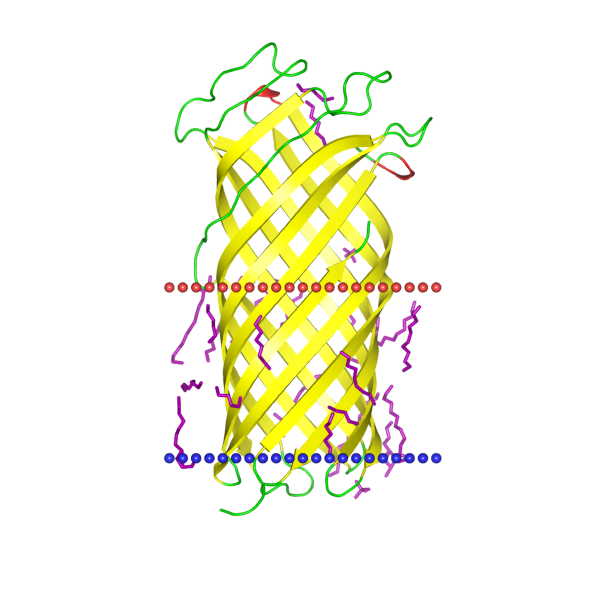
Белок состоит из 1 субъединицы. Координаты трансмембранных участков белка: 1(13-22),2(51-60),3(65-73),4(111-121), 5(126-136),6(174-183),7(188-197),8(224-235),9(239-248),10(282-292).
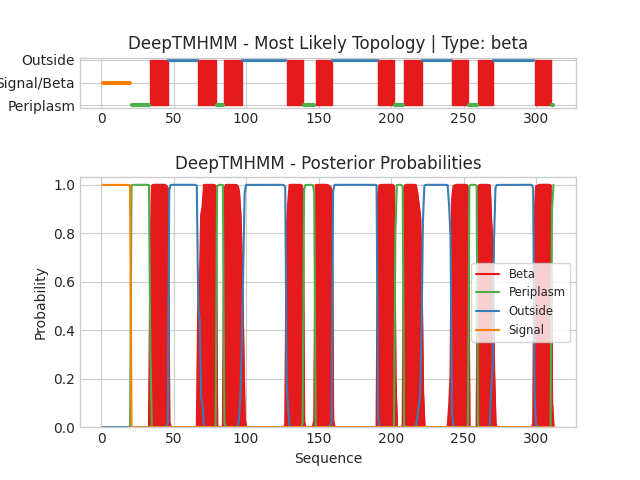
Для предсказания трансмембранных участков белка активатора плазминогена PLA (коагулаза/фибринолизин) из Yersinia pestis была использована программа DeepTMHMM. Аминокислотная последовательность белка была взята из базы данных UniProt (идентификатор COLY_YERPE).
Результаты предсказания:
Тип белка: β-баррельный (подтверждает наличие трансмембранных β-листов).
Трансмембранные сегменты:
1-й сегмент: 34-46
2-й сегмент: 67-79
3-й сегмент: 85-97
4-й сегмент: 128-139
5-й сегмент: 148-159
6-й сегмент: 191-202
7-й сегмент: 209-221
8-й сегмент: 242-253
9-й сегмент: 260-270
10-й сегмент: 299-310
На рисунке 2 показаны:
Most Likely Topology – схема расположения трансмембранных β-сегментов (в виде столбцов) и внеклеточных/периплазматических петель.
Posterior Probabilities – график вероятностей, подтверждающий достоверность предсказанных участков (пики соответствуют трансмембранным областям).
Текстовый отчёт DeepTMHMM можно найти здесь.
Вывод:
Программа DeepTMHMM подтвердила β-баррельную структуру белка COLY_YERPE, что согласуется с данными OPM. Было предсказано 10 трансмембранных сегментов, что соответствует данным OPM. Но наблюдается систематическое смещение координат сегментов между предсказаниями (в среднем на ~20 аминокислот). Это объясняется тем, что в OPM использовалась урезанная последовательность (293 аа) из PDB-структуры, а в DeepTMHMM анализировалась полная последовательность из UniProt (312 аа), включающая N-концевую сигнальную последовательность (~20 аа).
2. Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
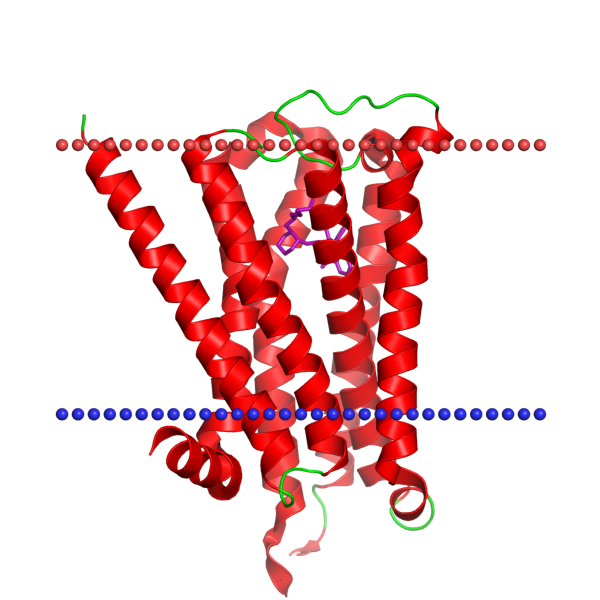
Далее был исследован мускариновый ацетилхолиновый рецептор М2 (англ. Muscarinic acetylcholine receptor M2), имеющий идентификатор PDB 5ZKB и UniProt ACM2_HUMAN (P08172). Этот белок относится к классу G-белок-связанных рецепторов (GPCR) и экспрессируется у человека (Homo sapiens). Основная функция рецептора заключается в передаче сигналов через взаимодействие с G-белками: он ингибирует аденилатциклазу, активирует фосфолипазу C с последующим образованием инозитолтрифосфата (IP3) и высвобождением ионов кальция, а также модулирует работу калиевых каналов. Рецептор играет ключевую роль в регуляции сердечного ритма, сокращении гладких мышц и нейромедиаторной передаче. В структуре 5ZKB белок представлен одной цепью (A), формирующей 7 трансмембранных α-спиралей, характерных для GPCR.
Координаты трансмембранных участков белка из OPM: 1( 19- 46), 2( 61- 86), 3( 90- 119), 4( 141- 165), 5( 183- 207), 6( 389- 412), 7( 416- 440).
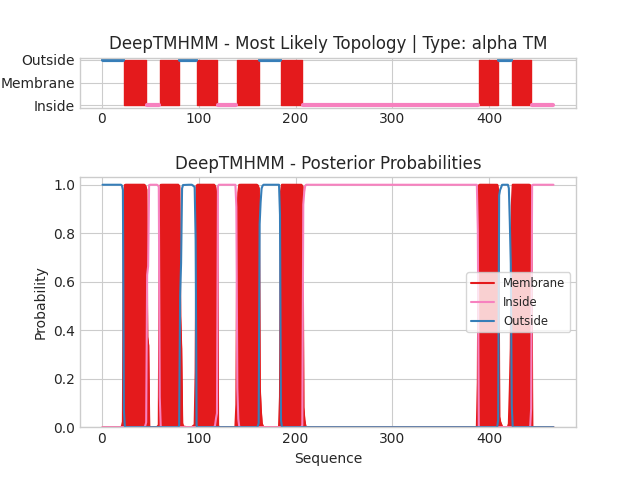
Далее были сопоставлены результаты предсказаний DeepTMHMM и экспериментальные данные OPM. Оба метода идентифицировали 7 характерных для GPCR трансмембранных α-спиралей. DeepTMHMM определил следующие границы спиралей: TM1 (23-46), TM2 (60-80), TM3 (98-119), TM4 (140-162), TM5 (185-207), TM6 (389-409) и TM7 (423-443) (текстовая выдача ), в то время как OPM показал несколько иные координаты: TM1 (19-46), TM2 (61-86), TM3 (90-119), TM4 (141-165), TM5 (183-207), TM6 (389-412) и TM7 (416-440). Наибольшие расхождения наблюдаются в N-концевых участках TM2 (разница 1-6 остатков) и TM3 (разница 8 остатков), а также в C-концевой области TM7 (разница 7-17 остатков). Различия в точных границах спиралей (в среднем 5-10 аминокислотных остатков) находятся в пределах погрешности. Можно отметить консервативность предсказаний в отношении шестой спирали (TM6), где расхождение составляет всего 3 остатка (389-409 vs 389-412), что может говорить об особой структурной и функциональной важности этого домена для работы рецептора.