Практикум 8. Сигналы в геноме.
1. Выбор и описание сигнала
В данном практикуме был выбран сигнал ядерной локализации - Nuclear localization sequence (NLS).
Краткая сводка о сигнале представлена в таблице ниже [1-2].
| Название сигнала | Nuclear localization sequence |
| Носитель сигнала | Белки, предназначенные для транспорта в ядро |
| Кому адресован | Импортин-α/β (ядерные транспортные рецепторы) |
| Предназначение | Распознавание сигнала комплексом импортинов с последующим транспортом белка через ядерные поры |
| Сила сигнала | Зависит от типа NLS (моно- или бипартитный), заряда и контекста последовательности. Классические NLS имеют высокую аффинность к импортину-α. |
Кроме того, интересно, что сигналы данного типа можно разделить на две группы: классический монопартитный NLS и бипартитный NLS.
| Монопартитный NLS | Бипартитный NLS | |
|---|---|---|
| Консенсус | Практически непрерывно расположенные основные аминокислоты (K или R) | Два кластера основных аминокислот, разделенных спейсером (10-12 аа) |
| Пример | PKKKRK (SV40 large T-antigen) | KRPAATKKAGQAKKKK (nucleoplasmin) |
2. Анализ с помощью NLStradamus
Нами было найдено несколько интерфейсов для поиска NLS, в том числе NLS Mapper и NLStradamus. Однако, согласно литературным источникам [3], использование NLStradamus дает статистически значимое улучшение качества поиска. NLStradamus использует скрытую марковскую модель (HMM) для предсказания ядерных сигналов локализации в белковых последовательностях. В случае NLStradamus как и в других подобных моделях используются 1. NLS-состояние: Соответствует участкам последовательности, которые являются NLS. Аминокислоты генерируются с частотами, характерными для известных NLS 2. Фоновое состояние: Соответствует участкам последовательности, которые не являются NLS. Аминокислоты генерируются с частотами, характерными для генома в целом.
Для проверки работы поиска с помощью NLStradamus был проведен поиск по последовтельности, наличие NLS заведомо известно - Large T antigen (Simian virus 40 (SV40)). Последовательность была взята из соотвествующей записи UniProt. Поиск проводился по порогу апостериорной вероятности равному 0.5. Веб интрефейс позволяет получить предсказнный NLS участок и графическое представление вероятности по позициям и относительное положенеи порога.
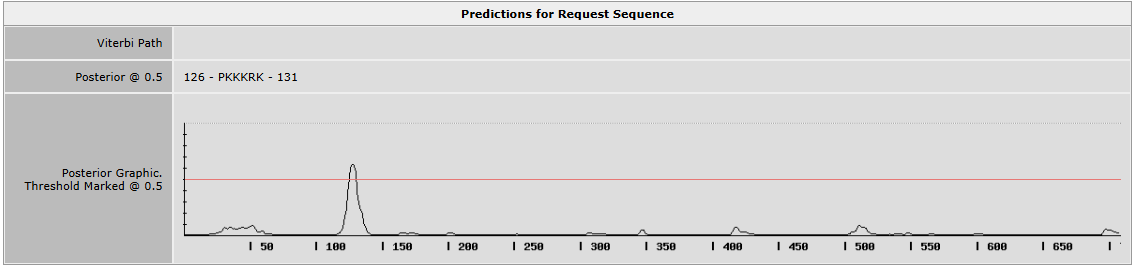
Рис.1 Графическая выдача программы NLStradamus
Литературные источники
1. Shaheen, Sharif. (2015). Equations to predict nuclear localization signal (NLS) peptide based on the hydropathy index ratio. Literati Journal of Pharm. Drug Del. Tech.. 1. 27-36. 10.15272/ljpddt.v1i2.13. 2. Chang, Chiung-Wen et al. (2014) Structural Basis of Interaction of Bipartite Nuclear Localization Signal from Agrobacterium VirD2 with Rice Importin-α. Molecular Plant, Volume 7, Issue 6, 1061 - 1064 3. Nguyen Ba AN, Pogoutse A, Provart N, Moses AM. NLStradamus: a simple Hidden Markov Model for nuclear localization signal prediction. BMC Bioinformatics. 2009 Jun 29;10(1):202.