Практикум 9. Поиск сигналов инициации транскрипции и трансляции
1. Цель работы
Поиск в геноме бактерии Clostridium estertheticum subsp. estertheticum сигналов, связанных с инициацией транскрипции (последовательности -10 и -35 для сигма-субъединицы РНК-полимеразы) и трансляции (последовательность Shine-Dalgarno).
2. Методы и шаги анализа
2.1 Подготовка данных
Загрузка аннотации генома в формате GFF:
wget https://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/001/877/035/GCF_001877035.1_ASM187703v1/GCF_001877035.1_ASM187703v1_genomic.gff.gz
gunzip GCF_001877035.1_ASM187703v1_genomic.gff.gz2.2 Выделение промоторных областей
awk '{if ($3=="gene") print $1 "\t" $4-100 "\t" $4 "\t" $9}' GCF_001877035.1_ASM187703v1_genomic.gff > promoters.bed
bedtools getfasta -fi GCF_001877035.1_ASM187703v1_genomic.fna -bed promoters.bed -fo promoters.fasta2.3 Поиск мотивов de novo с помощью MEME
meme promoters.fasta -o meme_results -dna -mod anr -nmotifs 5 -minw 6 -maxw 122.4 Контроль: анализ межгенных регионов
awk '$3 == "gene" {print $1 "\t" $4-100 "\t" $5 "\t" $7}' GCF_001877035.1_ASM187703v1_genomic.gff > genes.bed
bedtools complement -i genes_sorted.bed -g GCF_001877035.1_ASM187703v1_genomic.fna.fai > intergenic.bed
bedtools getfasta -fi GCF_001877035.1_ASM187703v1_genomic.fna -bed intergenic.bed -fo intergenic.fasta2.5 Валидация мотивов с помощью FIMO
fimo --o fimo_promoters meme_results/meme.xml promoters.fasta
fimo --o fimo_intergenic meme_results/meme.xml intergenic.fasta3. Результаты и интерпретация
1. Найденные мотивы:
Выдачу программы meme можно посмотреть по ссылке: meme.html
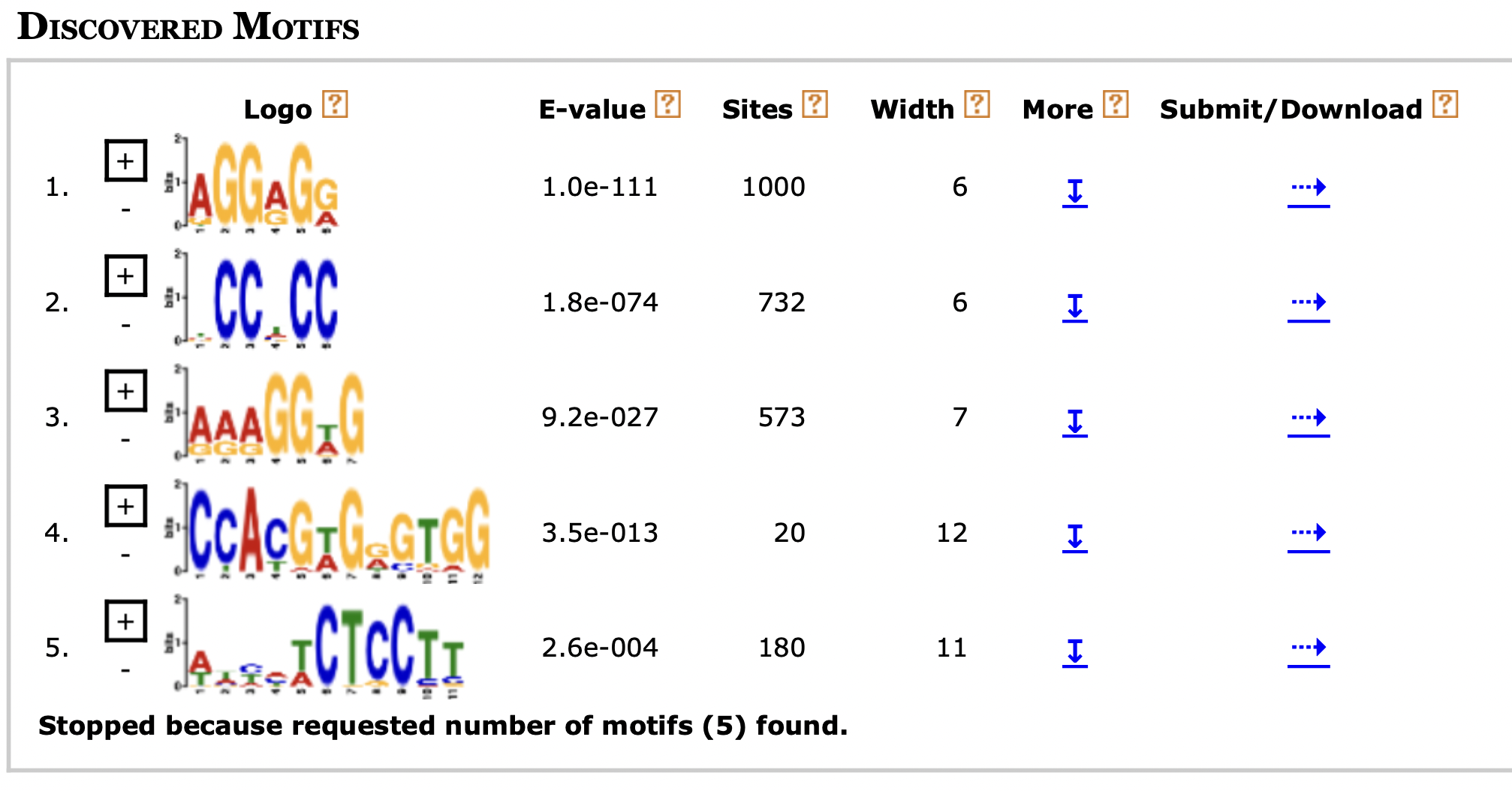
Самое маленькое значение E-value у первого мотива AGGAGG, это последовательность Шайна-Дальгарно, которая нужна для инициации трансляции. Третий мотив тоже похож на последовательность Шайна-Дальгарно, может она характерна для каких-то особенных генов. Второй мотив является значимой находкой, при этом он почти комплиментарен последовательности ШД, не могу пока сообразить как так получается. Четвертая и пятая находки имеют не такое низкое E-value, либо это случайные находки, либо какие-то редкие специфические сигналы.
2. Контроль качества:
Выдачу программы fimo можно посмотреть по ссылкам: выдача fimo для промоторных областей, выдача fimo для межгенных областей (с вырезанными промоторными областями).
Мотивы должны быть обогащены в промоторах (больше находок, меньшие p-value) по сравнению с межгенными регионами. Но у меня получились какие-то странные результаты, почему-то в обоих файлах описываются находки 3,4,5 мотивов, причем значения p-value почти не различаются между промоторными областями и межгенными, можно подумать, что 3,4,5 мотивы случайны. Но я не понимаю, а где находки 1 и 2 мотивов, их должно быть много.
Я решила немного изменить поиск, указала в параметрах meme -nmotifs 3, чтоб нашлось 3 мотива, и опять запустила fimo для промоторных областей. Результат: meme для 3 мотивов; fimo для 3 мотивов в промоторных областях. Программа fimo нашла только 3 мотив, 1 и 2 мотив не были найдены вообще. Возможно проблемма именно в них. Запустим поиск 2 мотивов (-nmotifs 2), а затем опять fimo. Результат: meme для 2 мотивов; fimo для 2 мотивов в промоторных областях. Программа fimo ничего не нашла. Непонятно, почему fimo не может найти самые распространенные патерны.