Резюме
Предметом исследования является активно использующаяся в промышленности грамотрицательная бактерия Acidithiobacillus ferrooxidans. В работе изучается геном бактерии. В частности, исследован кодоновый состав белок-кодирующих последовательностей, определены точки начала и конца репликации. Приведены статистические данные о геноме
Введение
Acidithiobacillus ferrooxidans - это грамотрицательная гамма-протеобактерия. Она является экстремально ацидофильным хемолитоавтотрофом, оптимальный pH среды для которой 2 [1]. Ac. ferrooxidans может окислять разные неорганические и органические субстраты как в аэробных так и в анаэробных условиях для получения энергии и электронов [2].
Эта бактерия интересна, в первую очередь, своими потенциальными и уже реализованными способами применения. Благодаря способности окислять восстановленные соединения серы до сульфатов, а ионы Fe2+ до Fe3+, Ac. ferrooxidans используется для выщелачивания сульфидных руд различных металлов; может использоваться для очистки от серы газов и твердых веществ; также Ac. ferrooxidans может применяться для выделения металлов из вторсырья [3]. Помимо этого бактерия образует магнитосомы, которые потенциально можно применять для доставки лекарств в определенные места [3,4].
Также Ac. ferrooxidans интересен как объект для изучения процессов биологического окисления железа и серы, метаболизма экстремофилов и прикрепления бактерий к субстрату [5,6]. Этому способствуют, в том числе, имеющиеся инструменты генетического редактирования этой бактерии [5].
В данном обзоре приведены стандартные статистические данные о геноме, исследован кодоновый состав белок-кодирующих генов, найдены ориджин и термиус репликации.
Материалы и методы
Для написания обзора были использованы- таблица генов GCF_013462805.1_ASM1346280v1_feature_table, взят с NCBI.
- документ, содержащий геном бактерии в fasta-формате GCF_013462805.1_ASM1346280v1_genomic.fna, взят с NCBI.
- Программа в google colaboratory на языке программирования python, считающая соотношения нуклеотидов в геноме и оценивающая их по критерию хи-квадрат
- Программы в bash: findtRNA определяет количество транспортных РНК, закодированных в геноме, findrRNA выводит количество рибосомальных РНК, закодированных в геноме, findribosomal выводит количество рибосомных белков, findhypothetical - количество гипотетических белков.
- Программа в google colaboratory на языке программирования python, считающая количество разных кодонов, кодирующих аминокислоты. Сначала вырезаются белок-кодирующие последовательности с учётом цепи на которой они расположены(если на - цепи, после вырезания строится комплементарная перевёрнутая последовательность), из них исключаются те, в которых есть сдвиг рамки считывания, затем подсчитываются все кодоны кроме старт и стоп-кодонов. После этого программой подсчитывается доля каждого кодона среди синонимичных. Далее программа строит таблицу, в которой отражена доля разных кодонов среди синонимичных для каждой аминокислоты (за исключением тех, которым соответствует один кодон)
- Программа в google colaboratory на языке программирования python, считающая GC-skew с шагом 500 нуклеотидов и размером окна 50000 по формуле GCskew =(G-C)/(G+C) где G, C - количество соответствующих нуклеотидов в окне, cumulative GC-skew - это сумма всех значений GC-skew до данной позиции. Подсчет происходит в последовательности нуклеотидов хромосомы, вырезаемой из последовательности в fasta-формате.
Результаты
Стандартные данные о геноме : Геном бактерии состоит из хромосомной днк длиной 3209933 пн и одной плазмиды p1 длиной 47104 пн. При помощи программы (3) была определена доля GC пар и отдельных нуклеотидов в хромосоме и в плазмиде. Результаты согласуются со вторым правилом Чаргаффа для G и C в хромосоме и в плазмиде и для A и T в хромосоме, так как p-value критерия хи-квадрат меньше 0,01. Однако для для соотношения A и T в плазмиде правило Чаргаффа не выполняется, при p-value 0.018 (см Таблицу 2).

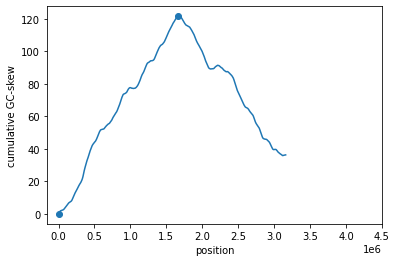
Ориджин и термиус:
При помощи программы (6) был посчитан cumulative GC-skew для хромосомы на основе этих данных построен график (смотри рис.1). Опираясь на эти данные можно предположить что в точке минимума 0 находится ориджин - точка начала репликации, а в точке максимума 1668000 термиус - точка окончания репликации, где встречаются разнонаправленные репликативные вилки.
Статистические данные о протеоме:
Используя инструменты электронных таблиц и таблицу генетических особенностей, можно выяснить, что на прямой цепочке ДНК закодировано 1648 генов, кодирующих белки, на комплементарной - 1520 (1).

С помощью программ, написанных в BASH было определено количество генов рибосомальных, транспортных и гипотетических белков, закодированных в геноме бактерии и их доля от общего количества белок-кодирующих генов (1,4) (смотри Таблицу 3).
При помощи инструментов гугл-таблиц была построена гистограмма длин белков, которая показывает типичную для прокариот картину преобладания небольших белков (смотри Рис.2).
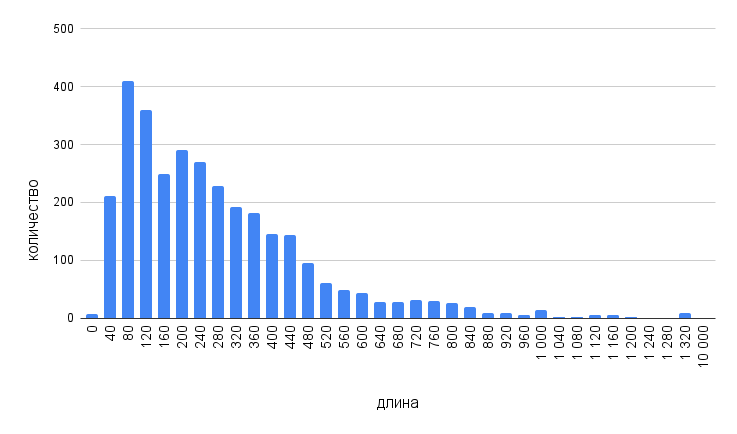
Статистические данные о генах, кодирующих некоторые нематричные РНК:
В геноме бактерии содержатся гены 59 нематричных РНК, в том числе 47 генов транспортных РНК, и 6 - рибосомальных. Доля РНК-кодирующих генов 1,86%. Данные были получены с помощью программ с использованием информации из таблицы генетических особенностей (1,4).
Исследование содержания различных кодонов в белок-кодирующих генах:
С помощью программы (5) было подсчитано соотношение кодонов, кодирующих разные аминокислоты в белок-кодирующих последовательностях (см таблицу S1). В статистику не попали 5 генов со сдвигом рамки считывания, так как в них невозможно подсчитать кодоны, не зная координаты сдвига.
Обсуждение
Обнаруженное отклонение отношения количества аденинов и тиминов в плазмиде от того, которое можно ожидать согласно правилу Чаргаффа может быть неслучайно, и, возможно, является следствием еще неизвестной закономерности.
Выявление локализации ориджина важно для понимания интенсивности экспрессии генов, так, гены, находящиеся вблизи ориджина часто экспрессируются гораздо интенсивнее, так как во время репликации гены, находящиеся вблизи ориджина дольше чем остальные находятся в удвоенном количестве (находятся на двух новосинтезированных цепочках). Следовательно, встраивание новых генов в районы рядом с ориджином может приводить к увеличенной наработке продуктов их экспрессии.[7]
Определение кодонового состава белок-кодирующих последовательностей может быть полезно для оптимизации генетического кода генетических конструкций, встраиваемых в геном бактерии, так как для более распространенных кодонов, как правило, более распространены тРНК с подходящим антикодоном. Если учитывать это при генетическом редактировании, можно повысить эффективность и скорость экспрессии встраиваемых белок-кодирующих генов благодаря соответствию кодонов кодирующих аминокислоты антикодонам самых распространённых тРНК в клетке.
Сопроводительные материалы
- Путь к таблице с особенностями генома на kodomo: /P/y22/term1/credits/GCF_013462805.1_ASM1346280v1_feature_table.txt Ссылка на него на сайте NCBI
- Путь к fasta-файлу с геномом бактерии на kodomo: /P/y22/term1/credits/GCF_013462805.1_ASM1346280v1_genomic.fna Ссылка на него на сайте NCBI
- Ссылка на программу в google colaboratory, считающую нуклеотидный состав генома
- Путь к программам на kodomo /home/students/y22/timurk/term1/scripts/findrRNA; /home/students/y22/timurk/term1/scripts/findtRNA; /home/students/y22/timurk/term1/scripts/findtransport; /home/students/y22/timurk/term1/scripts/findribosomal; /home/students/y22/timurk/term1/scripts/findhypothetical
- Ссылка на программу, считающую количество кодонов для аминокислот в генах и их долю от синонимичных
- Ссылка на программу, строящую график cumulative GC-skew
- S1 - таблица, содержащая информацию о доле кодонов, кодирующих каждую из аминокислот относительно других синонимичных кодонов.
Ссылки
- Jorge Valdés, Inti Pedroso, Raquel Quatrini, Robert J Dodson, Herve Tettelin, Robert Blake , Jonathan A Eisen and David S Holmes. Acidithiobacillus ferrooxidans metabolism: from genome sequence to industrial applications. BMC Genomics (2008). DOI:https://doi.org/10.1186/1471-2164-9-597
- Raquel Quatrini, Corinne Appia-Ayme , Yann Denis, Jeanine Ratouchniak, Felipe Veloso, Jorge Valdes, Claudia Lefimil, Simon Silver, Frank Roberto , Omar Orellana, François Denizot, Eugenia Jedlicki, David Holmes, Violaine Bonnefoy. Insights into the iron and sulfur energetic metabolism of Acidithiobacillus ferrooxidans by microarray transcriptome profiling. Hydrometallurgy (2006) DOI: https://doi.org/10.1016/j.hydromet.2006.03.030
- .Shuang Zhang, Lei Yan, Weijia Xing, Peng Chen, Yu Zhang, Weidong Wang. Acidithiobacillus ferrooxidans and its potential application. Springer. (2018) DOI:https://doi.org/10.1007/s00792-018-1024-9
- Jianbo Sun, Ying Li, Xing-Jie Liang, Paul C Wang. Bacterial Magnetosome: A Novel Biogenetic Magnetic Targeted Drug Carrier with Potential Multifunctions. Journal of Nanomaterials (2011). DOI:https://doi.org/10.1155/2011/46903
- Heejung Jung, Yuta Inaba, Scott Banta. Genetic engineering of the acidophilic chemolithoautotroph Acidithiobacillus ferrooxidans. Trends in biotechnology (2022). DOI: https://doi.org/10.1016/j.tibtech.2021.10.004
- T. Gehrke, R. Hallmann, K. Kinzler and W. Sand. The EPS of Acidithiobacillus ferrooxidans – a model for structure-function relationships of attached bacteria and and their physiology Research in Microbiology (pages 49-56, 2006)DOI: 10.2166/wst.2001.0365
- The Proximity of Ribosomal Protein Genes to oriC Enhances Vibrio cholerae Fitness in the Absence of Multifork Replication. Alfonso Soler-Bistué, Michaël Timmermans, Didier Mazel. ASM Journals (2017) DOI: https://doi.org/10.1128/mBio.00097-17
- Arakawa K, Tomita M. The GC Skew Index: A Measure of Genomic Compositional Asymmetry and the Degree of Replicational Selection. Evolutionary Bioinformatics. 2007;3. DOI:10.1177/117693430700300006