Результаты
Стандартные данные о геноме
Геном Pyrococcus abyssi состоит из одной кольцевой хромосомы и плазмиды pGT5
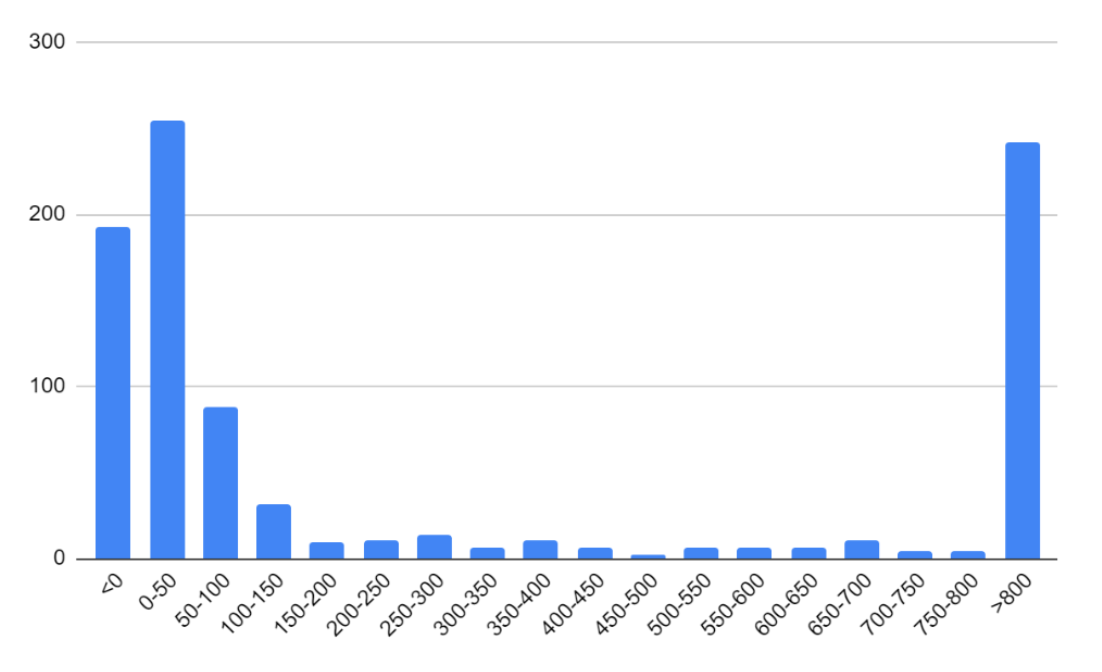
Из размеров ожидаемо, что плазмида не несет большого количества генов. Она содержит два гена кодирующих белки, при этом роль один из них, исходя из имеющихся данных, спрогнозирован, но не был выделен. Зато второй белок - инициатор репликации, ген которого в остальном геноме не встречается. По-видимому именно из-за него плазмида не потерялась в ходе отбора, а возможно, что вынесение этого белка в плазмиду функционально важно, так как посредством самостоятельной репликации плазмиды или даже ее размножения в клетке возможна быстрая наработка инициаторного белка. Посредством электронных таблиц были проанализированы расстояния между соседними последовательности в геноме (Диаграмма 1). Наличие отрицательных расстояний показывает, что не так мало последовательностей между собой перекрываются. Также заметно, что есть некоторое частое встречающееся расстояние около от 0 до 50, а далее последовательности распределяются на разные расстояния довольно случайно.
Диаграмма 1 По горизонтали - различные расстояния между двумя соседними генами. По вертикали - количество таких промежутков. Заметно большое количество отрицательный расстояний и видно, что случайное распределение на промежутке расстояний 150-800 распространяется и дальше.
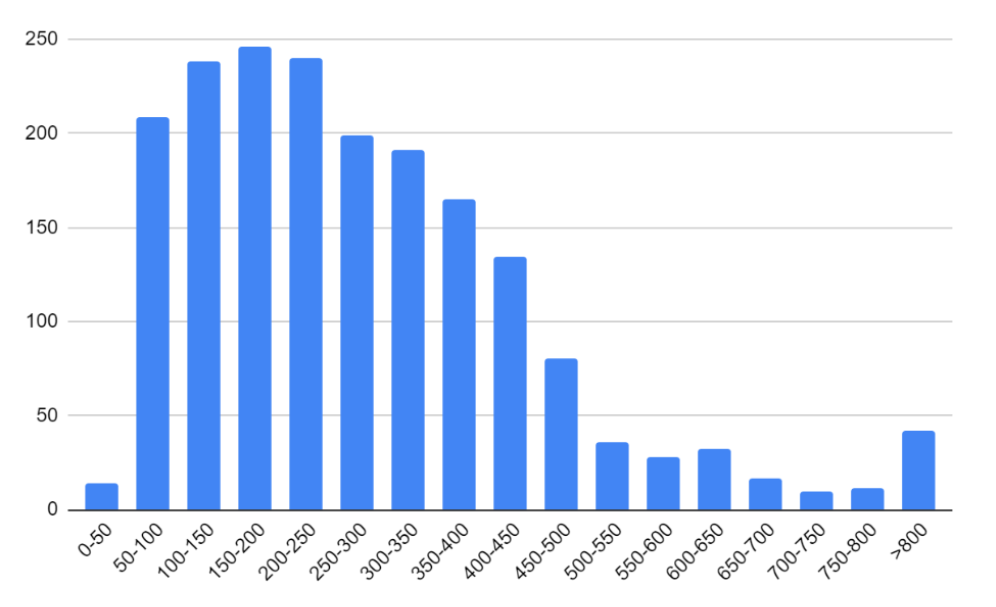
Аналогичным методом было оценено распределение длин белков (Диаграмма 2). На ней видно, что распределены вполне обычно: средняя длина порядка 300, не так много длинных белков, почти нет совсем коротких, так как, вероятно, в большинстве своем они мало функциональны для археи
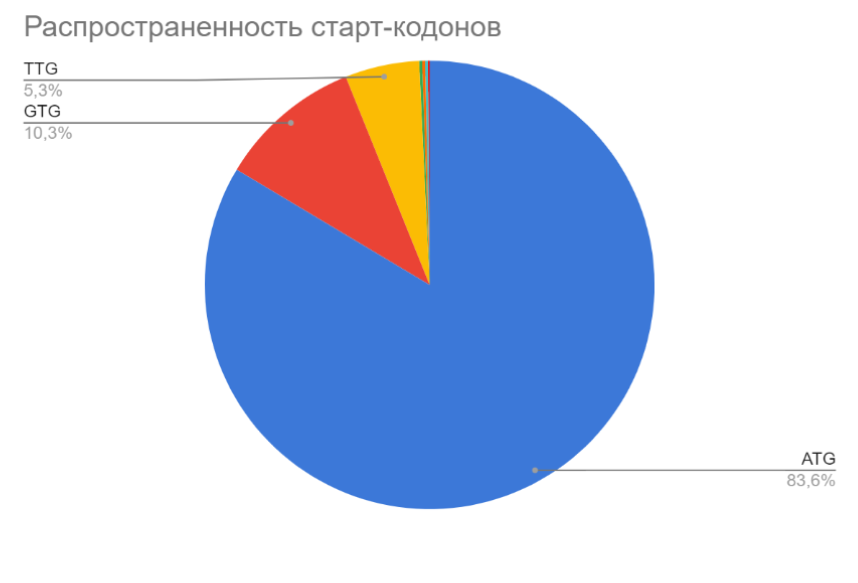
Диаграмма 2 По горизонтали - различные длины продуктов белок кодирующий последовательностей (в аминокислотных остатках. По вертикали - встречаемость таких длин. Заметно распределение, не слишком сильно отличающееся от стандартного. Одна вершина где-то около 150-200 аминокислотных остатков.. У прокариот встречаются различные старт кодоны, помимо привычного ATG. Путем написания программы на python[5] был совершен обсчет всех присутствующих в геноме старт кодонов. Несколько упуская детали, но более наглядно можно представить данные в виде секторной диаграммы (Диаграмма 3)
Диаграмма 3 На диаграмме изображены доли разных старт кодонов от их общего числа. Очевидно, что стандартный ATG в абсолютном большинстве. За ним также вполне характерный для прокариот GTG. Среди других, менее часто встречающихся, только TTG имеет какую-то значимую долю, остальные же встречаются в геноме фактически единично. Несмотря на то, что есть синонимичные кодоны, кодирующие одну и ту же кислоту, в ходе эволюции организма бывает равновесие смещается в сторону конкретных кодонов, и тогда, бывает, возникают ситуации, когда надо знать это отношение. Например такая задача может возникать в биотехнологии, когда нужно максимально эффективно синтезировать белок, исходя из “предпочтений” возможных продуцентов и состава продукта приходиться подгонять синонимичные замены в продукте под продуцента, или продуцента под продукт. Поэтому было решено определить долю разных аминокислот в белковых продуктов + и - цепей ДН суммарно. Для этого была написана программа на python[5], с заимствованием словаря codon_table из лекции по практической информатике ФББ 2023 первого курса. тРНК как и другие гены иногда реплицируются, иногда теряются. У организмов с небольшим геномом, вероятно, такие потери и приобретения значимы, так как обеспечение генома тоже затратный процесс, то возможно, у таких изменений есть функционал. С помощью python[5] были подсчитаны все гены тРНК, однако, не были никак учтены отдельные антикодоны у тРНК. Учесть это, и, возможно, оценить вобблинг-взаимодействие тРНК с разными кодонами возможно могло бы показать что-то интересное, но это уже задел на будущее.Также, пожалуй, для оценки необходимо сравнить результат с другими организмами.
Состав генома
Расстояния на + цепи между кодирующими последовательностями
Длина белков
Старт кодоны
Встречаемость синонимичных кодонов аминокислот в продуктах + и - цепи вместе.
тРНК
Сопроводительные материалы
Список литературы