Семейство доменов
| Таблица | Информация | комментарии (if any) | пояснения |
|---|---|---|---|
| AC pfam | PF00067 | Имеет вид PF…. (пять цифр) | |
| ID pfam | Cytochrome P450 | Гем-содержащие белки, участвуют в окислении различных веществ, но наиболее известны за окисление (=>ликвидацию) мутагенов и токсинов. | В верхней строке стрницы идёт сразу за AC. Short name -это не то. |
| #SEED | 50 | число последовательностей в выравнивании SEED.Найдёте по ссылке Alignments | |
| #All | 509k | Число всех белков с доменом семейства. В меню Proteins => all | |
| #SW | 2k | Число белков с доменом Swissprot. В меню Proteins => reviewed | |
| #architectures | 3969 | Число разных доменных архитектур. В меню architectures | |
| #3D | 1k | Число доменов с известной 3D структурой. Меню Structures | |
| Taxonomy | Крайне широко представлены у эукариот, также крайне много у бактерий. У архей крайне немного записей о такого рода белка. В целом же белки этого семейства очень распространены. | Число всех белков с доменом семейства по супер-царствам. | |
| #eukaryota | 376517 | ||
| #archaea | 1085 | ||
| #bacteria | 127607 |
Выравнивание Seed
| Таблица 11-3 | Информация |
|---|---|
| Выравнивание seed | 50 последовательностей, 558 колонок |
| Максимальный достоверный блок, включающие ВСЕ последовательности (МДБ-all) | 496-505 |
| 100% консервативные колонки в МДБ-all | 397, 400, 496, 499, 503 и 505 |
| "Максимальный достоверный блок, включающий НЕ ВСЕ последовательности. (МДБ-notAll). Если есть другие максимальные блоки с тем же подмножеством последовательностей, то опишите их." | 495-509, 456-465, 397-400 от 1й до 5й |
| 100% консервативные колонки в МДБ-notAll | 1:P 3:S 4:L 9:P 10:L 12:G 13:S 14:L 17:L 28:H 34:L 35:Q 38:Y 41:G 44:Y 49:G 61:Q 63:A 65:E 66:V 67:L 69:K 70:K 71:G 72:K 74:F 76:G 77:R 78:P 82:T 93:K 96:A 97:F 98:A 105:W 109:R 119:F 128:E 129:K 131:I 134:E 138:L 139:C 157:D 166:V 168:N 174:C 175:F 178:S 184:D 185:P 207:G 208:I 221:L 223:D 226:P 230:L 233:F 234:P 242:K 248:R 252:L 292:L 301:N 318:H 320:L 322:T 324:G 325:D 326:I 327:F 328:G 329:A 330:G 332:E 333:T 334:T 337:V 340:W 346:L 348:H 350:P 361:D 366:G 369:R 380:P 384:D 385:R 388:L 391:L 392:E 393:A 394:T 395:I 397:E 399:L 400:R 402:R 403:P 404:V 406:P 410:L 411:I 412:P 413:H 421:D 423:S 424:I 425:G 434:K 441:N 442:L 443:W 446:H 447:H 451:E 452:W 456:P 459:F 461:P 463:R 464:F 465:L 469:G 489:P 492:S 493:Y 494:L 495:P 496:F 497:G 498:A 499:G 501:R 503:C 505:G 506:E 508:L 509:A 512:E 514:F 521:L 522:Q 523:R 535:L 536:P 538:L 540:G 547:V 555:V |
| Участок выравнивания, в котором нет никаких достоверных подблоков, и потому маловероятно, что выравнивание на этом участке отражает ход эволюции. | 257-262 |
Карта локального сходства
| Таблица 11-5 | Информация | комментарии (if any) | пояснения |
|---|---|---|---|
| доменная архитпектура 1 | PF00067 | вберите доменую архитектуру с выбранным доменом | |
| Белок с архитектурой 1 | P00176 | укажите идентификатор последовательности с этой архитектурой | |
| доменная архитпектура 2 | PF00067 - PF00067 - PF00067 - PF00067 - PF00067 | вберите другую доменую архитектуру с выбранным доменом | |
| Белок с архитектурой 2 | Q4STF7 | укажите идентификатор последовательности с этой архитектурой |
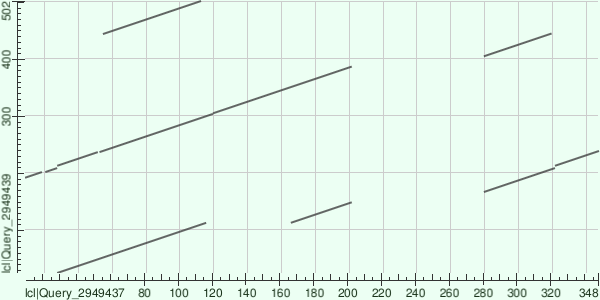
Карта локального сходства последовательностей. Как и было обозначено в названии доменной архитектуры, она состоит из шести фрагментов PF00067, более или менее полными частями, местами которые являются дупликациями.